Страница не найдена
Как пересечь границу России по земле и какая обстановка на погранпунктах
Что такое гипотиреоз и как я с ним живу уже 3 года
Как пользоваться нейросетью для генерации рисунков Midjourney: правильно составляем запрос
Посадить и любоваться: 12 неприхотливых садовых цветов
Всем интересно
См. все
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
См. все
все
Работа, ипотека и семья: вопросы, о которых стоит подумать из-за частичной мобилизации
Бронь и сохранение рабочих мест: трудовые гарантии для мобилизованных
«Начала соблюдать правило тарелки»: как я похудела на 15 кг за 9 месяцев
Что будет, если Россия перестанет поставлять газ в Европу
Вышла iOS 16: что нового и ради чего стоит обновиться
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Правда ли, что чайный гриб очень полезен?
17 стран, которые выдают туристические визы россиянам в 2022 году
Какие документы нужно иметь при себе, если вдруг остановят на улице для мобилизации?
В России началась частичная мобилизация
Студентам дали отсрочку от мобилизации: кто имеет на нее право
Пишут, что повестку о частичной мобилизации могут прислать через госуслуги: как на самом деле
Лучшее за полгода
См. все
Как я работала продавцом в супермаркетах
Бронь и сохранение рабочих мест: трудовые гарантии для мобилизованных
Работа, ипотека и семья: вопросы, о которых стоит подумать из-за частичной мобилизации
Как я открыла пункт выдачи заказов «Вайлдберриз»
Что такое гипотиреоз и как я с ним живу уже 3 года
Купить, снять, перевести: что можно делать с долларами и евро, а что нельзя
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Как женщина пришла в банк за вкладом в 37 000 $, а ей ответили, что денег нет
Пишут, что повестку о частичной мобилизации могут прислать через госуслуги: как на самом деле
Можно ли уехать из России после объявления о частичной мобилизации
Как пересечь границу России по земле и какая обстановка на погранпунктах
Как я перестала удалять волосы на теле и сколько на этом экономлю
В «Телеграме» распространяют «списки частичной мобилизации»: почему не стоит им верить
Как пользоваться нейросетью для генерации рисунков Midjourney: правильно составляем запрос
В ожидании светлого будущего: что такое синдром отложенной жизни
Как купить автомобиль с пробегом в Беларуси
Студенты, ИТ-специалисты, родители четырех детей: кто может получить отсрочку от мобилизации
Как правильно посадить и спилить дерево на своем участке
Где теперь покупать товары для дома: 60 вещей в духе IKEA и Zara Home за те же деньги
Частичная мобилизация в России: как будет проходить, кто подпадает, кого не призовут
Вышла iOS 16: что нового и ради чего стоит обновиться
Как натренировать удачу: 5 советов
Как и где сейчас купить наличную валюту?
Кто имеет право на новую выплату на детей от 8 до 17 лет
Как алкоголь влияет на психику и мозг: 6 выводов ученых
Как теперь заказывать товары в Россию из-за рубежа: 4 доступных способа
Как я получил две банковские карты в Казахстане
Могут ли мобилизовать в армию без военного билета?
Где путешественникам бронировать отели и квартиры: 10 российских сервисов
В России началась частичная мобилизация
Курсы помогут
См. все
Озеленить дом
Победить выгорание
Выбрать квартиру
Улучшить жизнь с помощью «Экселя»
Заработать на акциях
Начать инвестировать
Разобраться в благотворительности
Путешествовать безопасно
Зарабатывать на кредитке
Не прогадать с ипотекой
Защититься от мошенников
Не разориться на здоровье
Сортировать мусор
Рулить тачкой
Завести собаку
Быть самозанятым
Жить в России
Страница не найдена
Почему я больше не поеду в шопинг-тур в Финляндию
Могут ли мобилизовать в армию без военного билета?
Как женщина пришла в банк за вкладом в 37 000 $, а ей ответили, что денег нет
Работа, ипотека и семья: вопросы, о которых стоит подумать из-за частичной мобилизации
Всем интересно
См. все
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
См. все
Посадить и любоваться: 12 неприхотливых садовых цветов
Что будет, если Россия перестанет поставлять газ в Европу
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Как живет разработчик в Ереване с зарплатой 8333 $
Как пересечь границу России по земле и какая обстановка на погранпунктах
Студентам дали отсрочку от мобилизации: кто имеет на нее право
Вышла iOS 16: что нового и ради чего стоит обновиться
Какие документы нужно иметь при себе, если вдруг остановят на улице для мобилизации?
Стоит ли заменить кофе цикорием?
Частичная мобилизация в России: как будет проходить, кто подпадает, кого не призовут
В «Телеграме» распространяют «списки частичной мобилизации»: почему не стоит им верить
Правда ли, что чайный гриб очень полезен?
Лучшее за полгода
См. все
Как купить автомобиль с пробегом в Беларуси
Как женщина пришла в банк за вкладом в 37 000 $, а ей ответили, что денег нет
Вышла iOS 16: что нового и ради чего стоит обновиться
Студенты, ИТ-специалисты, родители четырех детей: кто может получить отсрочку от мобилизации
Как я открыла пункт выдачи заказов «Вайлдберриз»
Как пользоваться нейросетью для генерации рисунков Midjourney: правильно составляем запрос
Пишут, что повестку о частичной мобилизации могут прислать через госуслуги: как на самом деле
Что такое гипотиреоз и как я с ним живу уже 3 года
Как я получил две банковские карты в Казахстане
Как я перестала удалять волосы на теле и сколько на этом экономлю
Можно ли уехать из России после объявления о частичной мобилизации
В «Телеграме» распространяют «списки частичной мобилизации»: почему не стоит им верить
Частичная мобилизация в России: как будет проходить, кто подпадает, кого не призовут
Как пересечь границу России по земле и какая обстановка на погранпунктах
Как теперь заказывать товары в Россию из-за рубежа: 4 доступных способа
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Как я работала продавцом в супермаркетах
Где теперь покупать товары для дома: 60 вещей в духе IKEA и Zara Home за те же деньги
Как правильно посадить и спилить дерево на своем участке
В ожидании светлого будущего: что такое синдром отложенной жизни
Купить, снять, перевести: что можно делать с долларами и евро, а что нельзя
Работа, ипотека и семья: вопросы, о которых стоит подумать из-за частичной мобилизации
Как и где сейчас купить наличную валюту?
Могут ли мобилизовать в армию без военного билета?
Бронь и сохранение рабочих мест: трудовые гарантии для мобилизованных
Кто имеет право на новую выплату на детей от 8 до 17 лет
Как алкоголь влияет на психику и мозг: 6 выводов ученых
Как натренировать удачу: 5 советов
В России началась частичная мобилизация
Где путешественникам бронировать отели и квартиры: 10 российских сервисов
Курсы помогут
См. все
Озеленить дом
Победить выгорание
Выбрать квартиру
Улучшить жизнь с помощью «Экселя»
Заработать на акциях
Начать инвестировать
Разобраться в благотворительности
Путешествовать безопасно
Зарабатывать на кредитке
Не прогадать с ипотекой
Защититься от мошенников
Не разориться на здоровье
Сортировать мусор
Рулить тачкой
Завести собаку
Быть самозанятым
Жить в России
Страница не найдена
Частичная мобилизация в России: как будет проходить, кто подпадает, кого не призовут
«Начала соблюдать правило тарелки»: как я похудела на 15 кг за 9 месяцев
В «Телеграме» распространяют «списки частичной мобилизации»: почему не стоит им верить
Пишут, что повестку о частичной мобилизации могут прислать через госуслуги: как на самом деле
Всем интересно
См. все
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
См. все
все
Что будет, если Россия перестанет поставлять газ в Европу
Как алкоголь влияет на психику и мозг: 6 выводов ученых
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Почему я больше не поеду в шопинг-тур в Финляндию
Студенты, ИТ-специалисты, родители четырех детей: кто может получить отсрочку от мобилизации
Могут ли мобилизовать в армию без военного билета?
Бронь и сохранение рабочих мест: трудовые гарантии для мобилизованных
Посадить и любоваться: 12 неприхотливых садовых цветов
Как пользоваться нейросетью для генерации рисунков Midjourney: правильно составляем запрос
Стоит ли заменить кофе цикорием?
Как женщина пришла в банк за вкладом в 37 000 $, а ей ответили, что денег нет
Как пересечь границу России по земле и какая обстановка на погранпунктах
Лучшее за полгода
См. все
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Как я получил две банковские карты в Казахстане
Как я перестала удалять волосы на теле и сколько на этом экономлю
В России началась частичная мобилизация
Где теперь покупать товары для дома: 60 вещей в духе IKEA и Zara Home за те же деньги
Студенты, ИТ-специалисты, родители четырех детей: кто может получить отсрочку от мобилизации
Работа, ипотека и семья: вопросы, о которых стоит подумать из-за частичной мобилизации
Где путешественникам бронировать отели и квартиры: 10 российских сервисов
Купить, снять, перевести: что можно делать с долларами и евро, а что нельзя
Как теперь заказывать товары в Россию из-за рубежа: 4 доступных способа
Как женщина пришла в банк за вкладом в 37 000 $, а ей ответили, что денег нет
Кто имеет право на новую выплату на детей от 8 до 17 лет
В ожидании светлого будущего: что такое синдром отложенной жизни
Как пересечь границу России по земле и какая обстановка на погранпунктах
Как натренировать удачу: 5 советов
Пишут, что повестку о частичной мобилизации могут прислать через госуслуги: как на самом деле
Как правильно посадить и спилить дерево на своем участке
В «Телеграме» распространяют «списки частичной мобилизации»: почему не стоит им верить
Как я работала продавцом в супермаркетах
Могут ли мобилизовать в армию без военного билета?
Как пользоваться нейросетью для генерации рисунков Midjourney: правильно составляем запрос
Вышла iOS 16: что нового и ради чего стоит обновиться
Как купить автомобиль с пробегом в Беларуси
Можно ли уехать из России после объявления о частичной мобилизации
Что такое гипотиреоз и как я с ним живу уже 3 года
Как алкоголь влияет на психику и мозг: 6 выводов ученых
Частичная мобилизация в России: как будет проходить, кто подпадает, кого не призовут
Как я открыла пункт выдачи заказов «Вайлдберриз»
Как и где сейчас купить наличную валюту?
Бронь и сохранение рабочих мест: трудовые гарантии для мобилизованных
Курсы помогут
См. все
Озеленить дом
Победить выгорание
Выбрать квартиру
Улучшить жизнь с помощью «Экселя»
Заработать на акциях
Начать инвестировать
Разобраться в благотворительности
Путешествовать безопасно
Зарабатывать на кредитке
Не прогадать с ипотекой
Защититься от мошенников
Не разориться на здоровье
Сортировать мусор
Рулить тачкой
Завести собаку
Быть самозанятым
Жить в России
Страница не найдена
Как пересечь границу России по земле и какая обстановка на погранпунктах
Что такое гипотиреоз и как я с ним живу уже 3 года
662465+00:00″ itemprop=»datePublished»>11.08.22
Как пользоваться нейросетью для генерации рисунков Midjourney: правильно составляем запрос
Посадить и любоваться: 12 неприхотливых садовых цветов
Всем интересно
См. все
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
Дневники трат
Инвестиции для начинающих
Финансовая подушка
Льготы от государства
Как снять квартиру
Как погасить кредит
См. все
Работа, ипотека и семья: вопросы, о которых стоит подумать из-за частичной мобилизации
Бронь и сохранение рабочих мест: трудовые гарантии для мобилизованных
«Начала соблюдать правило тарелки»: как я похудела на 15 кг за 9 месяцев
Что будет, если Россия перестанет поставлять газ в Европу
Вышла iOS 16: что нового и ради чего стоит обновиться
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Правда ли, что чайный гриб очень полезен?
17 стран, которые выдают туристические визы россиянам в 2022 году
Какие документы нужно иметь при себе, если вдруг остановят на улице для мобилизации?
В России началась частичная мобилизация
Студентам дали отсрочку от мобилизации: кто имеет на нее право
Пишут, что повестку о частичной мобилизации могут прислать через госуслуги: как на самом деле
Лучшее за полгода
См. все
Как я работала продавцом в супермаркетах
Бронь и сохранение рабочих мест: трудовые гарантии для мобилизованных
Работа, ипотека и семья: вопросы, о которых стоит подумать из-за частичной мобилизации
Как я открыла пункт выдачи заказов «Вайлдберриз»
Что такое гипотиреоз и как я с ним живу уже 3 года
Купить, снять, перевести: что можно делать с долларами и евро, а что нельзя
«Думала о том, что уже не выдерживаю»: как я похудела на 20 кг за 5 месяцев
Как женщина пришла в банк за вкладом в 37 000 $, а ей ответили, что денег нет
Пишут, что повестку о частичной мобилизации могут прислать через госуслуги: как на самом деле
Можно ли уехать из России после объявления о частичной мобилизации
Как пересечь границу России по земле и какая обстановка на погранпунктах
Как я перестала удалять волосы на теле и сколько на этом экономлю
В «Телеграме» распространяют «списки частичной мобилизации»: почему не стоит им верить
Как пользоваться нейросетью для генерации рисунков Midjourney: правильно составляем запрос
В ожидании светлого будущего: что такое синдром отложенной жизни
Как купить автомобиль с пробегом в Беларуси
Студенты, ИТ-специалисты, родители четырех детей: кто может получить отсрочку от мобилизации
Как правильно посадить и спилить дерево на своем участке
Где теперь покупать товары для дома: 60 вещей в духе IKEA и Zara Home за те же деньги
Частичная мобилизация в России: как будет проходить, кто подпадает, кого не призовут
Вышла iOS 16: что нового и ради чего стоит обновиться
Как натренировать удачу: 5 советов
Как и где сейчас купить наличную валюту?
Кто имеет право на новую выплату на детей от 8 до 17 лет
Как алкоголь влияет на психику и мозг: 6 выводов ученых
Как теперь заказывать товары в Россию из-за рубежа: 4 доступных способа
Как я получил две банковские карты в Казахстане
Могут ли мобилизовать в армию без военного билета?
Где путешественникам бронировать отели и квартиры: 10 российских сервисов
В России началась частичная мобилизация
Курсы помогут
См. все
Озеленить дом
Победить выгорание
Выбрать квартиру
Улучшить жизнь с помощью «Экселя»
Заработать на акциях
Начать инвестировать
Разобраться в благотворительности
Путешествовать безопасно
Зарабатывать на кредитке
Не прогадать с ипотекой
Защититься от мошенников
Не разориться на здоровье
Сортировать мусор
Рулить тачкой
Завести собаку
Быть самозанятым
Жить в России
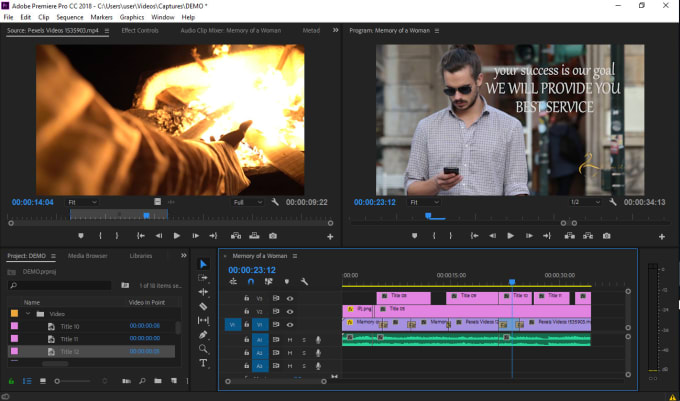
Топ-9 программ для монтажа видео на русском языке
Если вы начинаете свой путь в мире монтажа, посмотрите список ниже, чтобы подобрать подходящий редактор.
Уметь монтировать видео сегодня почти так же необходимо, как уметь читать. Ведь теперь нам уже недостаточно просто съемок из отпуска, мы привыкли видеть хорошо смонтированные клипы и видеозарисовки – чтобы там были титры и переходы между планами, интересные эффекты и смешные стикеры, закадровый голос и, конечно, фоновая музыка. Ну, а если вы ведете видеоблог или создаете обучающие ролики, то без отличных навыков монтажа вам просто не обойтись. Здесь мы приводим обзор лучших, по нашему мнению, программ для редактирования видео на компьютере. Читайте и выбирайте самый удобный инструмент для воплощения ваших идей.
Ну, а если вы ведете видеоблог или создаете обучающие ролики, то без отличных навыков монтажа вам просто не обойтись. Здесь мы приводим обзор лучших, по нашему мнению, программ для редактирования видео на компьютере. Читайте и выбирайте самый удобный инструмент для воплощения ваших идей.
Многофункциональная программа для монтажа видео с простым и понятным интерфейсом и богатым набором инструментов для обработки видео. Одна из лучших с точки зрения удобства использования. Освоить программу ничего не стоит даже не имея никакого опыта видеомонтажа, а результат получите классный.
Помимо базовых задач монтирования видеоряда: нарезки, склеивания, поворота и кадрирования, Movavi Video Editor Plus позволит вам стабилизировать картинку, приблизить или удалить объекты, выделить или скрыть отдельные зоны кадра. В вашем распоряжении также цветокоррекция, различные фильтры и спецэффекты, анимация, компоновка нескольких видео в одном кадре, замедление, ускорение и обратное проигрывание. А функция хромакей, то есть замена фона, дает большой простор для творчества: вы сможете перенести героя в любую другую обстановку. Стоит отметить большой выбор переходов, титров и стикеров разной тематики. Есть функции захвата видео с экрана, записи видео с вебкамеры и автоматического монтажа. Также с помощью программы можно улучшить качество уже снятого видео.
Стоит отметить большой выбор переходов, титров и стикеров разной тематики. Есть функции захвата видео с экрана, записи видео с вебкамеры и автоматического монтажа. Также с помощью программы можно улучшить качество уже снятого видео.
Как улучшить качество видео
Звуковую дорожку тоже можно изменять: вырезать ненужное, менять громкость, скорость проигрывания, подавлять шумы, проигрывать обратно, использовать появление/исчезание. В Movavi Video Editor Plus вы сможете записать свой голос для комментария, наложить дубляж и фоновую музыку, добавить аудиоэффекты. Приятным бонусом является встроенная коллекция музыки, видеоклипов, фонов и заставок. В программе можно выбрать любые из множества тематических наборов эффектов Movavi.
Взаимодействие с редактором технически очень просто – можно открыть программу впервые и сразу начать в ней работать, поэтому она отлично подходит для начинающих. Иконки и надписи информативны, есть всплывающие подсказки, многое можно сделать простым перетаскиванием. Важно, что есть возможность добавлять в проект любое количество видео- и аудиодорожек и треков титров. Можно выключать звук трека или клипа и откреплять звук от видео. Удобно и то, что масштаб временной шкалы меняется, а плеер можно открепить от окна программы. Любые ложные действия вы легко отмените возвратом назад на сколько угодно шагов – это особенно важно на первых порах. При работе над видео вы сможете сохранить его в виде проекта, чтобы вернуться к редактированию в любой момент. При экспорте готового фильма можно записать его в любом популярном формате, с настраиваемым качеством и разрешением. Если вам потребуется совет по работе с программой, зайдите на наш YouTube-канал: там вы найдете видео с обучением тому, как монтировать короткие видеозарисовки, делать интро и концовки к видео, как выкладывать результат на видеохостинге.
Важно, что есть возможность добавлять в проект любое количество видео- и аудиодорожек и треков титров. Можно выключать звук трека или клипа и откреплять звук от видео. Удобно и то, что масштаб временной шкалы меняется, а плеер можно открепить от окна программы. Любые ложные действия вы легко отмените возвратом назад на сколько угодно шагов – это особенно важно на первых порах. При работе над видео вы сможете сохранить его в виде проекта, чтобы вернуться к редактированию в любой момент. При экспорте готового фильма можно записать его в любом популярном формате, с настраиваемым качеством и разрешением. Если вам потребуется совет по работе с программой, зайдите на наш YouTube-канал: там вы найдете видео с обучением тому, как монтировать короткие видеозарисовки, делать интро и концовки к видео, как выкладывать результат на видеохостинге.
Программа имеет бесплатную пробную версию. Скачайте видеоредактор Movavi для Windows 7, 8, 10 или Mac и получите в свое распоряжение удобную программу на русском языке для профессионального нелинейного монтажа. Даже если вы новичок в этой сфере, Movavi Video Editor Plus позволит вам легко и быстро сделать хороший фильм!
Даже если вы новичок в этой сфере, Movavi Video Editor Plus позволит вам легко и быстро сделать хороший фильм!
Скачать бесплатно
Скачать бесплатно
Скачать бесплатно
Скачать бесплатно
Adobe® Premiere® Pro
Профессиональный инструмент для создания фильмов, клипов, рекламных роликов и видеоуроков. Работа в Adobe® Premiere® Pro требует специальной подготовки, то есть, в отличие от предыдущего видеоредактора, открыть программу и сразу начать монтировать не получится. В ней довольно сложный интерфейс, который надо конфигурировать и настраивать, уже зная, что и как. Кроме того, алгоритмы действий для многих инструментов не очевидны и многоступенчаты. Поэтому, если видеомонтаж не является вашей профессией, то такая программа будет скорее избыточной.
Вместе с тем, в руках мастера, это приложение дает очень качественный результат. Огромное количество функций, настроек и спецэффектов для видео и аудио позволяет создавать самые сложные проекты и сохранять их с точно заданными параметрами. Отметим, что некоторые инструменты требуют скачивания дополнительных плагинов. Видеоредактор интегрируется со всеми продуктами линейки Adobе и шаблонами Adobe® Effects®. В целом продукт довольно дорог, сложен и занимает много места на диске ПК или ноутбука. При умелом использовании позволяет создавать фильмы высокого уровня.
Отметим, что некоторые инструменты требуют скачивания дополнительных плагинов. Видеоредактор интегрируется со всеми продуктами линейки Adobе и шаблонами Adobe® Effects®. В целом продукт довольно дорог, сложен и занимает много места на диске ПК или ноутбука. При умелом использовании позволяет создавать фильмы высокого уровня.
Vegas Pro
Мощная программа профессионального уровня для редактирования видео и звука и создания качественной видеопродукции: фильмов, телепередач, музыкальных клипов, рекламных роликов.
Этот видеоредактор немного похож на предыдущий по уровню сложности и своему интерфейсу, ведь уже на этапе создания проекта нужно задавать множество специальных характеристик, которые незнакомы большинству людей имеющих дело с видео в повседневной жизни. Однако, если вы регулярно монтируете видео, скажем, для YouTube-канала и решите потратить время на освоение программы, то Vegas Pro может стать для вас подходящим рабочим вариантом.
Из явных плюсов программы: расширенная цветовая градация, возможность рисовать на видео, мастеринг аудио SOUND FORGE Pro и возможность использовать плагин для крутых спецэффектов. Хирургически точная обработка звука позволяет убирать искажения с аудиодорожки, восстанавливать звук, эффективно устранять фоновый шум. Что же касается спецэффектов в Vegas Pro, то они сравнимы с компьютерной графикой, используемой в большом кино, и позволяют превратить обычное видео в экшен или боевик. Из минусов – требовательность к ресурсам компьютера, так что слабый ПК не подойдет для этого видеоредактора. Часть пользователей отмечают также нестабильность программы.
Хирургически точная обработка звука позволяет убирать искажения с аудиодорожки, восстанавливать звук, эффективно устранять фоновый шум. Что же касается спецэффектов в Vegas Pro, то они сравнимы с компьютерной графикой, используемой в большом кино, и позволяют превратить обычное видео в экшен или боевик. Из минусов – требовательность к ресурсам компьютера, так что слабый ПК не подойдет для этого видеоредактора. Часть пользователей отмечают также нестабильность программы.
Pinnacle Studio
Этот видеоредактор подойдет для широкого круга пользователей, так как он не слишком сложен. Правда, занимает много места на компьютере, и это стоит учесть. Интерфейс программы классического типа: слева инструменты и функции, справа плеер для предпросмотра, внизу рабочая область с временной шкалой и треками. В базовой версии можно добавить не более шести треков.
Функционал программы включает все необходимое для того, чтобы создавать видео хорошего качества для повседневных нужд: семейных фильмов и видеороликов для Ютуба или соцсетей. Вы можете редактировать видео и звук, разделять экран, добавлять переходы и титры, аудио- и видеоэффекты. В программу включен захват экрана и запись голоса. Интересная возможность Pinnacle Studio – работа с масками с использованием трекинга, так можно применить какой-то эффект в видео лишь на заданном объекте, скажем откорректировать освещенность только на лице одного из героев. Недавно появился и инструмент индивидуальной анимации титров.
Вы можете редактировать видео и звук, разделять экран, добавлять переходы и титры, аудио- и видеоэффекты. В программу включен захват экрана и запись голоса. Интересная возможность Pinnacle Studio – работа с масками с использованием трекинга, так можно применить какой-то эффект в видео лишь на заданном объекте, скажем откорректировать освещенность только на лице одного из героев. Недавно появился и инструмент индивидуальной анимации титров.
Создавать более интересные видео и делать это гораздо проще и быстрее можно с Movavi Video Suite. Это – комплексная программа для работы с мультимедиа с современным функционалом и дружелюбным интуитивным интерфейсом. Удобный видеоредактор Mоvavi – составная часть этого уникального комплекса. Но кроме редактирования видео у вас в едином пространстве и всегда под рукой: инструмент для захвата видео с экрана и снятия скриншотов, высокоскоростной конвертер медиафайлов во все форматы и для любого устройства, утилиты для записи дисков, оцифровки VHS и TV-сигнала.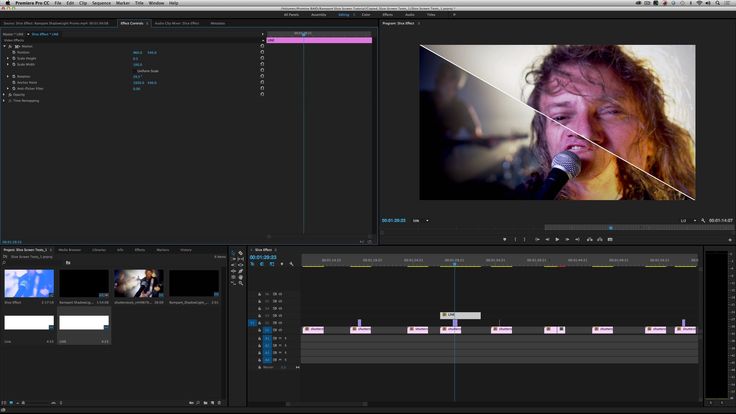
Этот универсальный инструмент специально продуман и создан так, чтобы вам не нужно было устанавливать никакие дополнительные программы, потому что все необходимое для создания отличного видео есть у вас в быстром доступе и перед глазами. Другие составные части мультимедийной среды Movavi Video Suite это: медиапроигрыватель, встроенные коллекции мультимедиа, эффектов и стикеров, хранилище файлов и недавних проектов, мобильный помощник, а также сообщество единомышленников Movavi Video Suite. А еще в программу включен доступ к дополнительным ресурсам: стокам и онлайн-курсам. При этом запросы программы по ресурсам весьма скромные.
Скачать бесплатно
Скачать бесплатно
Скачать бесплатно
Скачать бесплатно
DaVinci Resolve
Эту программу можно отнести к «тяжелой артиллерии» видеомонтажа. Ее используют в кинопроизводстве и телеиндустрии. В этом бесплатном видеоредакторе заложено множество возможностей для сложной профессиональной обработки видеоряда, которые будут эффективны в руках подготовленного специалиста. Однако для задач домашнего видео или видеоблоггинга 90% из них останутся невостребованными. Интерфейс средней сложности: интуитивным его не назовешь, но разобраться в нем вполне реально.
Однако для задач домашнего видео или видеоблоггинга 90% из них останутся невостребованными. Интерфейс средней сложности: интуитивным его не назовешь, но разобраться в нем вполне реально.
Самой сильной стороной DaVinci Resolve считается цветокоррекция, здесь доступны самые тонкие настройки картинки и даже можно сделать цветным черно-белый фильм. Впечатляет количество визуальных эффектов. В программе также есть функционал для продвинутой обработки звука, так используя ключевые точки, можно задать настройки отдельным фрагментам трека. Интересная деталь – наличие набора аудиопереходов, чтобы вставлять их между составными частями звуковой дорожки. Создатели этого программного решения предусмотрели возможность совместной работы над проектом, что может оказаться полезным.
Популярный бесплатный видеоредактор со множеством как базовых, так и продвинутых функций, среди которых нелинейное редактирование. Из очевидных преимуществ программы: работа со множеством медиаформатов и низкий запрос по ресурсам. В программе довольно просто разобраться, что дает возможность быстро перейти к практическому монтажу видео. Из существенных минусов – отсутствует запись голоса для создания закадровых пояснений.
В программе довольно просто разобраться, что дает возможность быстро перейти к практическому монтажу видео. Из существенных минусов – отсутствует запись голоса для создания закадровых пояснений.
Помимо простых манипуляций изменения видео (нарезка, удаление фрагментов, склеивание видео, поворот, кадрирование и т.п.) VSDC предлагает инструменты для работы над изображением внутри выбранного фрагмента видео: коррекцию цвета, наложение цветовых фильтров, применение других видеоэффектов, цензурирование части изображения. Полезной является также функция отслеживания заданного объекта, которая позволяет привязать к объекту графический элемент или надпись. Кроме того, вы можете обогатить свое видео, используя визуализатор музыки: программа умеет создавать на экране движущиеся графические объекты в такт звуковым волнам.
В видеоредактор включен конвертер файлов, функция записи DVD, захват экрана и видео с подключенных устройств, а также менеджер загрузки файлов с различных сайтов, который позволяет скачивать видео со многих веб-ресурсов.
Shotcut
Функциональный видеоредактор с открытым исходным кодом. Работает на всех основных платформах и включает множество полезных при монтаже инструментов и функций, как основных, так и продвинутых. Всеяден в смысле форматов и кодеков. Не всегда стабилен, но при сохранении каждого действия, это не слишком критично. Интерфейс программы нельзя назвать лаконичным и понятным с первого взгляда, но можно изменить его как вам удобно: просто скройте лишние вкладки или добавьте те, которые часто используете. Можно также выбрать цветовую гамму интерфейса: темную, светлую или системную.
Программа предлагает все классические возможности создания видео: компоновку и редактирование видео- и аудиодорожек, добавление переходов, вставку титров. Вы сможете обработать видео с помощью интересных фильтров и эффектов, которые могут применяться плавно, по заданным ключевым точкам. Учтите, что при работе с текстом, вы не найдете в редакторе демонстрационных клипов титров, результат будет виден только при наложении титра на ваше видео. Добавление переходов также отличается от обычного перетаскивания на видеотрек и имеет более сложный алгоритм. Стоит отметить, что в программе нет произвольного масштабирования шкалы времени на рабочем столе: есть только две заданные возможности. А вот при сохранении проекта вас ждет приятный сюрприз. Сохранение происходит в отдельной вкладке, и можно приступить к следующему проекту, не дожидаясь завершения экспорта текущего.
Добавление переходов также отличается от обычного перетаскивания на видеотрек и имеет более сложный алгоритм. Стоит отметить, что в программе нет произвольного масштабирования шкалы времени на рабочем столе: есть только две заданные возможности. А вот при сохранении проекта вас ждет приятный сюрприз. Сохранение происходит в отдельной вкладке, и можно приступить к следующему проекту, не дожидаясь завершения экспорта текущего.
CyberLink PowerDirector
Качественная программа для видеотворчества. Интерфейс средней сложности: в целом, классическое расположение окон и панелей, плюс возможность индивидуально настроить показ нужных вкладок. Стоит, однако, учесть, что работать придется в английском интерфейсе, потому что русский язык в программе отсутствует.
Видеоредактор предлагает широкий функционал, который при этом вполне доступен для применения и не специалисту в видеообработке, т. к. здесь вы не найдете пугающе профессиональных тонкостей и сверхсложных регулировок. В программе есть все основные инструменты, чтобы смонтировать видео.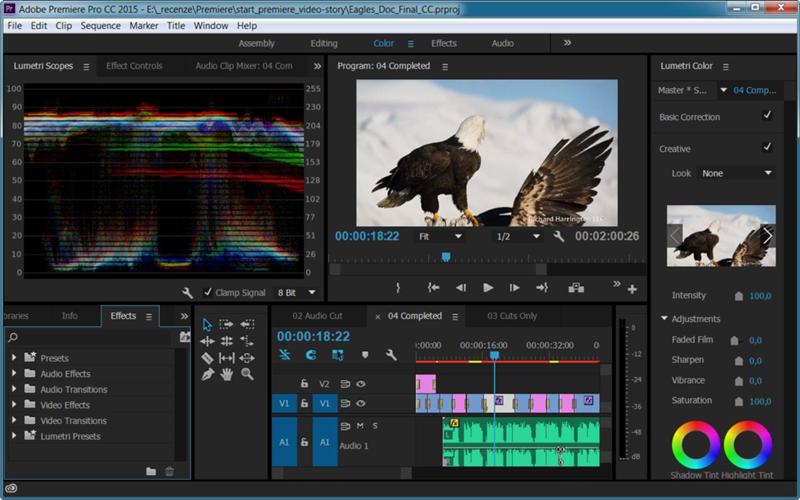 Вы сможете составить видеоряд, убрав лишние и добавив нужные кадры, настроить изображение, применить эффекты, добавить переходы и снабдить видео титрами. Есть возможность замедления или ускорения проигрывания, обратное проигрывание, зацикливание видео, стоп-моушен, стабилизация изображения, исправление искажения «рыбий глаз». Помимо этого в программе есть многое для более свободного творчества, например, большая коллекция анимированных персонализируемых титров и доступ к богатейшей медиатеке, которую можно использовать при создании своего видео. Из других продвинутых инструментов – улучшенный дизайнер масок, благодаря которому можно сверхточно вырезать элементы изображения и накладывать их на другие кадры, создавая реалистичный или сюрреалистичный эффект. Динамический контроль ключевых кадров позволяет задать траекторию титров или наложенных графических объектов при анимации. Программа включает большое количество спецэффектов.
Вы сможете составить видеоряд, убрав лишние и добавив нужные кадры, настроить изображение, применить эффекты, добавить переходы и снабдить видео титрами. Есть возможность замедления или ускорения проигрывания, обратное проигрывание, зацикливание видео, стоп-моушен, стабилизация изображения, исправление искажения «рыбий глаз». Помимо этого в программе есть многое для более свободного творчества, например, большая коллекция анимированных персонализируемых титров и доступ к богатейшей медиатеке, которую можно использовать при создании своего видео. Из других продвинутых инструментов – улучшенный дизайнер масок, благодаря которому можно сверхточно вырезать элементы изображения и накладывать их на другие кадры, создавая реалистичный или сюрреалистичный эффект. Динамический контроль ключевых кадров позволяет задать траекторию титров или наложенных графических объектов при анимации. Программа включает большое количество спецэффектов.
В нашем топе программ мы постарались отметить основные характеристики наиболее популярных приложений для видеомонтажа, которые можно использовать для создания домашнего видео, роликов для соцсетей, клипов для видеоблога или видеоуроков. Выбирайте подходящий вам видеоредактор, исходя из своих целей, опыта и количества времени, которое вы готовы потратить на освоение программы.
Выбирайте подходящий вам видеоредактор, исходя из своих целей, опыта и количества времени, которое вы готовы потратить на освоение программы.
Желаем вам удачи в творчестве!
Создавайте видео. Творите. Вдохновляйте.
* Бесплатная версия Movavi Видеоредактора Плюс имеет следующие ограничения: 7-дневный пробный период, водяной знак на сохраненных видео и, если вы сохраняете проект как аудиофайл, возможность сохранить только ½ длины аудио.
Остались вопросы?
Подпишитесь на рассылку о скидках и акциях
Подписываясь на рассылку, вы соглашаетесь на получение от нас рекламной информации по электронной почте и обработку персональных данных в соответствии с Политикой конфиденциальности Movavi. Вы также подтверждаете, что не имеете препятствий к выражению такого согласия. Читать Политику конфиденциальности.
Монтируем видео на облачном сервере в AWS / Хабр
Мой рабочий компьютер — Macbook Air 2020 (Intel), и его вполне хватает для написания кода и прочих задач. Однако, когда потребовалось смонтировать небольшой видеоролик, выяснилось что мощи моего ноута катастрофически не хватает, и я стал искать варианты.
Однако, когда потребовалось смонтировать небольшой видеоролик, выяснилось что мощи моего ноута катастрофически не хватает, и я стал искать варианты.
Что я пытался сделать
Я не занимаюсь профессиональным монтажом видео, и впервые уткнулся в эту тему пытаясь сделать вот такой небольшой демонстрационный ролик для Teamplify (с озвучкой помог Мэтт, наш учитель английского):
На первый взгляд, тут ничего сложного — это же просто трехминутный скринкаст с озвучкой и парой незатейливых эффектов. Однако, видимо по неопытности, я встретил на этом пути какое-то неожиданное количество подводных граблей. После нескольких неудачных попыток, я пришел к трем важным выводам:
Обязательно нужен сценарий, и его важно написать заранее, продумав дословно все что будет говориться и делаться в кадре;
Записать это единым куском практически невозможно — то замешкаешься где-то, то ошибешься, то интернет подтупит;
Нельзя просто так взять и записать качественный звук в домашних условиях.

В итоге, конечный результат я собирал в After Effects из множества отдельных коротких видео, скриншотов, и аудио-фрагментов, которые подгонялись по таймингу, и на них накладывались эффекты с переходами:
Фоновую музычку изначально добавлять не планировал, но оказалось что она прекрасно маскирует не совсем качественную запись с микрофона, так что добавил.
Монтаж видео требует ресурсов
Мои попытки работать с видео на Macbook Air были похожи на мучение. Оказалось, что After Effects любит побольше памяти, и моих 16ГБ явно не хватало. Ноутбучный проц и слабая видюха также не улучшали картину. Превью в After Effects работало с сильными лагами. Финальный рендер трехминутного ролика занимал 3 часа (!!).
Если бы у меня был настольный игровой комп с видюхой и кучей памяти, наверное, приключения на этом бы и закончились, но у меня такого не было, а идея вложить в него пару тысяч долларов (да и вообще иметь такую бандуру дома) мне не нравилась.
Что нам может предложить AWS?
Мне он предложил примерно следующее:
Регион eu-north-1 (Стокгольм), пинг до меня ~30мс (я живу в Вильнюсе), до Москвы чуть побольше, но тоже неплохо — около 40мс;
Сервер g4dn.
 2xlarge — 8 ядер, 32ГБ, Nvidia Tesla T4, 225 GB NVMe SSD;
2xlarge — 8 ядер, 32ГБ, Nvidia Tesla T4, 225 GB NVMe SSD;Образ винды с дровами от Nvidia;
Цену в $1.166 в час. И это кстати еще одна причина выбрать Стокгольм, ибо во Франкфурте на ~12% дороже.
Разумеется, можно взять и побольше, и поменьше. На момент написания статьи цены в Стокгольме такие (инстансы с Nvidia Tesla T4, на винде):
Подготовка к работе
Для коннекта к серверу я использовал Microsoft Remote Desktop. Сначала я думал, что мне потребуются какие-то специальные клиенты, рассчитанные на стриминг видео, но оказалось что и обычный Remote Desktop неплохо справляется, никаких особенных лагов я не замечал.
Остается решить вопрос с диском и доставкой исходников на него. Тут амазона предлагает нам два варианта — EBS и Instance Store. Отличаются они следующим:
EBS — подключаемый сетевой диск, по надежности примерно как RAID 1. Такой диск в вашем сервере обязательно будет как основной, по умолчанию он небольшого размера (достаточного чтобы влезла операционка), но вы можете при желании его расширить.
 Данные на EBS сохраняются и после выключения сервера. Платите вы за такой диск постоянно, даже если сервер выключен, в размере $0.0836 за ГБ в месяц (в версии gp3, burstable IOPS). У EBS есть
Данные на EBS сохраняются и после выключения сервера. Платите вы за такой диск постоянно, даже если сервер выключен, в размере $0.0836 за ГБ в месяц (в версии gp3, burstable IOPS). У EBS есть
Instance store — так называемый “эфемерный” диск, который может идти в комплекте к некоторым типам инстансов. В частности, к g4dn.2xlarge прилагается 225 GB NVMe SSD. Он быстрый, производительность ничем не ограничена, однако данные на нем полностью пропадают при выключении сервера (при перезагрузке — остаются).
Поскольку монтаж видео активно использует и диск тоже, использование Instance store выглядело для меня более привлекательным. По умолчанию он не монтируется как диск в Windows, поэтому нужно сделать небольшую настройку чтобы он автоматически подключался при запуске.
По умолчанию он не монтируется как диск в Windows, поэтому нужно сделать небольшую настройку чтобы он автоматически подключался при запуске.
Когда диск готов, нужно как-то доставить на него исходники. Я использую для этого pCloud (аналог Dropbox), который установлен и на моем ноуте, и на облачном сервере. При старте сервера на нем запускается небольшой батничек, который инициализирует расшаренную папку pCloud (для исходников и результатов) и заодно создает папку для кеша After Effects:
mkdir Z:\"Teamplify Videos" mkdir Z:\"AE Cache"
После этого стартует pCloud, который синхронизирует папку “Teamplify Videos” с исходниками. Отрендеренный результат я пишу в ту же папку, и таким образом он попадает обратно на мой компьютер.
Как возможную альтернативу Dropbox / pCloud можно еще рассмотреть что-то вроде ownCloud с хранилищем на S3. Если исходники будут выкачиваться из S3 в том же AWS регионе, что и ваш сервер, то это должно быть очень быстро, что может иметь значение при большом размере исходников.
Ну и заключительный штрих в настройке — я добавил CloudWatch alarm, который автоматически выключает сервер после пары часов неактивности, чтобы не забыть его случайно включенным и не тратить деньги.
И как это все работает?
Да вроде неплохо. Превью в After Effects нормальное, без лагов. Поскольку пинг до сервера хороший, работа на нем почти не отличается от работы с настольным компьютером. Рендер того ролика, который у меня на ноуте выполнялся три часа, на g4dn.2xlarge делается за 13-17 минут. День работы на таком сервере обходится в несколько долларов.
Из побочных плюсов — я сохраняю мобильность с ноутом, и могу работать с видео откуда угодно, лишь бы были нормальные интернеты.
P.S. После опубликования этой статьи @Stas911 подкинул идею попробовать NICE DCV вместо Remote Desktop для подключения к серверу (для EC2 — бесплатно), и я попробовал. Работает 🙂 Запускайте сервер на базе NICE DCV AMI (версия для g4), не забудьте добавить роль инстансу чтобы оно перестало ругаться на лицензию, и установите EC2Launch v2 чтобы инстанс видел собственную мета-дату.
EC2Launch вам также понадобится чтобы подключить Instance store как диск в винде. Для этого натыкайте в нем мышкой галочку «Initialize» в разделе Volumes и перезагрузите сервер:
Разработка пользовательских простых редакторов с помощью программного обеспечения
Этот веб-сайт предоставляет pegRNAs и вторичные sgRNAs для основных редакторов PE2, PE3 и PE3b для ClinVar патогенные варианты человека.
Если вы используете какие-либо ресурсы, доступные на этом веб-сайте, укажите:
Автоматизированный дизайн основных редакторов CRISPR для 56 000 вариантов патогенов человекаКраткое руководство:
- 1) Нажмите на Дизайнер главного редактора вкладка выше.

- 2) Введите символ гена или идентификатор ClinVar. Инструмент будет разрабатывать pegRNAs и
PE3/3b sgRNAs для просмотра и загрузки.
- 1) Нажмите на Индивидуальные главные редакторы ClinVar вкладка выше.
- 2) Введите все необходимые параметры дизайна (список генов, шаблон PAM, размер PBS и размер RTT) и укажите адрес электронной почты для отправки результатов дизайна.
- 3) Нажмите Дизайнерские главные редакторы и дождитесь подтверждающего сообщения.
 Ссылка для скачивания будет отправлена на указанный адрес электронной почты после завершения выполнения алгоритма проектирования.
Ссылка для скачивания будет отправлена на указанный адрес электронной почты после завершения выполнения алгоритма проектирования.
- 1) Нажмите на Главный редактор Weekly ClinVar вкладка под Дополнительные ресурсы выпадающее меню.
- 2) Массовая загрузка простых редакторов, щелкнув адрес ссылки или используя «wget», а затем адрес ссылки (щелчок правой кнопкой мыши + «копировать адрес ссылки»).
Лаборатория Санджана
Пожалуйста, выберите поле для поиска:
Генный символ Идентификатор варианта ClinVar
Первичное редактирование, чтобы сделать:
Модель заболевания ТерапевтическийВсе
Показать PE3(b) sgRNAs:
по крайней мере с 1 оптимальной пегРНКпо крайней мере с 1 пригодной к использованию пегРНКпо крайней мере с 1 пегРНКпоказать все sgРНК PE3(b)
Выберите сайт PAM:
Все НГГ НГ
Показать реагенты без нецелевых показателей (Хсу-Скотта)?
Да Нет
Введите минимальное количество очков вне цели (Хсу-Скотт):
- ПЭ2
- PE3
- PE3b
Отфильтровать отображаемые пегРНК по:
Предпочтительная и нецелевая оптическая пегРНКПредпочитаемая пегРНКЛюбая пегРНК
Скачать пегРНК PE2
Скачать PE3 sgRNAs
Скачать PE3b sgRNAs
Лаборатория Санджана
Пользователи, чьи требования к дизайну не отражены на вкладке «Конструктор основных редакторов», могут разработать основные редакторы ClinVar в соответствии со своими потребностями, которые будут отправлены им по электронной почте.
Укажите список символов генов, разделенных запятыми:
Выберите шаблон PAM:
НГГ
НГ
SpRY без PAM
Дизайн NG-PAM увеличит количество целевых вариантов и количество pegRNAs/sgRNAs, разработанных для каждого варианта, но займет больше времени для разработки. Дизайн без PAM восстановит практически все варианты, но на их создание уйдет больше всего времени.
PBS Размер:
Увеличение размера PBS и RTT приведет к увеличению количества целевых вариантов и количества pegRNAs/sgRNAs, разработанных для каждого варианта, но займет больше времени для разработки.
Размер RTT:
Укажите адрес электронной почты для отправки результатов:
Лаборатория Санджана
Пользовательские сценарии R могут быть адаптированы для разработки основных редакторов для любых видов, как показано в нашем руководстве (на примере дизайна основного редактирования в рыбках данио).
 После создания pegRNAs и sgRNAs с помощью R-скриптов мы оцениваем показатели совпадения/несоответствия цели (и пересечения с распространенными вариантами dbSNP, если применимо), используя сценарии, перечисленные в разделе
Флэш-анализ
а также
анализ пересечения
каталоги соответственно.
После создания pegRNAs и sgRNAs с помощью R-скриптов мы оцениваем показатели совпадения/несоответствия цели (и пересечения с распространенными вариантами dbSNP, если применимо), используя сценарии, перечисленные в разделе
Флэш-анализ
а также
анализ пересечения
каталоги соответственно.Sanjana Lab
Благодарим вас за использование веб-инструмента Prime Editing Design.
Весь исходный код для нашей работы доступен на нашем Гит архив. Мы также приветствуем обратную связь или вопросы: напишите нам по адресу [email protected].
Связанные ресурсы:
Для тех, кто заинтересован в разработке других типов реагентов CRISPR для автономного использования или мультиплексных подходов в сочетании с основным редактированием, воспользуйтесь другими нашими онлайн-инструментами:
Sanjana Lab
Программное обеспечение PrimeDesign для быстрого и упрощенного дизайна основных направляющих РНК для редактирования
Abstract
Основное редактирование (PE) — это универсальная технология редактирования генома, но разработка необходимых направляющих РНК более сложна, чем для стандартного CRISPR- на основе нуклеаз или основных редакторов. Здесь мы описываем PrimeDesign, удобное для пользователя комплексное веб-приложение и инструмент командной строки для разработки экспериментов PE. PrimeDesign можно использовать для приложений одиночного и комбинированного редактирования, а также для скрининга полногеномного мутагенеза и мутагенеза насыщения. Используя PrimeDesign, мы создаем PrimeVar, всеобъемлющую базу данных с возможностью поиска, которая включает в себя комбинации РНК-кандидатов для первичного редактирования (pegRNA) и nicking sgRNA (ngRNA) для установки или исправления > 68 500 патогенных генетических вариантов человека из базы данных ClinVar. Наконец, мы используем PrimeDesign для разработки pegRNAs/ngRNAs для внедрения различных патогенных вариантов человека в клетки человека.
Здесь мы описываем PrimeDesign, удобное для пользователя комплексное веб-приложение и инструмент командной строки для разработки экспериментов PE. PrimeDesign можно использовать для приложений одиночного и комбинированного редактирования, а также для скрининга полногеномного мутагенеза и мутагенеза насыщения. Используя PrimeDesign, мы создаем PrimeVar, всеобъемлющую базу данных с возможностью поиска, которая включает в себя комбинации РНК-кандидатов для первичного редактирования (pegRNA) и nicking sgRNA (ngRNA) для установки или исправления > 68 500 патогенных генетических вариантов человека из базы данных ClinVar. Наконец, мы используем PrimeDesign для разработки pegRNAs/ngRNAs для внедрения различных патогенных вариантов человека в клетки человека.
Введение
Прайм-редактирование — это недавно разработанный класс технологий редактирования генома клеток млекопитающих, который обеспечивает беспрецедентную точность при установке специфических замен, вставок и делеций в геном 1 , предлагая большую гибкость, чем нуклеазы CRISPR 2,3, 4 и базовые редакторы 5,6 . Самая эффективная система первичного редактирования, описанная на сегодняшний день (называемая PE3), состоит из трех компонентов: слитого белка CRISPR-Cas9никаза и сконструированная обратная транскриптаза (RT), основная направляющая РНК для редактирования (pegRNA) и никирующая sgRNA (ngRNA) (приложение, рис. 1). ПегРНК нацелена на слияние никазы Cas9 и RT с определенным геномным локусом, но также гибридизуется с одноцепочечной ДНК с разрывом, не являющейся целевой нитью (NTS) внутри Cas9-индуцированной R-петли, и служит матрицей для обратной транскрипции в создать «лоскут», который опосредует индукцию точных генетических изменений (дополнение рис. 1a–c). нгРНК направляет слияние никазы Cas9 и RT, чтобы надрезать нить, противоположную лоскуту, и тем самым смещает репарацию в сторону желаемого изменения, закодированного в лоскуте (приложение, рис. 1d, e). Сложность системы PE3 требует много времени для ручного конструирования необходимых компонентов pegRNA и ngRNA. Помимо необходимости разработки спейсера для обеих направляющих РНК, необходимо учитывать множество других параметров, которые могут повлиять на эффективность основного редактирования, в том числе: длина сайта связывания праймера (PBS), длина шаблона обратной транскрипции (RTT) и расстояние между ними.
Самая эффективная система первичного редактирования, описанная на сегодняшний день (называемая PE3), состоит из трех компонентов: слитого белка CRISPR-Cas9никаза и сконструированная обратная транскриптаза (RT), основная направляющая РНК для редактирования (pegRNA) и никирующая sgRNA (ngRNA) (приложение, рис. 1). ПегРНК нацелена на слияние никазы Cas9 и RT с определенным геномным локусом, но также гибридизуется с одноцепочечной ДНК с разрывом, не являющейся целевой нитью (NTS) внутри Cas9-индуцированной R-петли, и служит матрицей для обратной транскрипции в создать «лоскут», который опосредует индукцию точных генетических изменений (дополнение рис. 1a–c). нгРНК направляет слияние никазы Cas9 и RT, чтобы надрезать нить, противоположную лоскуту, и тем самым смещает репарацию в сторону желаемого изменения, закодированного в лоскуте (приложение, рис. 1d, e). Сложность системы PE3 требует много времени для ручного конструирования необходимых компонентов pegRNA и ngRNA. Помимо необходимости разработки спейсера для обеих направляющих РНК, необходимо учитывать множество других параметров, которые могут повлиять на эффективность основного редактирования, в том числе: длина сайта связывания праймера (PBS), длина шаблона обратной транскрипции (RTT) и расстояние между ними. сайты-мишени pegRNA и ngRNA.
сайты-мишени pegRNA и ngRNA.
Здесь мы представляем PrimeDesign, удобное веб-приложение (http://primedesign.pinellolab.org/) (рис. 1) и инструмент командной строки (https://github.com/pinellolab/PrimeDesign), который автоматизирует и, таким образом, упрощает дизайн pegRNAs и ngRNAs для единичного редактирования, комбинированного редактирования и скрининга мутагенеза по всему геному и насыщения. Мы используем PrimeDesign для создания PrimeVar, обширной базы данных потенциальных комбинаций направляющей РНК (pegRNA) и никирующей sgRNA (ngRNA) для установки или исправления > 68 500 патогенных генетических вариантов человека в базе данных ClinVar. Наконец, мы демонстрируем активность дизайнов pegRNA и ngRNA, рекомендованных PrimeDesign, посредством установки патогенных вариантов человека в клетки человека.
Рис. 1: Веб-приложение PrimeDesign. a PrimeDesign использует в качестве входных данных одну последовательность, кодирующую как исходную эталонную, так и желаемую отредактированную последовательности, b рекомендует кандидатную комбинацию пегРНК и нгРНК для установки интересующей редакции, c обеспечивает визуализацию последовательности интересующей редакции , выбранные конструкции пэгРНК и нгРНК и предсказанные вторичные структуры пегРНК, а d позволяет интерактивный дизайн как пегРНК, так и нгРНК, который можно загрузить в виде сводной таблицы.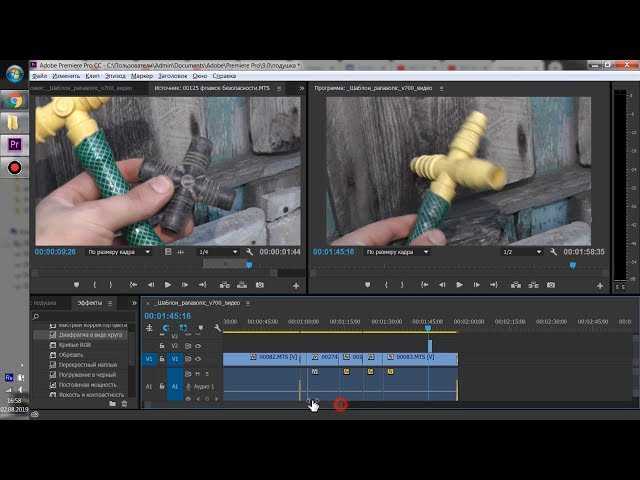
Полноразмерное изображение
Результаты
Возможности PrimeDesign
PrimeDesign использует один вход, который кодирует как исходный эталон, так и желаемые отредактированные последовательности (рис. 1a и приложение, примечание 1), рекомендует комбинацию пегРНК и нгРНК-кандидатов для установки интересующее редактирование (рис. 1b, приложение, рис. 2 и приложение, примечание 2), обеспечивает визуализацию последовательности основного события редактирования и предсказанных вторичных структур пегРНК (рис. 1с), а также перечисляет все возможные спейсеры пегРНК, расширения пегРНК , и нгРНК в пределах оптимизированных диапазонов параметров (ранее определенных группой Лю 1 ) для установки нужной редакции (рис. 1d). PrimeDesign позволяет пользователям ранжировать пегРНК на основе их предсказанной специфичности (оценка CFD 7 ), предоставляет важные аннотации для конструкций пегРНК (например, разрушение PAM) и нгРНК (например, PE3b), а также упрощает включение молчащих мутаций, разрушающих PAM, для улучшения редактирования. эффективность и чистота продукта (примечание 3 к приложению). Кроме того, PrimeDesign позволяет создавать пулы комбинаций пегРНК и нгРНК для скрининга полногеномного мутагенеза и мутагенеза с насыщением (http://primedesign.pinellolab.org/pooled) и ранжировать дизайны в соответствии с передовыми методами проектирования 1 . Функция мутагенеза с насыщением позволяет вводить мутации с разрешением одного основания или одной аминокислоты; PrimeDesign автоматически конструирует все правки в заданном пользователем диапазоне последовательностей и создает дизайны для установки этих правок (примечание 4 к приложению).
эффективность и чистота продукта (примечание 3 к приложению). Кроме того, PrimeDesign позволяет создавать пулы комбинаций пегРНК и нгРНК для скрининга полногеномного мутагенеза и мутагенеза с насыщением (http://primedesign.pinellolab.org/pooled) и ранжировать дизайны в соответствии с передовыми методами проектирования 1 . Функция мутагенеза с насыщением позволяет вводить мутации с разрешением одного основания или одной аминокислоты; PrimeDesign автоматически конструирует все правки в заданном пользователем диапазоне последовательностей и создает дизайны для установки этих правок (примечание 4 к приложению).
База данных PrimeVar
Чтобы проиллюстрировать полезность PrimeDesign, мы взяли патогенные генетические варианты человека из ClinVar 8 ( n = 69 481) и разработали пегРНК-кандидаты и нгРНК-кандидаты для коррекции этих патогенных аллелей. Из этих патогенных вариантов мы обнаружили, что 91,7% являются мишенями для по крайней мере одного спейсера pegRNA с максимальной длиной RTT 34 nt (рис. 2a и данные приложения 1). В среднем на каждый патогенный вариант было разработано 3,7 спейсера пегРНК, что представляет собой несколько вариантов первичного редактирования для исправления каждого варианта. Кроме того, 25,9% целевых патогенных вариантов включали по крайней мере одну пегРНК, которая нарушает последовательность PAM, что было связано с повышенной эффективностью редактирования и чистотой продукта. Стратегия PE3b (конструкция нгРНК, которые предпочтительно разрывают нередактируемую цепь после разрешения редактируемой нити лоскута) жизнеспособна для 790,5 % целевых вариантов (59,7 %, если учитывать только несовпадения в исходной последовательности; рис. 2b). Наконец, 11,9% целевых патогенных вариантов поддаются как стратегии разрушения PAM, так и стратегиям несовпадения семян PE3b.
2a и данные приложения 1). В среднем на каждый патогенный вариант было разработано 3,7 спейсера пегРНК, что представляет собой несколько вариантов первичного редактирования для исправления каждого варианта. Кроме того, 25,9% целевых патогенных вариантов включали по крайней мере одну пегРНК, которая нарушает последовательность PAM, что было связано с повышенной эффективностью редактирования и чистотой продукта. Стратегия PE3b (конструкция нгРНК, которые предпочтительно разрывают нередактируемую цепь после разрешения редактируемой нити лоскута) жизнеспособна для 790,5 % целевых вариантов (59,7 %, если учитывать только несовпадения в исходной последовательности; рис. 2b). Наконец, 11,9% целевых патогенных вариантов поддаются как стратегии разрушения PAM, так и стратегиям несовпадения семян PE3b.
a Распределение количества разработанных спейсеров пегРНК на вариант ClinVar. ПегРНК-кандидаты определяли на основе требования длины RTT <35 нуклеотидов и удлинения RT, чтобы иметь минимальную гомологию 5 нуклеотидов после редактирования. b 63 710 (91,7 %) целевых вариантов ClinVar, классифицированных по типу. Внутреннее кольцо (золото) представляет собой долю целевых вариантов по типу, где по крайней мере одна пегРНК может быть сконструирована для нарушения последовательности PAM (темное золото). Внешнее кольцо (серое) представляет долю целевых вариантов по типу, где по крайней мере одна нгРНК может быть сконструирована для стратегии PE3b, где несоответствие заключается в исходной последовательности (PAM-проксимальные нуклеотиды 1–10) (темно-серый). Подробности см. в дополнительных данных 1.
b 63 710 (91,7 %) целевых вариантов ClinVar, классифицированных по типу. Внутреннее кольцо (золото) представляет собой долю целевых вариантов по типу, где по крайней мере одна пегРНК может быть сконструирована для нарушения последовательности PAM (темное золото). Внешнее кольцо (серое) представляет долю целевых вариантов по типу, где по крайней мере одна нгРНК может быть сконструирована для стратегии PE3b, где несоответствие заключается в исходной последовательности (PAM-проксимальные нуклеотиды 1–10) (темно-серый). Подробности см. в дополнительных данных 1.
Полноразмерное изображение
Чтобы сделать все эти схемы редактирования праймов ClinVar более доступными, мы создали PrimeVar (http://primedesign.pinellolab.org/primevar), всеобъемлющую базу данных с возможностью поиска комбинаций пегРНК и нгРНК для установки или исправления. >68 500 патогенных генетических вариантов человека. С помощью референсного номера SNP dbSNP (rs#) или идентификатора вариации ClinVar легко доступны пегРНК-кандидаты и нгРНК в диапазоне длин PBS (10–17 нуклеотидов) и RTT (10–80 нуклеотидов).
Установка патогенных вариантов в клетки человека
Наконец, мы протестировали рекомендуемые комбинации пегРНК и нгРНК от PrimeDesign, чтобы установить 20 различных патогенных вариантов человека, связанных с генетическими заболеваниями, включая гемофилию А, мышечную дистрофию Дюшенна (МДД), МПС I и II и Болезнь Фабри в клетках HEK293T (рис. 3a, данные 2 приложения и примечание 2 к приложению). Мы наблюдали установку желаемого редактирования со средней частотой 10% или более для 7 из 20 (35%) целевых сайтов и со средней частотой 1–10% для 6 из 20 (30%) целевых сайтов. Для подмножества из семи желаемых мутаций мы разработали дополнительные пегРНК для оценки различий между PE3 и PE3b (рис. 3b). Как правило, мы наблюдали смешанные тенденции частоты желаемого редактирования и умеренное снижение побочных продуктов для PE3b по сравнению с PE3. Наконец, мы разработали подмножество из четырех дополнительных пегРНК, которые ввели молчащие мутации, разрушающие PAM (в дополнение к целевому патогенному варианту), и обнаружили, что эти конструкции приводят к среднему 1,8-кратному увеличению частоты желаемого редактирования (рис. 3c). ).
3c). ).
a Обзор эффективности первичного редактирования для установки 20 патогенных вариантов человека в клетки HEK293T с использованием рекомендаций PrimeDesign. Требуемое редактирование относится к чтениям секвенирования, содержащим только интересующее редактирование, в то время как побочный продукт относится к чтениям секвенирования, содержащим любые мутации за пределами только интересующего редактирования (т. е. вставки, желаемое редактирование и вставки). б Сравнение стратегий редактирования PE3 и PE3b. c Оценка влияния молчащих мутаций, нарушающих PAM, на эффективность основного редактирования. Среднее ± с.д. из n = 3 независимых биологических повтора. Некоторые данные, показанные в a , также представлены в b и c . Подробности см. в дополнительных данных 2. Исходные данные доступны в файле исходных данных.
Полноразмерное изображение
Обсуждение
Таким образом, PrimeDesign представляет собой всеобъемлющий и общий метод простого и автоматизированного проектирования пегРНК и нгРНК. Наш тест pegRNAs и ngRNAs, разработанных PrimeDesign для создания различных правок, показывает, что не все проекты дают желаемые изменения с высокой частотой, поэтому пользователям PrimeDesign может потребоваться уточнить выбор pegRNA даже после проверки первоначальных рекомендаций. Тем не менее, PrimeDesign должен значительно упростить сложный процесс разработки компонентов-кандидатов на первичное редактирование и, таким образом, расширить использование и доступность этой мощной и важной технологии 9.0235 9,10,11 .
Методы.
Молекулярное клонирование. Для клонирования пегРНК (дополнительные данные 2) фрагменты двухцепочечной ДНК для каркаса пегРНК, спейсера и 3′-удлинения были сформированы путем отжига олигонуклеотидов с совместимыми выступами для лигирования.
 Затем фрагменты лигировали с использованием лигазы Т4 (NEB) и клонировали в расщепленную BsaI pUC19.основанный на hU6-pegRNA-gg-акцепторный вектор входа (Addgene № 132777). Для клонирования гРНК (нгРНК) спейсерные олигонуклеотиды дуплексировали и лигировали в расщепленный BsmbI pUC19-основанный на hU6-SpCas9 вектор входа гРНК BPK1520 (Addgene № 65777). Все плазмиды pegRNA и ngRNA трансформировали в химически компетентную E.coli (XL1-Blue, Agilent). Плазмиды, используемые для трансфекции, были приготовлены в миди- (PE2) или мини-препарированном виде (гРНК) с использованием наборов Qiagen midi plus или miniprep.
Затем фрагменты лигировали с использованием лигазы Т4 (NEB) и клонировали в расщепленную BsaI pUC19.основанный на hU6-pegRNA-gg-акцепторный вектор входа (Addgene № 132777). Для клонирования гРНК (нгРНК) спейсерные олигонуклеотиды дуплексировали и лигировали в расщепленный BsmbI pUC19-основанный на hU6-SpCas9 вектор входа гРНК BPK1520 (Addgene № 65777). Все плазмиды pegRNA и ngRNA трансформировали в химически компетентную E.coli (XL1-Blue, Agilent). Плазмиды, используемые для трансфекции, были приготовлены в миди- (PE2) или мини-препарированном виде (гРНК) с использованием наборов Qiagen midi plus или miniprep.Клеточная культура
STR-аутентифицированный HEK29Клетки 3T (CRL-3216) выращивали в среде Игла, модифицированной Дульбекко (DMEM, Gibco), содержащей 10% эмбриональной бычьей сыворотки (FBS, Gibco) и 1% пенициллин-стрептомицинового антибиотика (Gibco). Клетки хранили в инкубаторе 2 с 5% СО при 37 °С. Клетки пассировали каждые 2-3 дня, когда клетки достигали 80% слияния. Клетки не превышали пассаж 13 для всех повторностей в этом эксперименте. Тестирование сред для культивирования клеток на микоплазму проводилось каждые 4 недели с помощью набора для обнаружения микоплазм MycoAlert PLUS (Lonza) и показало отрицательные результаты на протяжении всего исследования.
Клетки не превышали пассаж 13 для всех повторностей в этом эксперименте. Тестирование сред для культивирования клеток на микоплазму проводилось каждые 4 недели с помощью набора для обнаружения микоплазм MycoAlert PLUS (Lonza) и показало отрицательные результаты на протяжении всего исследования.
Трансфекции
Клетки HEK293T высевали в 96-луночные планшеты для культивирования клеток с плоским дном (Corning) для обработки ПЭ при 1,2 × 10 4 клеток/лунку. Трансфекцию проводили через 18–24 ч после посева 30 нг плазмиды PE2, 10 нг пегРНК и 3,3 нг плазмиды нгРНК на трансфекцию (на лунку, в 96-луночном планшете). TransIT-X2 (Mirus) использовали в качестве реагента для липофекции в количестве 0,3 мкл на трансфекцию.
Экстракция ДНК
После трансфекции (72 часа) клетки HEK293T промывали с помощью 1x PBS (Corning) и лизировали 43,5 мкл буфера для лизиса гДНК (100 мМ Трис, 200 мМ NaCl, 5 мМ ЭДТА, 0,05% SDS) , 1,25 мкл 1 М ДТТ (Sigma) и 5,25 мкл протеиназы К на лунку в течение 9Эксперименты на 6-луночном планшете. Планшеты помещали в шейкер (500 об/мин) при 55°C на ночь и экстрагировали гДНК с использованием 1,5-кратных парамагнитных шариков. Гранулы со связанной гДНК трижды промывали 70% этанолом с использованием лабораторной автоматизированной рабочей станции Biomek FX p (Beckman Coulter), а затем элюировали в 35 мкл 0,1x EB-буфера (Qiagen).
Планшеты помещали в шейкер (500 об/мин) при 55°C на ночь и экстрагировали гДНК с использованием 1,5-кратных парамагнитных шариков. Гранулы со связанной гДНК трижды промывали 70% этанолом с использованием лабораторной автоматизированной рабочей станции Biomek FX p (Beckman Coulter), а затем элюировали в 35 мкл 0,1x EB-буфера (Qiagen).
Целевое секвенирование ампликонов
Концентрации гДНК в нескольких образцах из разных пегРНК/репликатов измеряли с использованием набора Qubit dsDNA HS Assay Kit (Thermo Fisher). Первая ПЦР была проведена для амплификации интересующих областей генома (200–250 п.н.) с использованием 10–20 нг гДНК. Праймеры для PCR1 включали совместимые с Illumina последовательности адаптеров (дополнительные данные 2). Затем использовали устройство для считывания микропланшетов Synergy HT (BioTek) при 485/528 нм с системой количественного определения двухцепочечной ДНК Quantifluor (Promega) для измерения концентрации первых продуктов ПЦР. Затем продукты ПЦР из разных геномных ампликонов объединяли и очищали парамагнитными шариками 0,7x. Вторая ПЦР была проведена для прикрепления уникальных штрих-кодов к каждому ампликону с использованием 50–200 нг объединенных продуктов ПЦР1 и штрих-кодов, которые соответствуют индексам Illumina TruSeq CD. Продукты PCR2 снова очищали парамагнитными гранулами 0,7x и измеряли с помощью системы Quantifluor перед окончательным объединением. Окончательную библиотеку секвенировали с использованием Illumina Miseq (набор Miseq Reagent Kit v.2; 300 циклов, 2 × 150 п.н., парный конец). Файлы FASTQ были загружены с сайта BaseSpace (Illumina).
Вторая ПЦР была проведена для прикрепления уникальных штрих-кодов к каждому ампликону с использованием 50–200 нг объединенных продуктов ПЦР1 и штрих-кодов, которые соответствуют индексам Illumina TruSeq CD. Продукты PCR2 снова очищали парамагнитными гранулами 0,7x и измеряли с помощью системы Quantifluor перед окончательным объединением. Окончательную библиотеку секвенировали с использованием Illumina Miseq (набор Miseq Reagent Kit v.2; 300 циклов, 2 × 150 п.н., парный конец). Файлы FASTQ были загружены с сайта BaseSpace (Illumina).
Анализ
Данные секвенирования ампликонов были проанализированы с помощью CRISPResso версии 2.0.42 с режимом HDR. Нисходящий анализ был получен из CRISPResso_quantification_of_editing_frequency.txt. Частота желаемого редактирования была определена путем взятия HDR Unmodified и деления на Reads_aligned_all_amplicons, а частота побочного продукта была определена путем взятия суммы HDR Modified, Reference Modified, Ambiguous и деление на Reads_aligned_all_amplicons.
Анализ PrimeDesign вариантов ClinVar
Доступ к базе данных ClinVar был осуществлен 8 апреля -го 2020 года. Варианты были отфильтрованы со следующими условиями: (1) включали действительную координату GRCh48/hg38, (2) помечены как патогенные для столбца « ClinicalSignificance» и (3) содержал уникальный идентификатор, определяемый конкатенацией столбцов «Имя», «RS# (dbSNP)» и «VariationID». Все варианты с неоднозначным кодом IUPAC были преобразованы в отдельные записи с недвусмысленными основаниями для последующего анализа. После этих шагов общее количество вариантов ClinVar составило 69.,481. Входные последовательности были отформатированы для всех записей как для установки, так и для исправления этих патогенных вариантов. После запуска PrimeDesign на вариантах ClinVar кандидаты на дизайн pegRNA были отфильтрованы по двум критериям: (1) максимальная длина RTT 34 нуклеотида и (2) минимальная гомология 5 нуклеотидов после редактирования. ПегРНК с нарушенными аннотациями PAM имеют мутации в динуклеотиде GG мотива NGG, а нгРНК с PE3b , PE3b без семян и PE3b с семенами 9Аннотации 0026 имеют несовпадения в любом месте протоспейсера, несовпадения за пределами проксимальных нуклеотидов 1–10 PAM или несовпадения в проксимальных нуклеотидах 1–10 PAM соответственно.
Создание базы данных PrimeVar
Отфильтрованные варианты ClinVar из анализа PrimeDesign были использованы для создания всеобъемлющей базы данных потенциальных комбинаций pegRNA и ngRNA. Доступны первичные конструкции редактирования для установки и исправления патогенных генетических вариантов человека. PrimeDesign был запущен с диапазоном длины PBS от 10 до 17 нуклеотидов, диапазоном длины RTT от 10 до 80 нуклеотидов и диапазоном расстояний нгРНК от 0 до 100 нуклеотидов. Все конструкции пегРНК и нгРНК для каждого варианта хранятся в PrimeVar (http://primedesign.pinellolab.org/primevar).
Сводка отчета
Дополнительную информацию о дизайне исследования можно найти в Сводке отчета об исследовании природы, связанной с этой статьей.
Доступность данных
Все данные секвенирования целевого ампликона были депонированы под регистрационным номером BioProject PRJNA688137 в архиве считывания последовательностей (SRA), доступном по адресу: https://www. ncbi.nlm.nih.gov/bioproject/PRJNA688137. Информация, касающаяся клинических вариантов в рукописи, доступна по адресу: https://www.ncbi.nlm.nih.gov/clinvar/. Исходные данные приводятся вместе с настоящей статьей.
ncbi.nlm.nih.gov/bioproject/PRJNA688137. Информация, касающаяся клинических вариантов в рукописи, доступна по адресу: https://www.ncbi.nlm.nih.gov/clinvar/. Исходные данные приводятся вместе с настоящей статьей.
Доступность кода
PrimeDesign 12 был использован для разработки pegRNAs и ngRNAs (https://github.com/pinellolab/PrimeDesign). Данные секвенирования ампликонов анализировали с помощью CRISPResso версии 2.0.42 с режимом HDR (https://github.com/pinellolab/CRISPResso2).
История изменений
03 марта 2021 г.
В исходной версии этой статьи имя и фамилия автора Дж. Кит Джунг были помечены неправильно, что привело к неправильному индексированию. Сейчас эта ошибка исправлена. Версии PDF и HTML правильно отображали имя на момент публикации.
Ссылки
Anzalone, A.V. et al. Редактирование генома с помощью поиска и замены без двухцепочечных разрывов или донорской ДНК.
 Природа 576 , 149–157 (2019).
Природа 576 , 149–157 (2019).ОБЪЯВЛЕНИЯ КАС Статья Google ученый
Конг, Л. и др. Мультиплексная инженерия генома с использованием систем CRISPR/Cas. Наука 339 , 819–823 (2013).
ОБЪЯВЛЕНИЯ КАС Статья Google ученый
Мали, П. и др. Инженерия генома человека с помощью РНК с помощью Cas9. Наука 339 , 823–826 (2013).
ОБЪЯВЛЕНИЯ КАС Статья Google ученый
Хван, В. и др. Эффективное редактирование генома у рыбок данио с использованием системы CRISPR-Cas. Нац. Биотехнолог. 31 , 227–229 (2013).
КАС Статья Google ученый
Komor, A.C. et al. Программируемое редактирование целевого основания в геномной ДНК без расщепления двухцепочечной ДНК.
 Природа 533 , 420–424 (2016).
Природа 533 , 420–424 (2016).ОБЪЯВЛЕНИЯ КАС Статья Google ученый
Gaudelli, N.M. et al. Программируемое базовое редактирование AT в GC в геномной ДНК без расщепления ДНК. Природа 551 , 464–471 (2017).
ОБЪЯВЛЕНИЯ КАС Статья Google ученый
Доенч, Дж. Г. и др. Оптимизированный дизайн sgRNA для максимальной активности и минимизации нецелевых эффектов CRISPR-Cas9. Нац. Биотехнолог. 34 , 184–191 (2016).
Landrum, M.J. et al. ClinVar: общедоступный архив интерпретаций клинически значимых вариантов. Рез. нуклеиновых кислот. 44 , Д862–Д868 (2016).
КАС Статья Google ученый
Лин, К. и др. Основное редактирование генома риса и пшеницы.
 Нац. Биотехнолог. 38 , 582–585 (2020).
Нац. Биотехнолог. 38 , 582–585 (2020).Лю, Ю. и др. Эффективное создание моделей мышей с помощью основной системы редактирования. Сотовый Дисков. 6 , 27 (2020).
Kim, H.K. et al. Прогнозирование эффективности основных направляющих РНК для редактирования в клетках человека. Нац. Биотехнолог. https://doi.org/10.1038/s41587-020-0677-y (2020 г.).
Hsu, J.Y.H. et al. Программное обеспечение PrimeDesign для быстрого и упрощенного дизайна основных направляющих РНК для редактирования, pinellolab/PrimeDesign bioRxiv https://doi.org/10.5281/zenodo.4429461 (2021).
Ссылки на скачивание
Благодарности
LP поддерживается Премией развития карьеры Национального института исследования генома человека (NHGRI) (R00HG008399), премией Genomic Innovator Award (R35HG010717) и CEGS RM1HG009490. Дж.К.Дж. поддерживается NIH R35 GM118158, NIH RM1 HG009490, Роберта Б. Колвина, доктора медицины, заведующего кафедрой патологии, и премии Десмонда и Энн Хитвуд для ученых-исследователей MGH. Д.Р.Л. поддерживается Институтом трансформационных технологий в здравоохранении Меркина, грантами Национального института здравоохранения США U01AI142756, RM1HG009490, R01EB022376 и R35GM118062, а также HHMI. А.В.А. признает постдокторскую стипендию Джейн Коффин Чайлдс. Дж.Г. финансировалось Deutsche Forschungsgemeinschaft (DFG, Немецкий исследовательский фонд) – номер проекта 416375182.
Колвина, доктора медицины, заведующего кафедрой патологии, и премии Десмонда и Энн Хитвуд для ученых-исследователей MGH. Д.Р.Л. поддерживается Институтом трансформационных технологий в здравоохранении Меркина, грантами Национального института здравоохранения США U01AI142756, RM1HG009490, R01EB022376 и R35GM118062, а также HHMI. А.В.А. признает постдокторскую стипендию Джейн Коффин Чайлдс. Дж.Г. финансировалось Deutsche Forschungsgemeinschaft (DFG, Немецкий исследовательский фонд) – номер проекта 416375182.
Информация об авторе
Авторы и филиалы
Отделение биологической инженерии Массачусетского технологического института, Кембридж, Массачусетс, США
Джонатан Ю. Хсу
Отделение молекулярной патологии, Массачусетский госпиталь общего профиля 903, Чарлстаун, Массачусетс Y. Hsu, Julian Grünewald, Regan Szalay, Justine Shih, Kin Chung Lam, Karl Petri, J. Keith Joung и Luca Pinello
Центр исследований рака и Центр вычислительной и интегративной биологии, Massachusetts General Hospital, Charlestown, MA , США
Джонатан Ю.
 Хсу, Джулиан Грюневальд, Реган Салай, Жюстин Ши, Кин Чунг Лам, Карл Петри и Дж. Кейт Джунг
Хсу, Джулиан Грюневальд, Реган Салай, Жюстин Ши, Кин Чунг Лам, Карл Петри и Дж. Кейт ДжунгКафедра патологии, Гарвардская медицинская школа, Бостон, Массачусетс, США
Джулиан Гринвальд, Чунг Лам, Карл Петри, Дж. Кит Юнг и Лука Пинелло
Меркин Институт трансформационных технологий в здравоохранении, Институт Броуда Гарварда и Массачусетского технологического института, Кембридж, Массачусетс, США
Эндрю В. Анзалоне, Макс В. Шен и Дэвид Р. Лю
Кафедра химии и химической биологии, Гарвардский университет, Кембридж, Массачусетс, США
Эндрю В. Анзалон, Макс В. Шен и Дэвид Р. Лю
Медицинский институт Говарда Хьюза, Гарвардский университет, Кембридж, Массачусетс , США
Эндрю В. Анзалоне, Макс В. Шен и Дэвид Р. Лю
Программа вычислительной и системной биологии, Массачусетский технологический институт, Кембридж, Массачусетс, США
Макс В. Шен
Институт Гарварда и Массачусетского технологического института, Кембридж, Массачусетс, США
Лука Пинелло
Авторы
- Джонатан Ю.
 Хсу
ХсуПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Julian Grünewald
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Regan Szalay
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
- Justine Shih
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Andrew V. Anzalone
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Kin Chung Lam
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
- Max W. Shen
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Karl Petri
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- David R.
 Liu
LiuПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- J. Keith Joung
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Luca Pinello
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Contributions
J.Y.H. разработал ПраймДизайн. Д.И.Х. и Дж.Г. проектировал эксперименты. Р.С. и Дж.С. провел эксперименты и проанализировал данные. A.V.A., J.G., K.P. и K.C.L предоставили отзывы во время разработки PrimeDesign. М.В.С. участвовал в анализе ClinVar. Л.П., Дж.К.Дж. и Д.Р.Л. курировал проект и давал обратную связь и рекомендации. J.Y.H., L.P., J.K.J. и D.R.L. написал рукопись при участии всех других авторов.
Авторы переписки
Переписка с Дж. Кит Джунг или Лука Пинелло.
Декларации этики
Конкурирующие интересы
J. K.J. имеет финансовые интересы в Beam Therapeutics, Chroma Medicine (f/k/a YKY, Inc.), Editas Medicine, Excelsior Genomics, Pairwise Plants, Poseida Therapeutics, SeQure Dx, Inc., Transposagen Biopharmaceuticals и Verve Therapeutics (f/k/ Энкадия). Интересы J.K.J. были рассмотрены и управляются Massachusetts General Hospital and Partners HealthCare в соответствии с их политикой конфликта интересов. Дж.К.Дж. является соавтором патентов и патентных заявок, описывающих различные технологии редактирования генов. Д.Р.Л. является консультантом и соучредителем компаний Prime Medicine, Beam Therapeutics, Pairwise Plants и Editas Medicine, которые используют редактирование генома. LP имеет финансовые интересы в Edilytics и SeQure Dx, Inc. Интересы LP были проверены и управляются Massachusetts General Hospital and Partners HealthCare в соответствии с их политикой конфликта интересов. Все остальные авторы заявляют об отсутствии конкурирующих интересов.
K.J. имеет финансовые интересы в Beam Therapeutics, Chroma Medicine (f/k/a YKY, Inc.), Editas Medicine, Excelsior Genomics, Pairwise Plants, Poseida Therapeutics, SeQure Dx, Inc., Transposagen Biopharmaceuticals и Verve Therapeutics (f/k/ Энкадия). Интересы J.K.J. были рассмотрены и управляются Massachusetts General Hospital and Partners HealthCare в соответствии с их политикой конфликта интересов. Дж.К.Дж. является соавтором патентов и патентных заявок, описывающих различные технологии редактирования генов. Д.Р.Л. является консультантом и соучредителем компаний Prime Medicine, Beam Therapeutics, Pairwise Plants и Editas Medicine, которые используют редактирование генома. LP имеет финансовые интересы в Edilytics и SeQure Dx, Inc. Интересы LP были проверены и управляются Massachusetts General Hospital and Partners HealthCare в соответствии с их политикой конфликта интересов. Все остальные авторы заявляют об отсутствии конкурирующих интересов.
Дополнительная информация
Информация о рецензировании Nature Communications благодарит Андреа Вентура и других анонимных рецензентов за их вклад в рецензирование этой работы. Доступны отчеты рецензентов.
Доступны отчеты рецензентов.
Примечание издателя Springer Nature остается нейтральной в отношении юрисдикционных претензий в опубликованных картах и институциональной принадлежности.
Дополнительная информация
Дополнительная информация
Файл рецензирования
Сводка отчетности
Описание дополнительных дополнительных файлов
Дополнительные данные 1
Дополнительные данные 2
Дополнительные данные 2
. и разрешения
Открытый доступ Эта статья находится под лицензией Creative Commons Attribution 4.0 International License, которая разрешает использование, совместное использование, адаптацию, распространение и воспроизведение на любом носителе или в любом формате при условии, что вы укажете подлинного автора. s) и источник, предоставить ссылку на лицензию Creative Commons и указать, были ли внесены изменения. Изображения или другие сторонние материалы в этой статье включены в лицензию Creative Commons для статьи, если иное не указано в кредитной строке материала. Если материал не включен в лицензию Creative Commons статьи, а ваше предполагаемое использование не разрешено законом или выходит за рамки разрешенного использования, вам необходимо получить разрешение непосредственно от правообладателя. Чтобы просмотреть копию этой лицензии, посетите http://creativecommons.org/licenses/by/4.0/.
Изображения или другие сторонние материалы в этой статье включены в лицензию Creative Commons для статьи, если иное не указано в кредитной строке материала. Если материал не включен в лицензию Creative Commons статьи, а ваше предполагаемое использование не разрешено законом или выходит за рамки разрешенного использования, вам необходимо получить разрешение непосредственно от правообладателя. Чтобы просмотреть копию этой лицензии, посетите http://creativecommons.org/licenses/by/4.0/.
Перепечатка и разрешения
Об этой статье
Эта статья цитируется
Разработка и проведение экспериментов по первичному редактированию клеток млекопитающих.
- Джордан Л. Доман
- Александр А.
 Соуза
Соуза - Дэвид Р. Лю
Природные протоколы (2022)
Высокоэффективное первичное редактирование путем введения мутаций того же смысла в пегРНК или стабилизации ее структуры.
- Сяоса Ли
- Лина Чжоу
- Цзя Чен
Nature Communications (2022)
Random-PE: эффективная интеграция случайных последовательностей в геном млекопитающих путем первичного редактирования.
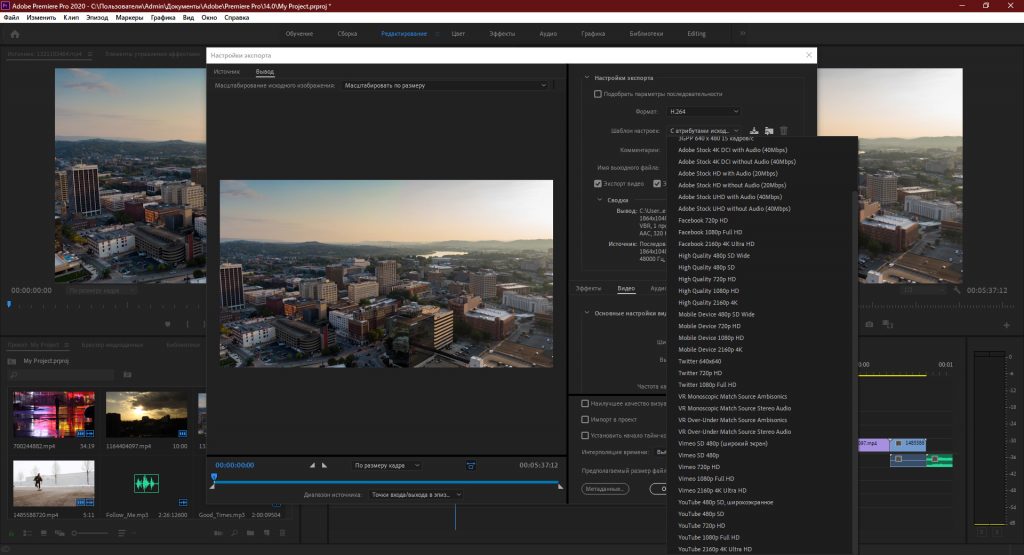
- Яогэ Цзяо
- Лифан Чжоу
- Шаохуа Яо
Молекулярная биомедицина (2021)
Easy-Prime: инструмент для проектирования простых редакторов на основе машинного обучения.
- Ичао Ли
- Цзинцзин Чен
- Юн Ченг
Биология генома (2021)
Высокоэффективное первичное редактирование с оптимизированными парными пегРНК в растениях
- Цюпэн Линь
- Шуай Джин
- Цайся Гао
Природные биотехнологии (2021)
Комментарии
Отправляя комментарий, вы соглашаетесь соблюдать наши Условия и Правила сообщества. Если вы обнаружите что-то оскорбительное или не соответствующее нашим условиям или правилам, отметьте это как неприемлемое.
Если вы обнаружите что-то оскорбительное или не соответствующее нашим условиям или правилам, отметьте это как неприемлемое.
pegIT — веб-инструмент для простого редактирования | Исследование нуклеиновых кислот
Журнальная статья
Мадс Вальдемар Андерсон,
Мэдс Вальдемар Андерсон
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google ученый
Якоб Халдруп,
Джейкоб Халдруп
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google ученый
Эмиль Аагард Томсен,
Эмиль Аагард Томсен
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google ученый
Йонас Холст Вольф,
Йонас Холст Вольф
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google ученый
Якоб Гим Миккельсен
Якоб Гим Миккельсен
Ищите другие работы этого автора на:
Оксфордский академический
пабмед
Google ученый
Nucleic Acids Research , том 49, выпуск W1, 2 июля 2021 г. , страницы W505–W509, https://doi.org/10.1093/nar/gkab427
, страницы W505–W509, https://doi.org/10.1093/nar/gkab427
Опубликовано:
01 июня 2021 г.
История статьиПолучено:
09 марта 2021 г.
Получена редакция:
12 апреля 2021 г.
Принято:
11 мая 2021 г.
Опубликовано:
01 июня 2021 г. 9008 PDF
- Содержание статьи
- Рисунки и таблицы
- видео
- Аудио
- Дополнительные данные
Цитировать
Cite
Мадс Вальдемар Андерсон, Якоб Халдруп, Эмиль Аагард Томсен, Йонас Холст Вольф, Якоб Гим Миккельсен, pegIT — веб-инструмент для простого редактирования, Nucleic Acids Research , том 49, выпуск W1, 2 июля 2021 г. , страницы W505–W509, https://doi.org/10.1093/nar/gkab427
, страницы W505–W509, https://doi.org/10.1093/nar/gkab427
Выберите формат Выберите format.ris (Mendeley, Papers, Zotero).enw (EndNote).bibtex (BibTex).txt (Medlars, RefWorks)
Закрыть
Разрешения
- Электронная почта
- Твиттер
- Фейсбук
- Подробнее
Фильтр поиска панели навигации Исследование нуклеиновых кислотЭтот выпускЖурналы NARНаука и математикаКнигиЖурналыOxford Academic Термин поиска мобильного микросайта
Закрыть
Фильтр поиска панели навигации Исследование нуклеиновых кислотЭтот выпускЖурналы NARНаука и математикаКнигиЖурналыOxford Academic Термин поиска на микросайте
Advanced Search
Abstract
Прайм-редактирование (PE) — это новый метод редактирования генома, основанный на CRISPR, который обеспечивает точное редактирование без разрывов двухцепочечной ДНК. PE, опосредованная Cas9-слитый белок с обратной транскриптазой основан на двухфункциональных основных направляющих РНК для редактирования (pegRNAs), которые служат как направляющими молекулами, так и матрицами, несущими желаемые изменения. Из-за такого разнообразия функций ручная разработка pegRNA подвержена ошибкам и не подходит для крупномасштабных установок. Здесь мы представляем pegIT, удобный веб-инструмент для быстрого дизайна pegRNA для многочисленных пользовательских правок, включая крупномасштабные настройки. pegIT находится в свободном доступе по адресу https://pegit.giehmlab.dk.
PE, опосредованная Cas9-слитый белок с обратной транскриптазой основан на двухфункциональных основных направляющих РНК для редактирования (pegRNAs), которые служат как направляющими молекулами, так и матрицами, несущими желаемые изменения. Из-за такого разнообразия функций ручная разработка pegRNA подвержена ошибкам и не подходит для крупномасштабных установок. Здесь мы представляем pegIT, удобный веб-инструмент для быстрого дизайна pegRNA для многочисленных пользовательских правок, включая крупномасштабные настройки. pegIT находится в свободном доступе по адресу https://pegit.giehmlab.dk.
Графический реферат
Открыть в новой вкладкеСкачать слайд
pegIT — это свободно доступный удобный веб-инструмент, который позволяет легко и эффективно создавать pegRNA для многочисленных, в том числе крупномасштабных, определяемых пользователем правок.
ВВЕДЕНИЕ
CRISPR/Cas9 (1,2) широко используется в молекулярной биологии и биомедицинских исследованиях. Первоначально технология была разработана для разрушения отдельных генов с помощью вставок, происходящих из негомологичного соединения концов (NHEJ), что привело к созданию полногеномных нокаутных библиотек (3). Кроме того, прецизионное редактирование, опосредованное добавлением матриц для направленной по гомологии репарации (HDR), стало инновационным в терапевтическом редактировании генов, в то время как развитие каталитически нарушенных и слитых Cas9варианты еще больше расширили набор инструментов CRISPR. Следовательно, новые методы редактирования генома используют ДНК-связывающие свойства эффекторов CRISPR, чтобы направлять ферменты, модифицирующие ДНК, к желаемому локусу-мишени. Слияние ферментов нуклеотидных деаминаз с каталитически нарушенными вариантами Cas9 привело к созданию редакторов оснований (4–6), которые могут устанавливать четыре переходные мутации (A→G, G→A, C→T и T→C) и две трансверсионные мутации ( C→G и G→C) с более высокой эффективностью за счет механизмов редактирования, которые с меньшей вероятностью вызывают нежелательные мутации, чем пути восстановления, включающие HDR.
Первоначально технология была разработана для разрушения отдельных генов с помощью вставок, происходящих из негомологичного соединения концов (NHEJ), что привело к созданию полногеномных нокаутных библиотек (3). Кроме того, прецизионное редактирование, опосредованное добавлением матриц для направленной по гомологии репарации (HDR), стало инновационным в терапевтическом редактировании генов, в то время как развитие каталитически нарушенных и слитых Cas9варианты еще больше расширили набор инструментов CRISPR. Следовательно, новые методы редактирования генома используют ДНК-связывающие свойства эффекторов CRISPR, чтобы направлять ферменты, модифицирующие ДНК, к желаемому локусу-мишени. Слияние ферментов нуклеотидных деаминаз с каталитически нарушенными вариантами Cas9 привело к созданию редакторов оснований (4–6), которые могут устанавливать четыре переходные мутации (A→G, G→A, C→T и T→C) и две трансверсионные мутации ( C→G и G→C) с более высокой эффективностью за счет механизмов редактирования, которые с меньшей вероятностью вызывают нежелательные мутации, чем пути восстановления, включающие HDR.
В последнее время первичное редактирование (PE), описанное как технология редактирования генома «поиск и замена», позволяет осуществлять целенаправленные вставки, делеции и все возможные переходы и трансверсии без двухцепочечных разрывов (DSB) или необходимости в экзогенной донорской ДНК. шаблоны. Основные редакторы (PE) состоят из никазы Cas9-H840A (Cas9n), слитой с обратной транскриптазой (RT). Комплекс Cas9n/RT запрограммирован направляющей РНК главного редактора (pegRNA), которая состоит из трех элементов: (i) обычного протоспейсера, определяющего геномный целевой локус, (ii) сайта связывания праймера (PBS), который служит в качестве шаблон для RT и (iii) шаблон RT, который содержит желаемое геномное редактирование (рис. 1). Cas9никаза, управляемая специфической пегРНК-мишенью, разрезает цепь, содержащую PAM, с образованием лоскута одноцепочечной ДНК (оцДНК). PBS, комплементарный лоскуту одноцепочечной ДНК, гибридизуется и инициирует обратную транскрипцию матрицы RT, таким образом, включая нуклеотиды, содержащие желаемое редактирование. Для повышения эффективности можно добавить дополнительную одиночную направляющую РНК (nsgRNA) для разрыва, метод, названный PE3. Использование nsgRNAs, специфичных для редактируемой цепи, предотвращает одновременный разрыв и, следовательно, образование DSB и indels, стратегия, названная PE3b. Первичное редактирование имеет огромный потенциал для лечения генетических заболеваний, создания трансгенных растений и мышей (7–9) и множество других приложений.
Для повышения эффективности можно добавить дополнительную одиночную направляющую РНК (nsgRNA) для разрыва, метод, названный PE3. Использование nsgRNAs, специфичных для редактируемой цепи, предотвращает одновременный разрыв и, следовательно, образование DSB и indels, стратегия, названная PE3b. Первичное редактирование имеет огромный потенциал для лечения генетических заболеваний, создания трансгенных растений и мышей (7–9) и множество других приложений.
Рис. 1.
Открыть в новой вкладкеСкачать слайд
Схематическое представление первичного редактирования. ( A ) Первичные редакторы (PE) состоят из слитого белка никазы Cas9 и обратной транскриптазы (RT). Этот комплекс связан с первичной направляющей РНК для редактирования (pegRNA), которая направляет комплекс PE к целевой последовательности и кодирует желаемую генетическую модификацию. ( B ) Комплекс PE связывается с локусом-мишенью и разрывает цепь PAM. ( C ) Гибридизация 3′-конца цепи PAM и 3′-конца pegRNA; сайт связывания праймеров (PBS) позволяет инициировать обратную транскрипцию с помощью RT. ( D ) RT осуществляет обратную транскрипцию последовательности пегРНК в 3′-цепь PAM. ( E ) Комплекс PE диссоциирует, и механизмы клеточной репарации расщепляют лоскуты ДНК, чтобы ввести редактирование. ( F ) Комплекс PE диссоциирует, и механизмы клеточной репарации разрешают лоскуты ДНК, чтобы ввести редактирование в виде гетеродуплекса. ( G ) Надрез неотредактированной нити смещает репарацию в сторону включения редактирования, что приводит к отредактированному гомодуплексу ( H ).
( D ) RT осуществляет обратную транскрипцию последовательности пегРНК в 3′-цепь PAM. ( E ) Комплекс PE диссоциирует, и механизмы клеточной репарации расщепляют лоскуты ДНК, чтобы ввести редактирование. ( F ) Комплекс PE диссоциирует, и механизмы клеточной репарации разрешают лоскуты ДНК, чтобы ввести редактирование в виде гетеродуплекса. ( G ) Надрез неотредактированной нити смещает репарацию в сторону включения редактирования, что приводит к отредактированному гомодуплексу ( H ).
По сравнению с современной технологией CRISPR/Cas9 и редактированием генов с использованием базовых редакторов, которые полагаются на sgRNAs для точного нацеливания, первичное редактирование основано на направляющих молекулах РНК, содержащих редактируемую последовательность. Следовательно, в то время как предыдущие технологии CRISPR требуют только создания олигонуклеотидов для протоспейсера, пегРНК также требуют создания удлиняющей последовательности, состоящей из сайта связывания праймера (PBS) и матрицы RT (RTT), которая кодирует желаемое редактирование. С дополнительными функциональными свойствами пегРНК это еще больше усложняет процесс проектирования.
С дополнительными функциональными свойствами пегРНК это еще больше усложняет процесс проектирования.
Широкое внедрение инструментов редактирования генома требует инструментов для быстрого планирования экспериментов, и недавно стали доступны другие инструменты дизайна pegRNA. С помощью pegFinder (10) и PrimeDesign (11) пользователи могут проектировать пегРНК, вручную вставляя последовательность дикого типа и отредактированную последовательность либо как отдельные, либо как объединенные входные данные. Это требует создания предполагаемых последовательностей редактирования и фланкирования, что может увеличить риск ошибок, внесенных пользователем, и снизить пропускную способность. Кроме того, pegFinder не поддерживает пакетные проекты, а PrimeDesign поддерживает только NGG PAM SpCas9 дикого типа.Никейский домен. Кроме того, эти инструменты могут создавать олигонуклеотиды только для метода клонирования, описанного Анзалоне и его коллегами (7). Мы стремились разработать инструмент, который объединяет эталонные геномы с простой номенклатурой редактирования, чтобы обеспечить быстрый и эффективный дизайн pegRNAs для предполагаемых изменений. Кроме того, мы стремились создать инструмент с модульной структурой, позволяющий быстро настраивать новые основные варианты редактирования и правила оценки CRISPR.
Кроме того, мы стремились создать инструмент с модульной структурой, позволяющий быстро настраивать новые основные варианты редактирования и правила оценки CRISPR.
Мы разработали pegIT (http://pegit.giehmlab.dk), веб-инструмент и утилиту командной строки для автоматизированного проектирования pegRNAs для ряда определяемых пользователем правок. Пользователь может выбрать гены и транскрипты для организмов, имеющихся в базе данных, или предоставить заданную пользователем последовательность. Затем пользователь может выбирать между различными видами редактирования и создавать пегРНК для введения или исправления конкретного варианта. Кроме того, для облегчения планирования экспериментов pegIT разрабатывает праймеры для ПЦР-амплификации целевого локуса. Несмотря на то, что основное редактирование, как сообщается, вызывает более низкий уровень нецелевого редактирования по сравнению с Cas9., может происходить нецелевое редактирование (7), и такие потенциальные нецелевые сайты спейсеров pegRNA и nsgRNA также указаны в программном обеспечении (рис. 2).
2).
Рисунок 2.
Открыть в новой вкладкеСкачать слайд
Концепция pegIT и рабочая схема. Пользователь выбирает последовательность ДНК из базы данных или предоставляет пользовательскую последовательность и спецификацию желаемого редактирования для выполнения. В качестве альтернативы можно получить варианты из ClinVar, и пользователь может выбрать, ввести или исправить конкретный вариант. pegIT также поддерживает загрузку текстового файла с указанием последовательностей ДНК и желаемых изменений для разработки нескольких пегРНК. Затем pegIT идентифицирует спейсеры-кандидаты для дизайна пегРНК, конструирует сайт связывания праймеров и последовательности удлинения шаблона обратной транскриптазы, а также идентифицирует nsgRNAs для PE3. Алгоритм pegIT показан здесь с настройками по умолчанию; однако настройка параметров алгоритма pegIT доступна на вкладке дополнительных параметров. Следуя дизайну pegRNA, pegIT предоставляет простой в использовании графический обзор разработанных pegRNAs и nsgRNAs, а также последовательностей олигонуклеотидов для различных протоколов клонирования и синтетических последовательностей pegRNA.
МАТЕРИАЛЫ И МЕТОДЫ
Входные данные для pegIT
pegIT может создавать пегРНК для редактирования пользователем или для вариантов в ClinVar. Для разработки pegRNAs для вариантов ClinVar пользователь может искать нужный вариант и создавать pegRNAs для установки или восстановления варианта. Поиск ClinVar осуществляется с помощью пакета BioPython (12)
. Для разработки пользовательских правок pegIT содержит базу данных генов и транскриптов, для которой пользователь может легко создавать пегРНК для внесения или исправления интересующих правок. Путем выбора организма и поиска интересующего гена выявляются соответствующие транскрипты, и может быть выбрана желаемая последовательность. В качестве альтернативы пользователь может вставить пользовательскую последовательность ДНК или предоставить геномные координаты. Когда в качестве входных данных используются идентификаторы последовательностей или геномные координаты, соответствующая геномная область извлекается с использованием twoBitToFa (13). После выбора последовательности пользователю предоставляется форма для ввода желаемого типа редактирования. При использовании пользовательских последовательностей мы рекомендуем вставлять не менее 200 нуклеотидов по обе стороны от желаемого редактирования.
После выбора последовательности пользователю предоставляется форма для ввода желаемого типа редактирования. При использовании пользовательских последовательностей мы рекомендуем вставлять не менее 200 нуклеотидов по обе стороны от желаемого редактирования.
Указание желаемого редактирования
После выбора последовательности пользователю предоставляется форма для ввода желаемого типа редактирования. В настоящее время pegIT поддерживает вставки, делеции и замены, а изменения аминокислот могут быть указаны для транскриптов, кодирующих белок, без предварительного знания последовательностей кодонов. Наконец, каталог общих эпитопных меток может быть включен в определенные положения, если это необходимо. В зависимости от типа редактирования пользователь вводит желаемое изменение. Для облегчения ввода выбранная последовательность визуализируется. Здесь пользователь может искать последовательности ДНК и выбирать определенные позиции для редактирования. Кроме того, экзоны и трансляции представлены в виде последовательности, что позволяет легко использовать формат ввода аминокислот. Дополнительные возможности включают возможность создания pegRNAs для исправления выбранного редактирования и/или введения молчащих мутаций в PAM для соответствующей pegRNA для повышения эффективности редактирования (7).
Кроме того, экзоны и трансляции представлены в виде последовательности, что позволяет легко использовать формат ввода аминокислот. Дополнительные возможности включают возможность создания pegRNAs для исправления выбранного редактирования и/или введения молчащих мутаций в PAM для соответствующей pegRNA для повышения эффективности редактирования (7).
В качестве альтернативы, пользователи, желающие спроектировать pegRNAs для многих правок, могут загрузить текстовый файл с указанием желаемых правок. Наконец, пользователь выбирает желаемый домен нуклеазы PE, например, SpCas9 и клонирующий вектор. После выбора правок pegIT возвращает pegRNAs и nsgRNAs для введения или восстановления указанных вариантов.
Выбор спейсеров пегРНК и дизайн последовательностей удлинения
После указания редактирования pegIT идентифицирует потенциальные спейсеры, которые могут локализовать комплекс РЕ вблизи целевой последовательности. Когда в качестве входных данных используются идентификаторы последовательностей или геномные координаты, соответствующая геномная область извлекается с использованием twoBitToFa (13). В смысловой последовательности дикого типа ищут спейсеры, которые разрываются выше желаемого изменения, а в антисмысловой цепи ищут спейсеры, которые разрываются ниже по ходу транскрипции. pegIT возвращает до 5 спейсеров (по умолчанию) или указанное пользователем количество спейсеров для дальнейшего проектирования последовательностей расширения. Спейсеры pegRNA оцениваются в зависимости от того, нарушен ли PAM (i) в результате редактирования, (ii) по расстоянию до редактирования и в случае SpCas9., (iii) по целевому счету спейсера.
Когда в качестве входных данных используются идентификаторы последовательностей или геномные координаты, соответствующая геномная область извлекается с использованием twoBitToFa (13). В смысловой последовательности дикого типа ищут спейсеры, которые разрываются выше желаемого изменения, а в антисмысловой цепи ищут спейсеры, которые разрываются ниже по ходу транскрипции. pegIT возвращает до 5 спейсеров (по умолчанию) или указанное пользователем количество спейсеров для дальнейшего проектирования последовательностей расширения. Спейсеры pegRNA оцениваются в зависимости от того, нарушен ли PAM (i) в результате редактирования, (ii) по расстоянию до редактирования и в случае SpCas9., (iii) по целевому счету спейсера.
При настройках по умолчанию последовательности расширений разрабатываются на основе ранее установленных правил (7). Если указано пользователем, pegIT сначала пытается ввести молчащие мутации, чтобы нарушить последовательность PAM спейсера. pegIT начинается с PBS из 13 нуклеотидов, если содержание G/C отклоняется от 40 до 60%, рекурсивно добавляется дополнительный нуклеотид до максимальной длины 20 нуклеотидов. Если ни один из идентифицированных PBS не находится в этом диапазоне содержания G/C, pegIT выбирает последовательность PBS путем подсчета баллов на основе отклонения содержания G/C от 50% с длиной последовательности в качестве решающего фактора, отдавая предпочтение более коротким последовательностям. Матрица обратной транскриптазы (RTT) разработана так, чтобы иметь как минимум 10 гомологичных оснований после последнего отредактированного нуклеотида. Для более крупных правок длина измененных нуклеотидов увеличивается в два раза, максимум до 34 оснований. Основываясь на выборе домена нуклеазы PE и вектора клонирования, выбор и дизайн pegRNAs адаптируются к ним, например, для SpCas9., RTT, которые приводят к удлинению последовательностей, начинающихся с «C», отбрасываются, поскольку сообщалось, что они приводят к более низкой эффективности, предположительно, за счет нарушения структуры sgRNA (7). Для экспрессии с помощью промоторов U6 pegRNAs, содержащие мотив TTTT, отбрасываются, чтобы обеспечить эффективную продукцию pegRNA за счет снижения риска преждевременной терминации во время синтеза pegRNA.
Если ни один из идентифицированных PBS не находится в этом диапазоне содержания G/C, pegIT выбирает последовательность PBS путем подсчета баллов на основе отклонения содержания G/C от 50% с длиной последовательности в качестве решающего фактора, отдавая предпочтение более коротким последовательностям. Матрица обратной транскриптазы (RTT) разработана так, чтобы иметь как минимум 10 гомологичных оснований после последнего отредактированного нуклеотида. Для более крупных правок длина измененных нуклеотидов увеличивается в два раза, максимум до 34 оснований. Основываясь на выборе домена нуклеазы PE и вектора клонирования, выбор и дизайн pegRNAs адаптируются к ним, например, для SpCas9., RTT, которые приводят к удлинению последовательностей, начинающихся с «C», отбрасываются, поскольку сообщалось, что они приводят к более низкой эффективности, предположительно, за счет нарушения структуры sgRNA (7). Для экспрессии с помощью промоторов U6 pegRNAs, содержащие мотив TTTT, отбрасываются, чтобы обеспечить эффективную продукцию pegRNA за счет снижения риска преждевременной терминации во время синтеза pegRNA.
Отбор nsgRNAs
Удаление противоположной цепи может привести к более высокой эффективности редактирования. Для идентификации таких nsgRNAs идентифицируют все спейсеры в отредактированной последовательности в пределах 100 нуклеотидов от редактирования. Использование nsgRNAs, специфичных для редактируемой последовательности, сводит к минимуму одновременные разрывы и введение двухцепочечных разрывов (7), стратегия, названная PE3b. pegIT отдает приоритет nsgRNA, которые не совпадают в последовательности дикого типа. Для SpCas9nsgRNAs с несовпадениями оценивают с помощью CFD (14) по сравнению с последовательностью дикого типа. Затем наилучший кандидат выбирается на основе наименьшего балла за последовательность дикого типа; неспецифические sgRNA оцениваются на основе расстояния> 50 от сайта разрыва pegRNA, за которым следует их оценка на мишени (14).
Нецелевой поиск
Спейсерные последовательности pegRNA и nsgRNA сопоставляются с эталонным геномом выбранного организма с помощью Bowtie (15) с не более чем тремя несовпадениями, используя команду «bowtie -v 3 -a –best -y» .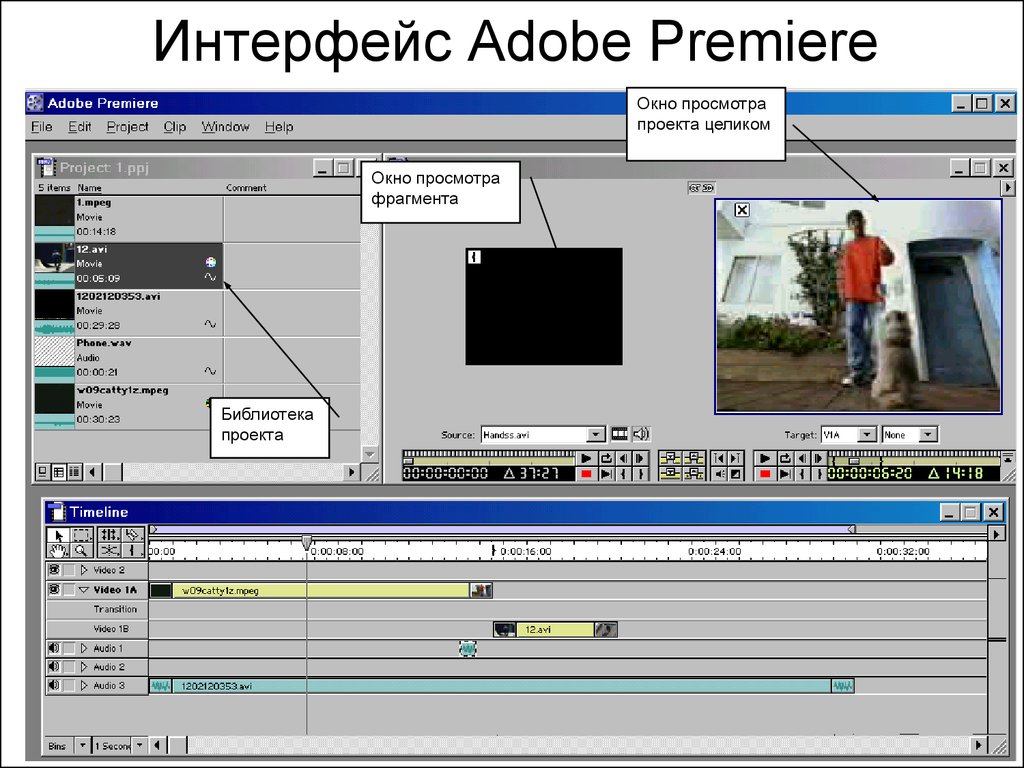 Последовательности ДНК, соответствующие совпадающим совпадениям, включая 3 нуклеотида ниже по течению, извлекаются с использованием twoBitToFa (13) и фильтруются для отбрасывания совпадений, которые не являются соседними с NAG или NGG PAM.
Последовательности ДНК, соответствующие совпадающим совпадениям, включая 3 нуклеотида ниже по течению, извлекаются с использованием twoBitToFa (13) и фильтруются для отбрасывания совпадений, которые не являются соседними с NAG или NGG PAM.
Дизайн грунтовки
Праймеры разработаны с использованием Primer3 (16). С параметрами по умолчанию, размер праймера 18–25 нт, оптимальный 22 нт, размер продукта 150–300 п.н., праймер T m 57–63°C, оптимум 60°C. Для проверки специфичности праймеров 3 пары праймеров, прямое-прямое, прямое-обратное и обратное-обратное для каждой пары праймеров сопоставляются с использованием Bowtie в парном режиме, с до 3 несоответствий и размером от одной трети минимального размера продукта до 3. раза больше размера продукта. С настройками по умолчанию команда «bowtie -v 3 -k 50 –best -I 50 -X 900 -y’ используется.
Страница результатов
На странице результатов пользователь может проверить разработанные пегРНК. Для заданий с несколькими правками отображается список обработанных правок. Дизайн pegRNA с самым высоким рейтингом можно просмотреть непосредственно в этой таблице, а альтернативные дизайны pegRNA можно просмотреть, нажав кнопку Details. Отдельные задания редактирования отправляются непосредственно на страницу сведений. Когда pegRNAs будут разработаны, можно загрузить файл Excel, содержащий результаты. На странице сведений спейсеры pegRNA и их расширения визуализируются для быстрой проверки. Дополнительная информация представлена в прилагаемой таблице, включая расстояние между Cas9никирование и желаемое редактирование, а также нарушена ли pegRNA PAM редактированием или молчащими мутациями (необязательно). Олиго для клонирования предложенной пегРНК можно получить прямо из этой таблицы. Поскольку первичное редактирование является новой технологией, оптимальные правила дизайна для pegRNAs еще не полностью установлены, и может потребоваться оптимизация для конкретного локуса. Чтобы упростить тестирование различных расширений пегРНК и нсгРНК, pegIT предоставляет список альтернативных кандидатов в нсгРНК и комбинации последовательностей PBS и RTT, а также олигонуклеотиды для клонирования.
Для заданий с несколькими правками отображается список обработанных правок. Дизайн pegRNA с самым высоким рейтингом можно просмотреть непосредственно в этой таблице, а альтернативные дизайны pegRNA можно просмотреть, нажав кнопку Details. Отдельные задания редактирования отправляются непосредственно на страницу сведений. Когда pegRNAs будут разработаны, можно загрузить файл Excel, содержащий результаты. На странице сведений спейсеры pegRNA и их расширения визуализируются для быстрой проверки. Дополнительная информация представлена в прилагаемой таблице, включая расстояние между Cas9никирование и желаемое редактирование, а также нарушена ли pegRNA PAM редактированием или молчащими мутациями (необязательно). Олиго для клонирования предложенной пегРНК можно получить прямо из этой таблицы. Поскольку первичное редактирование является новой технологией, оптимальные правила дизайна для pegRNAs еще не полностью установлены, и может потребоваться оптимизация для конкретного локуса. Чтобы упростить тестирование различных расширений пегРНК и нсгРНК, pegIT предоставляет список альтернативных кандидатов в нсгРНК и комбинации последовательностей PBS и RTT, а также олигонуклеотиды для клонирования. Их можно найти в представлении для соответствующей pegRNA или в загружаемом файле Excel. Файл Excel содержит сводную таблицу, в которой перечислены проекты с лучшим рейтингом для каждого редактирования. Альтернативные спейсеры и расширения пегРНК можно найти на отдельном листе для каждого редактирования.
Их можно найти в представлении для соответствующей pegRNA или в загружаемом файле Excel. Файл Excel содержит сводную таблицу, в которой перечислены проекты с лучшим рейтингом для каждого редактирования. Альтернативные спейсеры и расширения пегРНК можно найти на отдельном листе для каждого редактирования.
Для оптимизации дизайна направляющей РНК pegIT сообщает о целевых показателях, ранее разработанных для экспериментов по нокауту SpCas9 как для пегРНК, так и для нсгРНК (14). Для отбора нсгРНК PE3b pegIT также сообщает о нецелевом показателе для аллеля дикого типа для SpCas9 (14). Кроме того, количество и положение предполагаемых сайтов связывания для pegRNAs и nsgRNAs в выбранном организме прогнозируются с точностью до трех несоответствий. Подробный обзор предсказанных сайтов связывания можно найти в соответствующих подробных представлениях.
Версия для командной строки
В дополнение к веб-серверу исходный код можно загрузить и запустить локально как в виде командной строки, так и в виде веб-приложения, которое подходит для более крупных запросов, конфиденциальных данных или внутренних линий сотовой связи. . Все функции, доступные на веб-сервере, также доступны в версии для командной строки.
. Все функции, доступные на веб-сервере, также доступны в версии для командной строки.
ОБСУЖДЕНИЕ
После адаптации CRISPR/Cas9 для редактирования генома (1,17–19) наше понимание факторов, определяющих дизайн sgRNAs для эффективного редактирования генома, быстро изменилось. Точно так же базовые редакторы (4) претерпели быстрое развитие с момента их первоначального создания. Чтобы приспособиться к ожидаемому развитию основной технологии редактирования, pegIT был разработан по модульному принципу. Нуклеазы, клонирующие плазмиды, правила проектирования и редактирования разрабатываются независимо друг от друга в виде «плагинов», что позволяет легко настраивать оптимизированные правила подсчета пегРНК и новые варианты основного редактирования. Это позволяет поддерживать и развивать pegIT. Пользователи могут запросить добавление правок, доменов нуклеаз, организмов и правил клонирования. В будущих обновлениях, помимо добавления новых организмов, новых вариантов PE, а также правил дизайна и клонирования, также могут быть предусмотрены правки для изменений фенотипа, например. путем включения правок для настройки экспрессии генов на основе моделей прогнозирования скорости трансляции (20).
путем включения правок для настройки экспрессии генов на основе моделей прогнозирования скорости трансляции (20).
В целом, pegIT представляет собой новую платформу для быстрого и удобного дизайна pegRNAs и nsgRNAs для реализации первичного редактирования в основных экспериментах и терапии.
ДОСТУПНОСТЬ ДАННЫХ
Исходный код программного обеспечения pegIT доступен по адресу https://github.com/dkmva/pegit и https://github.com/dkmva/pegit-front.
ФИНАНСИРОВАНИЕ
Фонд Лундбек [R230-2016-2986, R324-2019-1832]. Финансирование платы за открытый доступ: Орхусский университет.
Заявление о конфликте интересов . Ни один не заявил.
Примечания
Текущий адрес: Мадс Вальдемар Андерсон, Global Biopharm Research Rare Disease Research, Novo Nordisk, Måløv, 2760, Дания.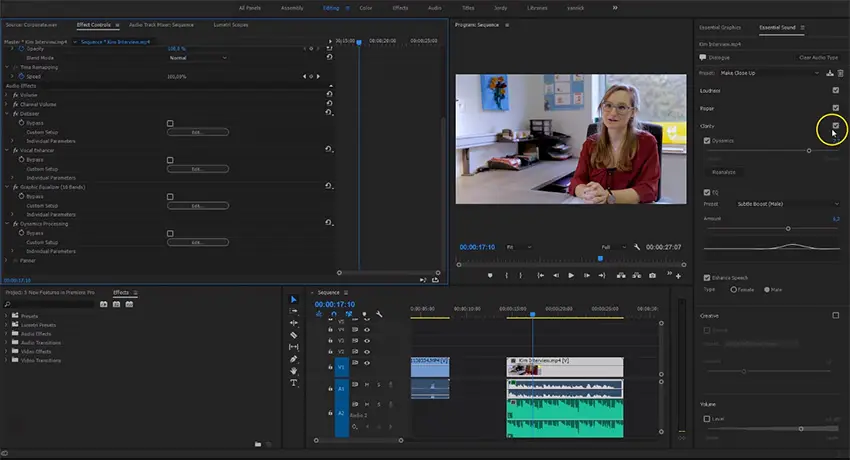
REFERENCES
1.
Jinek
M.
,
Chylinski
K.
,
Fonfara
I.
,
Hauer
M.
,
Doudna
Дж.А.
,
Шарпантье
E.
Программируемая ДНК-эндонуклеаза, управляемая двойной РНК, в адаптивном бактериальном иммунитете
.
Наука
.
2012
;
337
:
816
–
821
.
2.
Ран
Ф. А.
А.
,
Хсу
П.Д.
,
Райт
Дж.
,
Агарвала
В.
,
Скотт
Д.А.
,
Zhang
F.
Геномная инженерия с использованием системы CRISPR-Cas9
.
Нац. протокол
2013
;
8
:
2281
–
2308
.
3.
Шалем
О.
,
Санджана
Н.В.
,
Хартениан
E.
,
Ши
X.
,
Скотт
Д. А.
А.
,
Миккельсен
Т.С.
,
Хекл
Д.
,
Эберт
Б.Л.
,
Корень
Д.Е.
,
Дёнч
Д.Г.
и др. .
Скрининг нокаута CRISPR-Cas9 в масштабе генома в клетках человека
.
Наука
.
2014
;
343
:
84
–
87
.
4.
Рис
Х.А.
,
Лю
Д.Р.
Базовое редактирование: прецизионная химия генома и транскриптома живых клеток
.
Нац. Преподобный Жене.
2018
;
19
:
770
–
788
.
5.
Курт
И.К.
,
Zhou
R.
,
Iyer
S.
,
Garcia
S.P.
,
Miller
B.R.
,
Лангнер
Л.М.
,
Грюневальд
Дж.
,
Юнг
2.К.
Базовые редакторы CRISPR C-to-G для индуцирования целевых трансверсий ДНК в клетках человека
.
Нац. Биотехнолог.
2021
;
39
:
41
–
46
.
6.
Zhao
D.
,
Li
J.
,
Li
S.
,
Xin
X.
,
Hu
M
,
Цена
М.А.
,
Россер
С.Дж.
,
Bi
C.
,
Zhang
X.
Редакторы оснований гликозилазы позволяют изменять основания C-to-A и C-to-G
.
Нац. Биотехнолог.
2021
;
39
:
35
–
40
.
7.
Анзалон
А.В.
,
Рэндольф
П.Б.
,
Дэвис
J.R.
,
Соуза
А.А.
,
Коблан
Л.В.
,
Levy
J.M.
,
Chen
P.J.
,
Wilson
C.
,
1.1.
..
,
1.1.
,
1.1.
,
1. 1.
1.
,
1.1.
.
,
Рагурам
А.
и др. .
Редактирование генома методом «поиск и замена» без двухцепочечных разрывов или донорской ДНК
.
Природа
.
2019
;
576
:
149
–
157
.
8.
Lin
Q.
,
Zong
Y.
,
Xue
C.
,
Wang
S.
,
Jin
S
,
Чжу
З.
,
Ван
Ю.
,
Анзалон
А.В.
,
Рагурам
А.
,
Доман
Дж.Л.
и др. .
Основное редактирование генома риса и пшеницы
.
Нац. Биотехнолог.
2020
;
38
:
582
–
585
.
9.
Лю
Ю.
,
Li
X.
,
He
S.
,
Huang
S.
,
Li
C.
,
Chen
Y.
,
LIU
Z.
,
HUANG
X.
,
WANG
X.
Эффективное поколение моделей мыши с основным эдитингом
.
Сотовый Дисков.
2020
;
6
:
27
.
10.
Чоу
Р.Д.
,
Чен
Дж.С.
,
Shen
J.
,
Chen
S.
Веб-инструмент для разработки руководств по редактированию РНК
.
Нац. Биомед. англ.
2020
;
5
:
190
–
194
.
11.
Хсу
Д.Ю.
,
Grünewald
J.
,
Szalay
R.
,
Shih
J.
,
Anzalone
A.V.
,
Лам
К.К.
,
Шен
МВт
,
Петри
К.
,
Лю
Д.Р.,
Юнг
Дж.К.
и др. .
Программное обеспечение PrimeDesign для быстрого и упрощенного проектирования основных направляющих для редактирования РНК
.
Нац. коммун.
2021
;
12
:
1034
.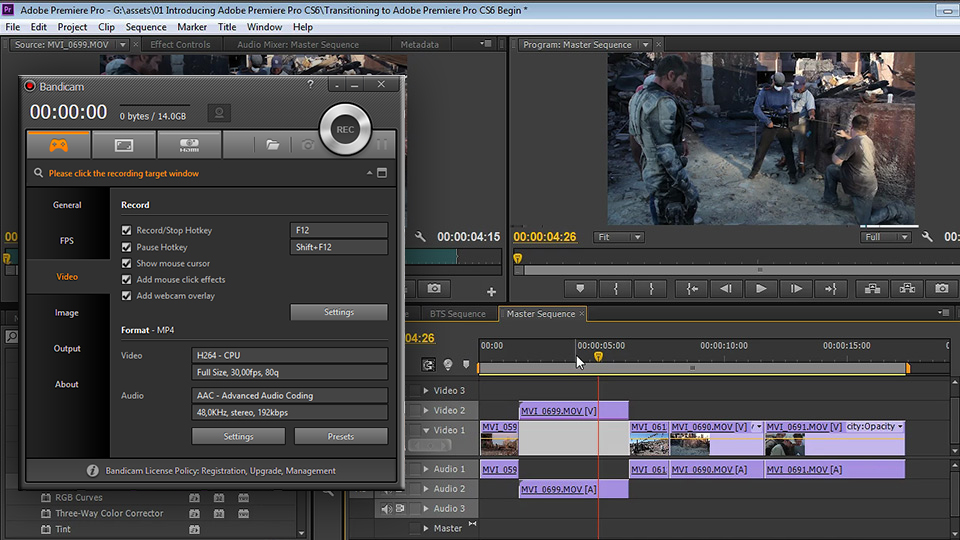
12.
Петух
PJa
,
Антао
T.
,
Чан
.JT.9003,
Чепмен
B.a
,
Cox
C.J.
,
Dalke
A.
,
Friedberg
I.
,
Hamelryck
T.
,
Kauff
F.
,
Wilczynski
B.
и др. .
Biopython: бесплатные инструменты Python для вычислительной молекулярной биологии и биоинформатики
.
Биоинформатика
.
2009
;
25
:
1422
–
1423
.
13.
Карольчик
Д.
,
Хинрикс
А.С.
,
Фьюри
Т.С.
,
Роскин
К.М.
,
Сугнет
К.В.0032
W.J.
Инструмент поиска данных обозревателя таблиц UCSC
.
Рез. нуклеиновых кислот.
2004
;
32
:
493
–
496
.
14.
Дёнч
Дж.Г.
,
Fusi
N.
,
Sullender
M.
,
Hegde
M.
,
Vaimberg
EW
,
Донован
К.Ф.
,
Smith
I.
,
Tothova
Z.
,
Wilen
C.
,
Orchard
R.
et al. .
Оптимизированная конструкция sgRNA для максимальной активности и минимизации побочных эффектов CRISPR-Cas9
.
Нац. Биотехнолог.
2016
;
34
:
184
–
191
.
.
Сверхбыстрое и эффективное с точки зрения памяти сопоставление коротких последовательностей ДНК с геномом человека
.
Геном Биол.
2009
;
10
:
R25
.
16.
Untergasser
A.
,
Cutcutache
I.
,
Koressaar
T.
,
Ye
J.
,
Faircloth
B.C.
,
Remm
M.
,
Rozen
S.G.
Primer3 – новые возможности и интерфейсы
.
Рез. нуклеиновых кислот.
2012
;
40
:
e115
.
17.
Cong
L.
,
Ran
F.A.
,
Cox
D.
,
Lin
S.
,
Barretto
R.
,
Хабиб
Н.
,
Хсу
П.Д.
,
Ву
Х.
,
Jiang
W.
,
Marraffini
L.A.
и др. .
Мультиплексная инженерия генома с использованием систем CRISPR/Cas
.
Science (Нью-Йорк, штат Нью-Йорк)
.
2013
;
339
:
819
–
823
.
18.
Мали
П.
,
Ян
Л.
,
Эсвельт
К.М.
,
Aach
J.
,
Guell
M.
,
DiCarlo
J.E.
,
Norville
J.E.
,
Church
G.M.
Инженерия генома человека с помощью РНК с помощью Cas9
.
Наука
.
2013
;
339
:
823
–
826
.
19.
Jinek
M.
,
East
A.
,
Cheng
A.
,
,
.
,
Дудна
J.
РНК-программированное редактирование генома в клетках человека
.
eLife
.
2013
;
2
:
e00471
.
20.
Образец
П.Дж.
,
Пресняк
В.
,
МакФадьен
И.Дж.
,
Моррис
Д. Р.
Р.
,
Seelig
G.
Дизайн 5′-UTR человека и предсказание эффекта варианта на основе анализа массивно-параллельной трансляции
.
Нац. Биотехнолог.
2019
;
37
:
803
–
809
.
© Автор(ы), 2021 г. Опубликовано Oxford University Press от имени Nucleic Acids Research.
Это статья в открытом доступе, распространяемая в соответствии с условиями некоммерческой лицензии Creative Commons Attribution (http://creativecommons.org/licenses/by-nc/4.0/), которая разрешает повторное использование, распространение, и воспроизведение на любом носителе при условии надлежащего цитирования оригинальной работы. По вопросам коммерческого повторного использования обращайтесь по адресу journals.permissions@oup. com
com
© Автор(ы), 2021 г. Опубликовано Oxford University Press от имени Nucleic Acids Research.
Раздел проблем:
Проблема веб-сервера
Скачать все слайды
Реклама
Цитаты
Альтметрика
Дополнительная информация о метриках
Оповещения по электронной почте
Оповещение об активности статьи
Предварительные уведомления о статьях
Оповещение о новой проблеме
Оповещение о теме
Получайте эксклюзивные предложения и обновления от Oxford Academic
Ссылки на статьи по номеру
Последний
Самые читаемые
Самые цитируемые
Мониторинг взаимодействия лекарство-мишень посредством амплификации, опосредованной вовлечением мишени, на массивах и на месте
Исследование убиквитинирования белка в живых клетках
Белки TRIPLE PHD FINGERS необходимы для опосредованного комплексом SWI/SNF позиционирования +1 нуклеосомы и определения места начала транскрипции у арабидопсиса 9. 0032
0032
EmAtlas: исчерпывающий атлас для изучения пространственно-временной активации в эмбриогенезе млекопитающих
microbioTA: атлас микробиома в тканях множественных заболеваний Homo sapiens и Mus musculus
Реклама
Prime Editing: редактирование генома при редких генетических заболеваниях без двухцепочечных разрывов или донорской ДНК
Статья, опубликованная в Nature (Anzalone et al., 2019) сообщает о разработке экспериментального подхода к редактированию генома, который опосредует все возможные преобразования оснований, «вставки» и комбинации в геноме человека без необходимости использования двухцепочечных разрывов (DSB) или шаблонов донорской ДНК (дДНК). Первичное редактирование, новый метод редактирования генома, использует более длинную, чем обычно, одиночную направляющую РНК (гРНК), известную как первичное редактирование гРНК (пегРНК), и слитый белок, состоящий из никазы Cas9 H840A, слитой со сконструированной обратной транскриптазой (RT). ) фермент. Первичное редактирование, описываемое как технология редактирования оснований «поиск и замена», предоставляет желаемую генетическую конструкцию в расширении гРНК, которая затем преобразуется в ДНК с помощью фермента RT. Новый подход устраняет необходимость в совместной доставке корректирующей ДНК-матрицы, выполняет все возможные замены нуклеотидов (в том числе замены для значительной части генетических нарушений), устраняет сдвиги рамки считывания, вызванные вставками, и вносит меньше нецелевых правок по сравнению с обычным CRISPR. -Кас устройства. Прайм-редактирование — это захватывающее новое дополнение к существующим системам редактирования CRISPR, которое во многих случаях может быть даже улучшением. Однако основное редактирование ставит новые задачи. Преодоление этих препятствий и применение простого редактирования in vivo , приведет к новым методам редактирования генома для лечения редких генетических заболеваний.
) фермент. Первичное редактирование, описываемое как технология редактирования оснований «поиск и замена», предоставляет желаемую генетическую конструкцию в расширении гРНК, которая затем преобразуется в ДНК с помощью фермента RT. Новый подход устраняет необходимость в совместной доставке корректирующей ДНК-матрицы, выполняет все возможные замены нуклеотидов (в том числе замены для значительной части генетических нарушений), устраняет сдвиги рамки считывания, вызванные вставками, и вносит меньше нецелевых правок по сравнению с обычным CRISPR. -Кас устройства. Прайм-редактирование — это захватывающее новое дополнение к существующим системам редактирования CRISPR, которое во многих случаях может быть даже улучшением. Однако основное редактирование ставит новые задачи. Преодоление этих препятствий и применение простого редактирования in vivo , приведет к новым методам редактирования генома для лечения редких генетических заболеваний.
Введение: технологии программируемого редактирования генома
Прецизионное редактирование генома — универсальный и мощный инструмент генной терапии.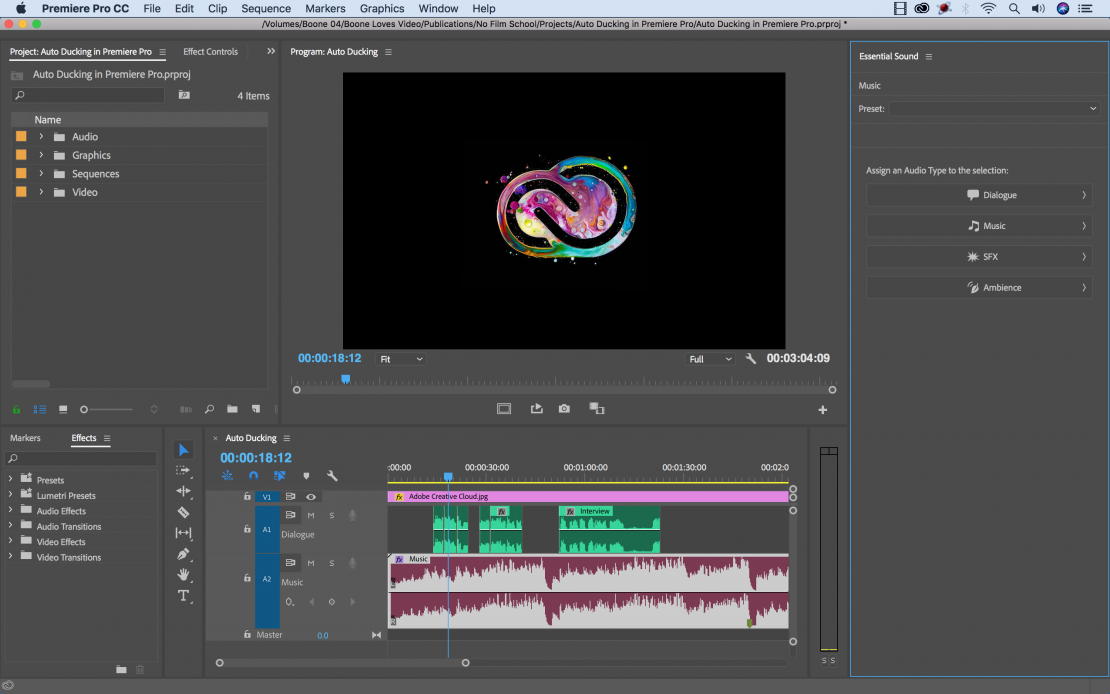 С момента разработки систем CRISPR/Cas для редактирования генома (рис. 1А) эта область постоянно совершенствовалась (Charpentier and Doudna, 2013; Doudna and Charpentier, 2014; Hsu et al., 2014; Sternberg and Doudna, 2015; Комор и др., 2017). Новая биотехнология включает в себя формирование сайт-специфического DSB, за которым следуют два основных типа механизмов репарации: негомологичное соединение концов (NHEJ; Davis and Chen, 2013) и репарация, направленная на гомологию (HDR; Li and Heyer, 2008). . Активированный тип механизма молекулярной репарации зависит от генома, клеточной гетерогенности и цикла клеточного деления (Hsu et al., 2014; Komor et al., 2017).
С момента разработки систем CRISPR/Cas для редактирования генома (рис. 1А) эта область постоянно совершенствовалась (Charpentier and Doudna, 2013; Doudna and Charpentier, 2014; Hsu et al., 2014; Sternberg and Doudna, 2015; Комор и др., 2017). Новая биотехнология включает в себя формирование сайт-специфического DSB, за которым следуют два основных типа механизмов репарации: негомологичное соединение концов (NHEJ; Davis and Chen, 2013) и репарация, направленная на гомологию (HDR; Li and Heyer, 2008). . Активированный тип механизма молекулярной репарации зависит от генома, клеточной гетерогенности и цикла клеточного деления (Hsu et al., 2014; Komor et al., 2017).
Рисунок 1 . Структура и функции основного редактора. Обычный редактор ДНК CRISPR (A) . CRISPR основан на способности гРНК CRISPR нацеливать эндонуклеазу Cas9 на точные участки генома, где Cas9 вводит DSB. Редакторы оснований не переваривают двойную цепь, а вместо этого химически изменяют одиночные основания с помощью ферментов деаминаз, таких как TadA ( B , редактор оснований ДНК) и ADAR ( C , редактор оснований РНК). Главный редактор (Д) включает более длинную, чем обычно, гРНК, известную как пегРНК, и слитый белок, состоящий из никазы Cas9 H840A, слитой с модифицированным ферментом RT. Элемент Cas9 главного редактора расщепляет геномную ДНК, а элемент RT полимеризует ДНК на цепи с разрывом на основе последовательности pegRNA (E) . Адаптировано из Anzalone et al. (2019) и Мацукас (2018a).
Главный редактор (Д) включает более длинную, чем обычно, гРНК, известную как пегРНК, и слитый белок, состоящий из никазы Cas9 H840A, слитой с модифицированным ферментом RT. Элемент Cas9 главного редактора расщепляет геномную ДНК, а элемент RT полимеризует ДНК на цепи с разрывом на основе последовательности pegRNA (E) . Адаптировано из Anzalone et al. (2019) и Мацукас (2018a).
Таким образом, индукция точной мутации, вызванной инструментом редактирования генома, в значительной степени зависит от HDR, происходящего в DSB локус через матрицу дДНК, содержащую желаемую конструкцию (Yang et al., 2014; Song and Stieger, 2017; Bollen et al., 2018). Хотя системы CRISPR/Cas могут эффективно создавать DSB в определенной геномной последовательности (Cong et al., 2013; Jinek et al., 2013; Savic et al., 2018), HDR в клетках млекопитающих неэффективен или непригоден из-за низкой врожденная скорость HDR и препятствия при доставке дДНК на месте (Cong et al. , 2013; Mali et al., 2013). Коэффициенты коррекции обычных инструментов редактирования генома составляют 0,1–5%, и, как правило, они вносят множество случайных вставок в целевую геномную последовательность в результате клеточного ответа на DSB (Cox et al., 2015; Hilton and Gersbach). , 2015). Недавно были разработаны инструменты редактирования оснований с помощью CRISPR/Cas, позволяющие обойти эти ограничения (рис. 1B,C; Komor et al., 2016; Nishida et al., 2016; Kim et al., 2017; Gehrke et al., 2018). ; Li et al., 2018; Sharon et al., 2018; Wang et al., 2018). Было опубликовано несколько отличных обзорных статей о различных типах настраиваемых базовых редакторов (Rees and Liu, 2018; Molla and Yang, 2019).; Ян и др., 2019). Следовательно, эти инструменты редактирования базы не будут подробно описаны здесь.
, 2013; Mali et al., 2013). Коэффициенты коррекции обычных инструментов редактирования генома составляют 0,1–5%, и, как правило, они вносят множество случайных вставок в целевую геномную последовательность в результате клеточного ответа на DSB (Cox et al., 2015; Hilton and Gersbach). , 2015). Недавно были разработаны инструменты редактирования оснований с помощью CRISPR/Cas, позволяющие обойти эти ограничения (рис. 1B,C; Komor et al., 2016; Nishida et al., 2016; Kim et al., 2017; Gehrke et al., 2018). ; Li et al., 2018; Sharon et al., 2018; Wang et al., 2018). Было опубликовано несколько отличных обзорных статей о различных типах настраиваемых базовых редакторов (Rees and Liu, 2018; Molla and Yang, 2019).; Ян и др., 2019). Следовательно, эти инструменты редактирования базы не будут подробно описаны здесь.
Вкратце, редакторы базы генерируют мутации при разрешении одной базы. Все четыре переходные мутации, C → T, G → A, A → G и T → C, могут быть введены в геном с помощью уже описанных редакторов оснований CRISPR/Cas. Редакторы оснований цитозина (Komor et al., 2016; Nishida et al., 2016; Kim et al., 2017; Gehrke et al., 2018; Li et al., 2018; Matsoukas, 2018a; Wang et al., 2018). ) может установить мутацию C-G в TA, в то время как редакторы оснований аденина (Gaudelli et al., 2017; Kim et al., 2019) может преобразовать базовую диаду AT в диаду G-C. В РНК преобразование аденина в инозин также возможно с помощью редактора оснований РНК (рис. 1C; Cox et al., 2017; Matsoukas, 2018b). Поскольку многие генетические заболевания и расстройства возникают в результате нуклеотидных замен и добавлений или делеций нуклеотидов, редактирование оснований имеет важное значение в изучении патогенеза человека.
Редакторы оснований цитозина (Komor et al., 2016; Nishida et al., 2016; Kim et al., 2017; Gehrke et al., 2018; Li et al., 2018; Matsoukas, 2018a; Wang et al., 2018). ) может установить мутацию C-G в TA, в то время как редакторы оснований аденина (Gaudelli et al., 2017; Kim et al., 2019) может преобразовать базовую диаду AT в диаду G-C. В РНК преобразование аденина в инозин также возможно с помощью редактора оснований РНК (рис. 1C; Cox et al., 2017; Matsoukas, 2018b). Поскольку многие генетические заболевания и расстройства возникают в результате нуклеотидных замен и добавлений или делеций нуклеотидов, редактирование оснований имеет важное значение в изучении патогенеза человека.
Однако редакторы базы не могут установить все мутации трансверсии. Например, базовые редакторы, о которых сообщается в настоящее время, не могут вводить восемь мутаций трансверсии (C → A, C → G, G → C, G → T, A → C, A → T, T → A и T → G), такие как мутация T·A-to-A·T необходима для точной коррекции наиболее распространенной этиологии серповидно-клеточной анемии (SCD). SCD, гомозиготная мутация (от A → T) в шестом кодоне (E6V) гена b-глобина человека (HBB), превращает глутамат в валин, который синтезирует дефектные белки b-глобина и приводит к аномальным эритроцитам. Вакульскас и др., 2018). Предполагается, что HDR-опосредованная коррекция гена HBB в аутологичных гемопоэтических стволовых клетках и клетках-предшественниках может быть безопасным и эффективным подходом к генной терапии ВСС.
SCD, гомозиготная мутация (от A → T) в шестом кодоне (E6V) гена b-глобина человека (HBB), превращает глутамат в валин, который синтезирует дефектные белки b-глобина и приводит к аномальным эритроцитам. Вакульскас и др., 2018). Предполагается, что HDR-опосредованная коррекция гена HBB в аутологичных гемопоэтических стволовых клетках и клетках-предшественниках может быть безопасным и эффективным подходом к генной терапии ВСС.
Кроме того, не сообщалось ни о каком экспериментальном подходе без DSB, обеспечивающем целевые делеции, такие как удаление дупликации из 4 оснований, вызывающей болезнь Тея-Сакса (HEXA 1278+TATC; McGinniss et al., 2002), или целевые вставки, такие как вставка из 3 оснований, необходимая для точной коррекции наиболее распространенной этиологии муковисцидоза (см. Lukacs and Verkman, 2012). Что касается МВ, делеция остатка фенилаланина в положении ΔF508 присутствует в одном или обоих аллелях примерно у 90% пациентов с МВ. Интересно, что эти проблемы стимулируют разработку новых и современных технологий точного редактирования генома.
В этой статье описывается первичное редактирование, новый инструмент редактирования генома, который был разработан для расширения масштабов и возможностей существующих методов лечения редких генетических заболеваний на основе CRISPR/Cas. В статье также обсуждаются новые проблемы, связанные с новой биотехнологией, и предлагаются возможные направления будущих исследований.
Prime Editors: Расширение набора инструментов для редактирования генома
Целевые трансверсии, вставки и делеции проблематично индуцировать или восстанавливать эффективно и без избыточных побочных продуктов в большинстве клеточных типов, даже несмотря на то, что они в совокупности составляют большинство известных патогенных аллелей. Интересно, что статья, недавно опубликованная в журнале Nature (Anzalone et al., 2019) сообщает об инструменте, разработанном для устранения ограничений базового редактирования, описанных выше. Основное редактирование, новейший инструмент редактирования базы, использует тот же механизм, что и обычные системы CRISPR/Cas, опосредуя все 12 возможных преобразований базы в базу и их комбинации, но без присвоения DSB в целевой последовательности или использования шаблона дДНК. Основное редактирование включает более длинную, чем обычно, гРНК, известную как пегРНК, и гибридный белок, состоящий из никазы Cas9 H840A, слитой со сконструированным ферментом RT (рис. 1D).
Основное редактирование включает более длинную, чем обычно, гРНК, известную как пегРНК, и гибридный белок, состоящий из никазы Cas9 H840A, слитой со сконструированным ферментом RT (рис. 1D).
Эта статья (Anzalone et al., 2019) следует за другими важными публикациями (Gaudelli et al., 2017; Kim et al., 2017) той же исследовательской группы, в которых описывались первые молекулярные устройства для редактирования оснований. В последней статье Anzalone et al. (2019) проверили, может ли гРНК быть расширена за счет включения дополнительных нуклеотидов. Некоторые из этих дополнительных нуклеотидов будут служить матрицей для синтеза новой последовательности ДНК, в то время как другие будут связываться с цепью ДНК, противоположной ожидаемому сайту связывания гРНК, чтобы использовать эту геномную локус в качестве праймера для инициации ОТ. С созданием удлиненной пегРНК Anzalone et al. (2019) удалось не только направить варианты фермента Cas9 в соответствующий локус , но также установить желаемое редактирование и активировать фермент RT (рис.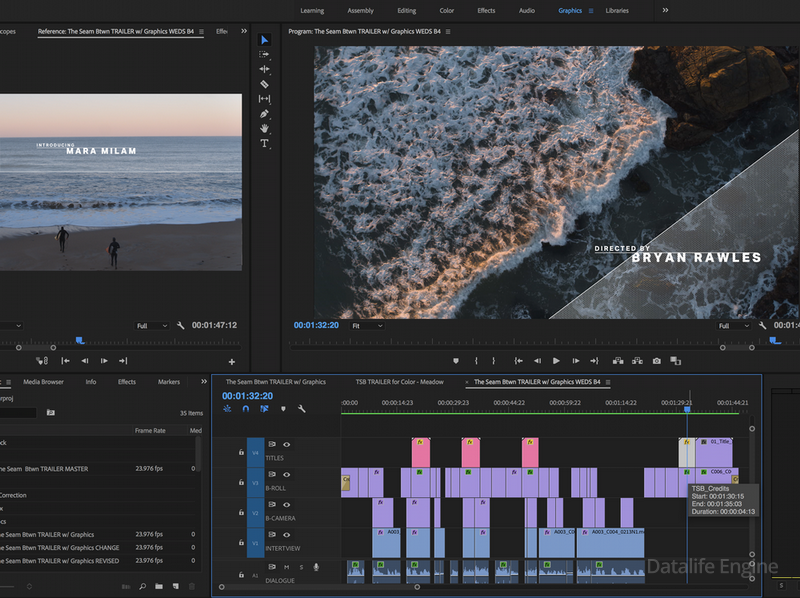 1E).
1E).
Этот новый экспериментальный подход был дополнен созданием трех дополнительных устройств. Первое молекулярное устройство (PE1) было создано путем слияния никазы Cas9 H840A и фермента RT вируса мышиного лейкоза Малони дикого типа (WT). В этой конструкции Cas9Никазный домен H840A основного редактирующего слитого белка (PEFP) надрезает только одну из полинуклеотидных цепей для последующего восстановления, тогда как домен RT генерирует комплементарную ДНК (кДНК) путем копирования пегРНК (несущей желаемую конструкцию) для восстановления сегмента надрезанного фрагмента. Нить ДНК. В этом устройстве Anzalone et al. (2019) также увеличили длину сайта пегРНК, который связывает праймерный участок ДНК. Применение устройства PE1 привело к незначительным, но все же обнаруживаемым изменениям генома.
В устройстве PE2 Anzalone et al. (2019) улучшили термостабильность, процессивность и сродство ДНК-РНК-субстрата RT-компонента PEFP путем введения пяти специфических мутаций. Этот пентамутантный фермент RT встраивается в PE1, создавая устройство PE2. Устройство PE2 привело к 5,1-кратному повышению эффективности мутации основных точек редактирования и более методично вносило целевые вставки и делеции, чем PE1.
Устройство PE2 привело к 5,1-кратному повышению эффективности мутации основных точек редактирования и более методично вносило целевые вставки и делеции, чем PE1.
В третьем молекулярном устройстве (PE3) Anzalone et al. (2019) ввели вторую гРНК в дополнение к пегРНК. Дополнительная гРНК представляла собой стандартную гРНК, направляющую элемент никазы Cas9 H840A PEFP на разрыв геномной ДНК в соседнем сайте, но на цепи, противоположной исходному разрыву. Они применили этот подход из-за опасений, что эффективное редактирование одной цепи, наблюдаемое с устройством PE2, может быть подавлено из-за несоответствия между сконструированными и не сконструированными цепями ДНК. Установив разрыв на неинженерной цепи ДНК, Anzalone et al. (2019) предположил, что механизм репарации ДНК может заменить исходный локус желаемым сегментом. Интересно, что устройство PE3 было более эффективным, когда дополнительная гРНК конструировалась так, чтобы соответствовать вновь отредактированной последовательности, введенной пегРНК. Устройство PE3 с новой характеристикой гРНК было обозначено как PE3b. Группа также наблюдала повышение эффективности, когда они разработали pegRNA для мутации исходного мотива, прилегающего к протоспейсеру (PAM).
Устройство PE3 с новой характеристикой гРНК было обозначено как PE3b. Группа также наблюдала повышение эффективности, когда они разработали pegRNA для мутации исходного мотива, прилегающего к протоспейсеру (PAM).
Редакторы базы ДНК неизменно доказали свою эффективность для установки точных точечных мутаций в геном самых разных модельных систем [рассмотрено в (Sharon et al., 2018; Molla and Yang, 2019).)]. В статье, опубликованной Anzalone et al. (2019), устройства PE2, PE3 и PE3b сравнивали с редакторами оснований цитидина/аденина и с системой HDR. Интересно, что было показано, что эти PE-устройства функционируют дополнительным образом к системам базового редактора, в зависимости от желаемого локуса . Кроме того, основное редактирование по своим специфическим возможностям выгодно отличалось от HDR. Как и все технологии редактирования генома, базовые редакторы могут работать с ДНК в нецелевых геномных 9 областях.0025 локус . Тем не менее, было показано, что первичное редактирование приводит к гораздо меньшему нецелевому редактированию, чем Cas9, в известных нецелевых локусах Cas9 .
Prime Editing Therapeutics
Anzalone et al. (2019) проверили способность первичного редактирования устанавливать, а затем исправлять несколько патогенных мутаций, в том числе мутации, которые, как известно, вызывают ВСС и болезни Тея-Сакса. В случае ВСС они использовали устройство PE3 для установки мутации HBB E6V в клеточной линии HEK293T. Этот подход привел к эффективности 44% и вставкам 4,8%. Чтобы скорректировать аллель HBB E6V обратно на HBB WT, они обработали гомозиготный HBB E6V HEK29.Клетки 3T с PE3 и пегРНК запрограммированы на прямой возврат мутации HBB E6V к фенотипу WT HBB. Все протестированные устройства с пегРНК опосредовали эффективную коррекцию HBB E6V до WT HBB (26–52% эффективности) и 2,8 ± 0,70% инделей. Кроме того, установка PAM-модифицирующей молчаливой мутации в устройстве PE3 повысила эффективность редактирования и чистоту продукта до 58% коэффициента коррекции с 1,4% вставок.
Что касается болезни Тея-Сакса, они создали мутантный фенотип, используя устройство PE3 для установки вставки из 4 п. н. в HEXA с эффективностью 31% и 0,8% делениями. Чтобы воссоздать фенотип WT, применение устройства PE3 привело к редактированию ≥20%, тогда как применение устройства PE3b привело к эффективности 33% с 0,32% вставок.
н. в HEXA с эффективностью 31% и 0,8% делениями. Чтобы воссоздать фенотип WT, применение устройства PE3 привело к редактированию ≥20%, тогда как применение устройства PE3b привело к эффективности 33% с 0,32% вставок.
Анзалоне и др. (2019) также удалось ввести мутацию, придающую устойчивость к прионным заболеваниям у людей (Mead et al., 2009) и мышей (Asante et al., 2015). Устройство PE3 использовалось для установки защитной трансверсии G·C-to-T·A в PRION PROTEIN 9.0026 в клеточной линии HEK293T, создавая мутантный аллель G127V, придающий устойчивость к прионным заболеваниям. Наиболее эффективная пегРНК с использованием устройства PE3 приводила к 53% установке G127V с 1,7% инделей.
Полученные результаты были дополнительно улучшены за счет введения первичного редактирования в первичные кортикальные нейроны мышей с использованием лентивирусной системы доставки. Чтобы определить, возможно ли первичное редактирование в постмитотических, терминально дифференцированных первичных клетках, они трансдуцировали первичные кортикальные нейроны от мышей E18. 5 с помощью лентивирусной системы доставки PE3, в которой белковые компоненты PE2 экспрессировались из промотора нейрон-специфического синапсин вместе с биомаркер зеленого флуоресцентного белка (Kügler et al., 2003). Было показано, что устройство PE3 было более эффективным (7,1%), генерировало меньше побочных продуктов и с меньшим нецелевым редактированием по сравнению с обычным CRISPR/Cas9.системы редактирования генома. Это указывает на то, что постмитотические, терминально дифференцированные первичные клетки могут переносить первичное редактирование.
5 с помощью лентивирусной системы доставки PE3, в которой белковые компоненты PE2 экспрессировались из промотора нейрон-специфического синапсин вместе с биомаркер зеленого флуоресцентного белка (Kügler et al., 2003). Было показано, что устройство PE3 было более эффективным (7,1%), генерировало меньше побочных продуктов и с меньшим нецелевым редактированием по сравнению с обычным CRISPR/Cas9.системы редактирования генома. Это указывает на то, что постмитотические, терминально дифференцированные первичные клетки могут переносить первичное редактирование.
Обсуждение
Разработка первичного редактирования является важным дополнением к набору инструментов редактирования генома. Основное редактирование — это самый последний из инструментов, разработанных для устранения ограничений CRISPR/Cas и калибровки процесса редактирования генома. Для академического использования технология в настоящее время может быть получена из репозитория Addgene (Kamens, 2015).
Так же, как и первичные редакторы, редакторы цитидиновых оснований и редакторы адениновых оснований могут эффективно устанавливать переходные мутации с небольшим количеством вставок. Возможно, есть особые случаи, когда обычные базовые редакторы более желательны. Например, если цель locus расположен в окне редактирования канонической базы, редактирование базы имеет более высокую эффективность и меньше вставок, чем основное редактирование. Однако применение редактирования оснований может быть ограничено нежелательным сторонним редактированием из-за присутствия множественных цитидиновых или адениновых оснований или из-за отсутствия PAM, расположенного ~15 ± 2 нт от целевого локуса . С другой стороны, первичное редактирование устраняет необходимость в совместной доставке корректирующей конструкции ДНК, фактор, который может усугубить стандартные проблемы при доставке оборудования для редактирования генома.
Возможно, есть особые случаи, когда обычные базовые редакторы более желательны. Например, если цель locus расположен в окне редактирования канонической базы, редактирование базы имеет более высокую эффективность и меньше вставок, чем основное редактирование. Однако применение редактирования оснований может быть ограничено нежелательным сторонним редактированием из-за присутствия множественных цитидиновых или адениновых оснований или из-за отсутствия PAM, расположенного ~15 ± 2 нт от целевого локуса . С другой стороны, первичное редактирование устраняет необходимость в совместной доставке корректирующей конструкции ДНК, фактор, который может усугубить стандартные проблемы при доставке оборудования для редактирования генома.
Однако основное редактирование ставит новые задачи. Основные редакторы могут быть не в состоянии обеспечить большие вставки или делеции ДНК, на которые способны обычные системы CRISPR/Cas9. Кроме того, тот факт, что желаемая последовательность должна быть закодирована в обширной молекуле РНК, вызывает опасения относительно ее стабильности; чем длиннее становится цепь РНК, тем больше вероятность того, что на нее будут воздействовать внутриклеточные ферменты, разрушающие РНК. Кроме того, благодаря наличию ОТ в молекулярном устройстве случайные кДНК потенциально могут быть включены в геном. Кроме того, поскольку задействованные белковые конструкции слишком велики, это может повлиять на доставку полноразмерного терапевтического белка с помощью одного аденоассоциированного вирусного вектора.
Кроме того, благодаря наличию ОТ в молекулярном устройстве случайные кДНК потенциально могут быть включены в геном. Кроме того, поскольку задействованные белковые конструкции слишком велики, это может повлиять на доставку полноразмерного терапевтического белка с помощью одного аденоассоциированного вирусного вектора.
Таким образом, предстоит еще много работы, прежде чем главные редакторы можно будет использовать для лечения пациентов с редкими генетическими заболеваниями. Это включает в себя подходы к оптимизации основных редакторов, максимальному повышению их эффективности в различных типах клеток и изучению потенциальных эффектов основного редактирования на разных клеточных линиях. Кроме того, необходимы дополнительные эксперименты с моделями и механизмами редких заболеваний на клеточном и организменном уровнях, а также изучение новых механизмов доставки в модельных системах животных для обеспечения потенциальных подходов к терапевтическим применениям у человека. В заключение следует отметить, что первичное редактирование, несомненно, является еще одним обоюдоострым мечом в области методов редактирования генома для лечения редких генетических заболеваний, предлагая более точные возможности и эффективность базового редактирования.
Вклад автора
Автор подтверждает, что является единственным автором этой работы и одобрил ее публикацию.
Финансирование
Исследования в лаборатории ИМ поддерживаются премией Jenkinson TIRI, Университетом Болтона, Великобритания, и Колледжем Рене Декарта, Великобритания.
Конфликт интересов
Автор заявляет, что исследование проводилось в отсутствие каких-либо коммерческих или финансовых отношений, которые могли бы быть истолкованы как потенциальный конфликт интересов.
Ссылки
Anzalone, A.V., Randolph, P.B., Davis, J.R., Sousa, A.A., Koblan, L.W., Levy, J.M., et al. (2019). Редактирование генома с помощью поиска и замены без двухцепочечных разрывов или донорской ДНК. Природа 576, 149–157. doi: 10.1038/s41586-019-1711-4
Полный текст CrossRef | Google Scholar
Асанте Э. А., Смидак М., Гримшоу А., Хоутон Р., Томлинсон А., Джилани А. и др. (2015). Встречающийся в природе вариант человеческого прионного белка полностью предотвращает прионные заболевания.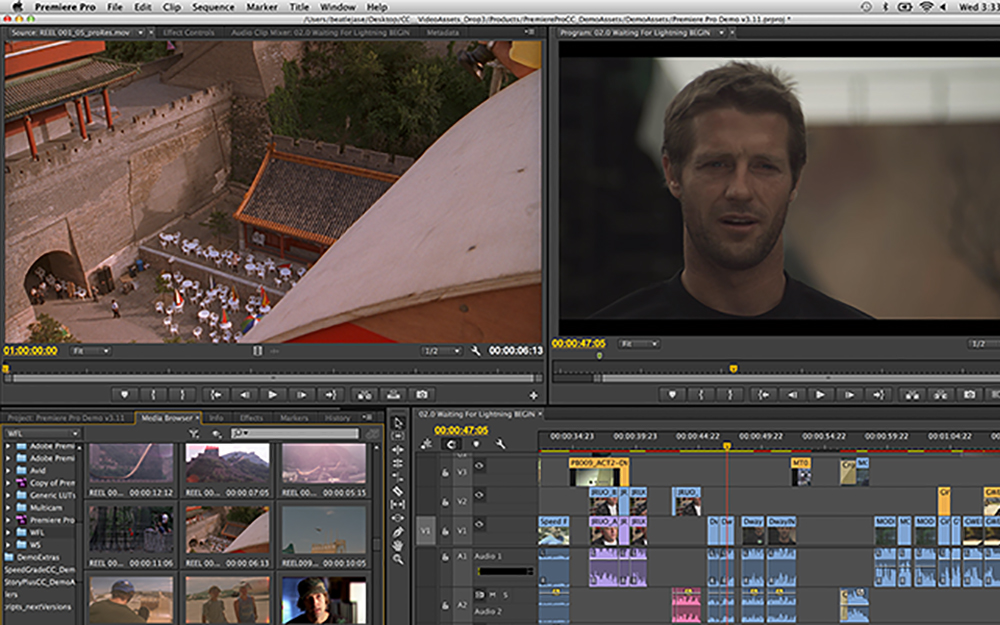 Природа 522, 478–481. doi: 10.1038/nature14510
Природа 522, 478–481. doi: 10.1038/nature14510
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Боллен Ю., Пост Дж., Ку Б.К. и Снипперт Х.Дж.Г. (2018). Как создавать современные системы генетических моделей: стратегии оптимального редактирования генома с помощью CRISPR. Рез. нуклеиновых кислот . 46, 6435–6454. doi: 10.1093/nar/gky571
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Шарпантье, Э., и Дудна, Дж. А. (2013). Переписывание генома. Природа 495:50–51. doi: 10.1038/495050a
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Конг Л., Ран Ф. А., Кокс Д., Лин С., Барретто Р., Хабиб Н. и др. (2013). Мультиплексная инженерия генома с использованием систем CRISPR/Cas. Наука 339, 819–823. doi: 10.1126/science.1231143
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Кокс Д. Б. Т., Гутенберг Дж. С., Абудайе О. О., Франклин Б., Келлнер М. Дж., Юнг Дж. и др. (2017). Редактирование РНК с помощью CRISPR-Cas13. Наука 358, 1019–1027. doi: 10.1126/science.aaq0180
О., Франклин Б., Келлнер М. Дж., Юнг Дж. и др. (2017). Редактирование РНК с помощью CRISPR-Cas13. Наука 358, 1019–1027. doi: 10.1126/science.aaq0180
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Кокс, Д. Б. Т., Платт, Р. Дж., и Чжан, Ф. (2015). Терапевтическое редактирование генома: перспективы и проблемы. Нац. Мед . 21, 121–131. doi: 10.1038/nm.3793
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Дэвис, А. Дж., и Чен, Д. Дж. (2013). Репарация двухцепочечных разрывов ДНК посредством негомологичного соединения концов. Перевод. Рак Рез . 2, 130–143. doi: 10.3978/j.issn.2218-676X.2013.04.02
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Дудна, Дж. А., и Шарпантье, Э. (2014). Новый рубеж инженерии генома с CRISPR-Cas9. Science 346:1258096.doi: 10.1126/science.1258096
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Gaudelli, N. M., Komor, A.C., Rees, H.A., Packer, M.S., Badran, A.H., Bryson, D.I., et al. (2017). Программируемое редактирование оснований T в GC в геномной ДНК без расщепления ДНК. Природа 551, 464–471. doi: 10.1038/nature24644
M., Komor, A.C., Rees, H.A., Packer, M.S., Badran, A.H., Bryson, D.I., et al. (2017). Программируемое редактирование оснований T в GC в геномной ДНК без расщепления ДНК. Природа 551, 464–471. doi: 10.1038/nature24644
Полный текст CrossRef | Google Scholar
Герке Дж. М., Сервантес О., Клемент М. К., Ву Ю., Зенг Дж., Бауэр Д. Э. и др. (2018). Базовый редактор apobec3a-cas9 с минимальными посторонними и нецелевыми действиями. Нац. Биотехнолог . 36, 77–982. doi: 10.1038/nbt.4199
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Хилтон, И. Б., и Герсбах, Калифорния (2015). Включение функциональной геномики с помощью геномной инженерии. Рез. генома . 25, 1442–1455. doi: 10.1101/gr.1
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Сюй, П. Д., Ландер, Э. С., и Чжан, Ф. (2014). Разработка и применение CRISPR-Cas9 для геномной инженерии. Сотовый 157, 1262–1278. doi: 10. 1016/j.cell.2014.05.010
1016/j.cell.2014.05.010
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Джинек М., Ист А., Ченг А., Лин С., Ма Э. и Дудна Дж. (2013). РНК-программированное редактирование генома в клетках человека. Элиф . 2:e00471. doi: 10.7554/eLife.00471
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Каменс, Дж. (2015). Репозиторий addgene: международный некоммерческий ресурс плазмид и данных. Рез. нуклеиновых кислот . 43: D1152–D1157. doi: 10.1093/nar/gku893
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Ким Х.С., Чон Ю.К., Хур Дж.К., Ким Дж.-С. и Бэ С. (2019). Редакторы оснований аденина катализируют превращения цитозина в клетках человека. Нац. Биотехнолог . 37, 1145–1148. doi: 10.1038/s41587-019-0254-4
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Ким Ю. Б., Комор А. К., Леви Дж. М., Пакер М. С., Чжао К. Т. и Лю Д. Р. (2017). Увеличение масштаба нацеливания на геном и точность редактирования базы с помощью сконструированных слияний Cas9-цитидиндезаминазы. Нац. Биотехнолог . 35, 371–376. doi: 10.1038/nbt.3803
Увеличение масштаба нацеливания на геном и точность редактирования базы с помощью сконструированных слияний Cas9-цитидиндезаминазы. Нац. Биотехнолог . 35, 371–376. doi: 10.1038/nbt.3803
PubMed Abstract | Полный текст перекрестной ссылки | Академия Google
Комор, А.С., Ким, Ю.Б., Пакер, М.С., Зурис, Дж.А., и Лю, Д.Р. (2016). Программируемое редактирование целевого основания в геномной ДНК без расщепления двухцепочечной ДНК. Природа 533, 420–424. doi: 10.1038/nature17946
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Komor, A.C., Zhao, K.T., Packer, M.S., Gaudelli, N.M., Waterbury, A.L., Koblan, L.W., et al. (2017). Улучшенное ингибирование эксцизионной репарации оснований и белок бактериофага Mu Gam дает C:G-to-T: редакторы оснований с более высокой эффективностью и чистотой продукта. Науч. Доп. 3:eaao4774. doi: 10.1126/sciadv.aao4774
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Кюглер С. , Килич Э. и Бер М. (2003). Промотор гена синапсина 1 человека обеспечивает высоко специфичную для нейронов долговременную экспрессию трансгена из аденовирусного вектора в головном мозге взрослой крысы в зависимости от трансдуцированной области. Джин Тер . 10, 337–347. doi: 10.1038/sj.gt.3301905
, Килич Э. и Бер М. (2003). Промотор гена синапсина 1 человека обеспечивает высоко специфичную для нейронов долговременную экспрессию трансгена из аденовирусного вектора в головном мозге взрослой крысы в зависимости от трансдуцированной области. Джин Тер . 10, 337–347. doi: 10.1038/sj.gt.3301905
PubMed Abstract | Полный текст перекрестной ссылки | Академия Google
Ли, X., и Хейер, В. Д. (2008). Гомологическая рекомбинация в репарации ДНК и толерантности к повреждениям ДНК. Сотовое разрешение . 18, 99–113. doi: 10.1038/cr.2008.1
Полный текст CrossRef | Google Scholar
Li, X., Wang, Y., Liu, Y., Yang, B., Wang, X., Wei, J., et al. (2018). Базовое редактирование слиянием Cpf1-цитидиндезаминазы. Нац. Биотехнолог . 36, 324–327.doi: 10.1038/nbt.4102
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Лукач Г.Л. и Веркман А.С. (2012). CFTR: сворачивание, неправильное сворачивание и исправление конформационного дефекта ΔF508. Тенденции Мол. Мед . 18, 81–91. doi: 10.1016/j.molmed.2011.10.003
Тенденции Мол. Мед . 18, 81–91. doi: 10.1016/j.molmed.2011.10.003
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Мали П., Ян Л., Эсвельт К. М., Аах Дж., Гуэлл М., ДиКарло Дж. Э. и др. (2013). Инженерия генома человека с помощью РНК с помощью Cas9. Наука 339, 823–826. doi: 10.1126/science.1232033
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Мацукас, И. Г. (2018a). Комментарий: программируемое редактирование оснований A·T в G·C в геномной ДНК без расщепления ДНК. Фронт. Гене . 9:21. doi: 10.3389/fgene.2018.00021
Полный текст CrossRef | Google Scholar
Мацукас, И. Г. (2018b). Комментарий: Редактирование РНК с помощью CRISPR-Cas13. Фронт. Гене . 9:134. doi: 10.3389/fgene.2018.00134
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Макгиннисс М.Дж., Браун Д.Х., Фулвайлер А., Мартен М., Лим-Стил Дж.С.Т. и Кабак М.М. (2002). Восемь новых мутаций в гене HEXA.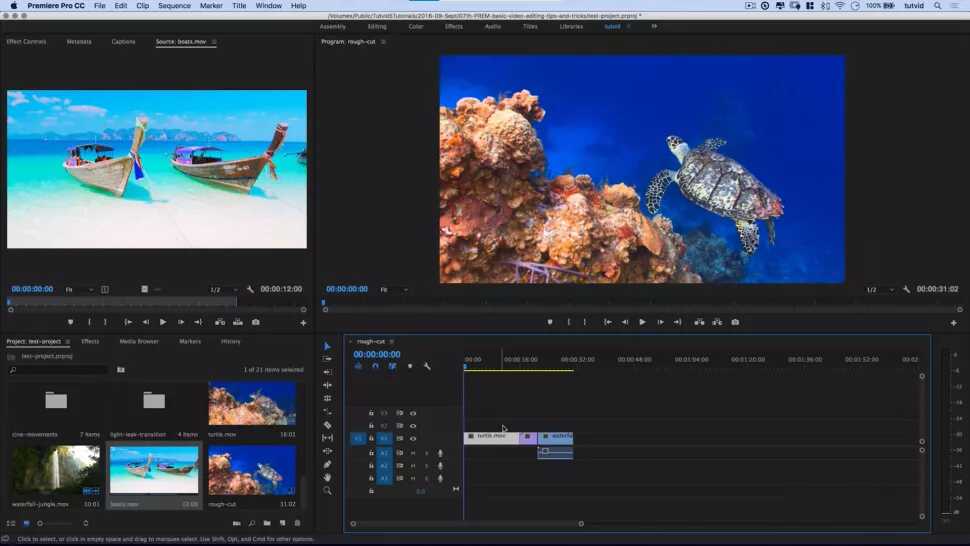 Жен. Мед . 4, 158–161. doi: 10.1097/00125817-200205000-00010
Жен. Мед . 4, 158–161. doi: 10.1097/00125817-200205000-00010
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Мид С., Уитфилд Дж., Поултер М., Шах П., Апхилл Дж., Кэмпбелл Т. и др. (2009). Новый вариант защитного прионного белка, который колокализуется при воздействии куру. Н. англ. Дж. Мед . 361, 2056–2065. doi: 10.1056/NEJMoa0809716
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Молла, К. А., и Ян, Ю. (2019). CRISPR/Cas-опосредованное базовое редактирование: технические аспекты и практическое применение. Тенденции Биотехнологии . 37, 1121–1142. doi: 10.1016/j.tibtech.2019.03.008
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Нисида К., Аразое Т., Ячи Н., Банно С., Какимото М., Табата М. и др. (2016). Направленное редактирование нуклеотидов с использованием гибридных прокариотических и позвоночных адаптивных иммунных систем. Наука 353:aaf8729. doi: 10.1126/science.aaf8729
doi: 10.1126/science.aaf8729
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Рис, Х. А., и Лю, Д. Р. (2018). Редактирование базы : прецизионная химия генома и транскриптома живых клеток. Нац. Преподобный Жене . 19, 770–788. doi: 10.1038/s41576-018-0059-1
Полный текст CrossRef | Google Scholar
Savic, N., Ringnalda, F.C.A.S., Lindsay, H., Berk, C., Bargsten, K., Li, Y., et al. (2018). Ковалентное связывание матрицы репарации ДНК с нуклеазой CRISPR-Cas9 усиливает репарацию, направленную на гомологию. Элиф . 7:e33761. doi: 10.7554/eLife.33761
PubMed Abstract | Полный текст перекрестной ссылки | Академия Google
Шарон, Э., Чен, С.А.А., Хосла, Н.М., Смит, Дж.Д., Притчард, Дж.К., и Фрейзер, Х.Б. (2018). Функциональные генетические варианты, выявленные путем массового параллельного точного редактирования генома. Сотовый 175, 544–557.e16. doi: 10.1016/j.cell.2018.08.057
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Сонг Ф. и Стигер К. (2017). Оптимизация матрицы донора ДНК для направленной по гомологии репарации двухцепочечных разрывов. Мол. тер. Нуклеиновые кислоты . 7, 53–60. doi: 10.1016/j.omtn.2017.02.006
и Стигер К. (2017). Оптимизация матрицы донора ДНК для направленной по гомологии репарации двухцепочечных разрывов. Мол. тер. Нуклеиновые кислоты . 7, 53–60. doi: 10.1016/j.omtn.2017.02.006
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Штернберг С. Х. и Дудна Дж. А. (2015). Расширение набора инструментов биолога с помощью CRISPR-Cas9. Мол. Сотовый . 58, 568–574. doi: 10.1016/j.molcel.2015.02.032
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Вакульскас, К.А., Девер, Д.П., Реттиг, Г.Р., Терк, Р., Якоби, А.М., Коллингвуд, М.А., и др. (2018). Высокоточный Cas9мутант, доставляемый в виде рибонуклеопротеинового комплекса, обеспечивает эффективное редактирование генов в гемопоэтических стволовых клетках человека и клетках-предшественниках. Нац. Мед . 24, 1216–1224. doi: 10.1038/s41591-018-0137-0
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Ван X., Ли Дж. , Ван Ю., Ян Б., Вэй Дж., Ву Дж. и др. (2018). Эффективное редактирование оснований в метилированных областях с помощью слияния apobec3a-cas9 человека. Нац. Биотехнолог . 36, 946–949. doi: 10.1038/nbt.4198
, Ван Ю., Ян Б., Вэй Дж., Ву Дж. и др. (2018). Эффективное редактирование оснований в метилированных областях с помощью слияния apobec3a-cas9 человека. Нац. Биотехнолог . 36, 946–949. doi: 10.1038/nbt.4198
Реферат PubMed | Полный текст перекрестной ссылки | Google Scholar
Ян Б., Ян Л. и Чен Дж. (2019). Разработка и применение базовых редакторов. Крис. Дж . 2, 91–104. doi: 10.1089/crispr.2019.0001
PubMed Abstract | Полный текст перекрестной ссылки | Google Scholar
Ян Л., Ян Дж. Л., Бирн С., Пан Дж. и Черч Г. М. (2014). CRISPR/Cas9-направленное редактирование генома культивируемых клеток. Курс. протокол Мол. Биол . 107, 31.1.1-31.1.17. дои: 10.1002/0471142727.mb3101s107
Реферат PubMed | Полный текст перекрестной ссылки | Google Scholar
Веб-инструмент для разработки руководства по прайм-редактированию РНК
- Список журналов
- Рукописи авторов HHS
- PMC7882013
Nat Biomed Eng. Авторская рукопись; доступно в PMC 2021 28 марта.
Авторская рукопись; доступно в PMC 2021 28 марта.
Опубликовано в окончательной редакции как:
Nat Biomed Eng. 2021 февраль; 5(2): 190–194.
Published online 2020 Sep 28. doi: 10.1038/s41551-020-00622-8
PMCID: PMC7882013
NIHMSID: NIHMS1625906
PMID: 32989284
, 1, 2, 3, 4, и , 4, 5, 6, 7, и , 1, 36, 2, 0235 3 and 1, 2, 3, 4, 7, 8, 9, 10, 11, 12, 13, 14 , *
Информация об авторе Информация об авторских правах и лицензиях Отказ от ответственности
- Дополнительные материалы
- Заявление о доступности данных .
 Однако дизайн направляющих РНК первичного редактирования (pegRNAs), которые необходимо настраивать для каждого редактирования, может быть сложным и занимать много времени. По сравнению с одиночными направляющими РНК (sgRNAs), pegRNAs имеют дополнительное 3′-удлинение, состоящее из сайта связывания праймера и матрицы обратной транскрипции. Здесь мы сообщаем о веб-инструменте, который мы назвали pegFinder (http://pegfinder.sidichenlab.org), для быстрого проектирования пегРНК из эталонных и отредактированных последовательностей ДНК. pegFinder может включать в свою систему ранжирования прогнозы оценки целевого и нецелевого sgRNA и номинирует вторичные никирующие sgRNA для повышения эффективности редактирования. Cas9также поддерживаются варианты с расширенными диапазонами таргетинга. Чтобы облегчить дальнейшие эксперименты, pegFinder создает исчерпывающую таблицу пегРНК-кандидатов, а также олигонуклеотидные последовательности для клонирования.
Однако дизайн направляющих РНК первичного редактирования (pegRNAs), которые необходимо настраивать для каждого редактирования, может быть сложным и занимать много времени. По сравнению с одиночными направляющими РНК (sgRNAs), pegRNAs имеют дополнительное 3′-удлинение, состоящее из сайта связывания праймера и матрицы обратной транскрипции. Здесь мы сообщаем о веб-инструменте, который мы назвали pegFinder (http://pegfinder.sidichenlab.org), для быстрого проектирования пегРНК из эталонных и отредактированных последовательностей ДНК. pegFinder может включать в свою систему ранжирования прогнозы оценки целевого и нецелевого sgRNA и номинирует вторичные никирующие sgRNA для повышения эффективности редактирования. Cas9также поддерживаются варианты с расширенными диапазонами таргетинга. Чтобы облегчить дальнейшие эксперименты, pegFinder создает исчерпывающую таблицу пегРНК-кандидатов, а также олигонуклеотидные последовательности для клонирования.Технологии, основанные на кластерных регулярно расположенных коротких палиндромных повторах (CRISPR), получили широкое распространение в качестве мощных инструментов для целенаправленных геномных манипуляций 1 .
/cc0076d6e2a8d21.s.siteapi.org/img/26b867ebb77ab1960b0973d095f63ebe70b0e85f.jpg) Недавно была разработана новая основанная на CRISPR стратегия точного редактирования генома, которая позволяет напрямую вписывать различные геномные изменения в целевые сайты, не требуя двухцепочечных разрывов (DSB) или донорских матриц 2 . Этот подход, называемый первичным редактированием, включает два ключевых компонента: 1) каталитически нарушенную CRISPR-ассоциированную протеин 9 (Cas9) никазу, слитую с обратной транскриптазой (PE2), и 2) многофункциональную направляющую РНК первичного редактирования (pegRNA), которая определяет мишень. сайте и в дальнейшем выступает в качестве шаблона для обратной транскрипции (ОТ). pegRNAs похожи на одиночные направляющие РНК (sgRNAs), но дополнительно имеют настраиваемое удлинение на 3′-конце. 3’-удлинение состоит из матрицы RT, которая кодирует желаемое редактирование, и сайта связывания праймеров (PBS), который отжигается с целевым геномным сайтом для праймирования реакции RT 9.0235 2 . Вторичные разрывы sgRNAs также могут быть использованы для потенциального увеличения эффективности первичного редактирования путем разрыва противоположной нити, таким образом благоприятствуя редактируемой нити во время разрешения гетеродуплекса (PE3) 2 .
Недавно была разработана новая основанная на CRISPR стратегия точного редактирования генома, которая позволяет напрямую вписывать различные геномные изменения в целевые сайты, не требуя двухцепочечных разрывов (DSB) или донорских матриц 2 . Этот подход, называемый первичным редактированием, включает два ключевых компонента: 1) каталитически нарушенную CRISPR-ассоциированную протеин 9 (Cas9) никазу, слитую с обратной транскриптазой (PE2), и 2) многофункциональную направляющую РНК первичного редактирования (pegRNA), которая определяет мишень. сайте и в дальнейшем выступает в качестве шаблона для обратной транскрипции (ОТ). pegRNAs похожи на одиночные направляющие РНК (sgRNAs), но дополнительно имеют настраиваемое удлинение на 3′-конце. 3’-удлинение состоит из матрицы RT, которая кодирует желаемое редактирование, и сайта связывания праймеров (PBS), который отжигается с целевым геномным сайтом для праймирования реакции RT 9.0235 2 . Вторичные разрывы sgRNAs также могут быть использованы для потенциального увеличения эффективности первичного редактирования путем разрыва противоположной нити, таким образом благоприятствуя редактируемой нити во время разрешения гетеродуплекса (PE3) 2 . Чтобы повысить специфичность и уменьшить вероятность образования нежелательных DSB, вторичные разрывные sgRNA могут быть сконструированы таким образом, что они становятся активными только после успешного первичного редактирования (PE3b).
Чтобы повысить специфичность и уменьшить вероятность образования нежелательных DSB, вторичные разрывные sgRNA могут быть сконструированы таким образом, что они становятся активными только после успешного первичного редактирования (PE3b).Эти дополнительные компоненты значительно усложняют дизайн пегРНК по сравнению со стандартными sgRNA. В частности, было продемонстрировано, что точная длина матрицы RT и последовательности PBS значительно влияет на эффективность первичного редактирования 2-4 . Вдохновленные разнообразием инструментов, которые были разработаны для идентификации кандидатных sgRNAs в целевой последовательности ДНК 5-11 , мы стремились создать удобное приложение для разработки pegRNAs. Поэтому мы разработали pegFinder, оптимизированный веб-инструмент, который быстро проектирует и ранжирует pegRNA-кандидаты для указанной пользователем генетической модификации. Веб-портал pegFinder находится в свободном доступе по адресу http://pegfinder.sidichenlab.
 org (дополнительный рисунок 1).
org (дополнительный рисунок 1).Открыть в отдельном окне
pegFinder: конструктор пегРНК для первичного редактирования CRISPR.
a . Схема рабочего процесса pegFinder для разработки пегРНК для первичного редактирования CRISPR. b , Критерии, по которым pegFinder идентифицирует спейсеры-кандидаты пегРНК, подходящие для желаемого редактирования генома. c , Обзор факторов, учитываемых pegFinder при ранжировании и выборе спейсеров-кандидатов pegRNA. d , Принципы дизайна, согласно которым pegFinder отдает приоритет последовательностям PBS и матрицам RT, которые составляют 3′-удлинение пегРНК.
pegFinder просто требует двух входных данных: 1) последовательность ДНК дикого типа / эталона целевого сайта и 2) отредактированная / желаемая последовательность ДНК (дополнительный рис. 2 и дополнительная таблица 1). Для рассмотрения прогнозируемых показателей соответствия/несоответствия пользователь может дополнительно включить результаты дизайнера Broad sgRNA (https://portals.
 broadinstitute.org/gpp/public/analysis-tools/sgrna-design) 6 ,7 или CRISPRscan (https://www.crisprscan.org/?page=sequence) 8 с использованием последовательности ДНК дикого типа в качестве входных данных (). При желании также можно указать предварительно выбранную спейсерную последовательность sgRNA. После проверки входных данных pegFinder сначала идентифицирует различия между последовательностями ДНК дикого типа и отредактированными, выполняя выравнивание Needleman-Wunsch с штрафами за аффинные пробелы 12 . Используя выравнивание, pegFinder выбирает один спейсер sgRNA из подходящих кандидатов. pegFinder отдает приоритет спейсерам, чьи целевые сайты будут нарушены после основного редактирования, дополнительно учитывая расстояние между сайтом с псевдонимом и желаемыми правками (-). Если это предусмотрено, pegFinder будет учитывать целевые/нецелевые баллы при выборе кандидатов-спейсеров. Затем
broadinstitute.org/gpp/public/analysis-tools/sgrna-design) 6 ,7 или CRISPRscan (https://www.crisprscan.org/?page=sequence) 8 с использованием последовательности ДНК дикого типа в качестве входных данных (). При желании также можно указать предварительно выбранную спейсерную последовательность sgRNA. После проверки входных данных pegFinder сначала идентифицирует различия между последовательностями ДНК дикого типа и отредактированными, выполняя выравнивание Needleman-Wunsch с штрафами за аффинные пробелы 12 . Используя выравнивание, pegFinder выбирает один спейсер sgRNA из подходящих кандидатов. pegFinder отдает приоритет спейсерам, чьи целевые сайты будут нарушены после основного редактирования, дополнительно учитывая расстояние между сайтом с псевдонимом и желаемыми правками (-). Если это предусмотрено, pegFinder будет учитывать целевые/нецелевые баллы при выборе кандидатов-спейсеров. ЗатемpegFinder идентифицирует соответствующий шаблон RT и последовательность PBS для создания желаемого редактирования путем оценки положения отредактированных оснований и содержания GC sgRNA соответственно (и дополнительная рис.
 2). Кроме того, pegFinder идентифицирует вторичные sgRNA, которые надрезают 40-150 нуклеотидов (настройка по умолчанию) на противоположной цепи от первичной sgRNA (PE3), а также надрезают sgRNA, которые становятся активными только после первичного редактирования (PE3b). Чтобы облегчить быструю экспериментальную реализацию, pegFinder дополнительно генерирует последовательности олигонуклеотидов, которые можно напрямую использовать для клонирования пегРНК в стандартные плазмидные векторы (дополнительная рис. 2).
2). Кроме того, pegFinder идентифицирует вторичные sgRNA, которые надрезают 40-150 нуклеотидов (настройка по умолчанию) на противоположной цепи от первичной sgRNA (PE3), а также надрезают sgRNA, которые становятся активными только после первичного редактирования (PE3b). Чтобы облегчить быструю экспериментальную реализацию, pegFinder дополнительно генерирует последовательности олигонуклеотидов, которые можно напрямую использовать для клонирования пегРНК в стандартные плазмидные векторы (дополнительная рис. 2).Учитывая, что первичное редактирование было разработано совсем недавно, правила, управляющие дизайном pegRNA, изучены не полностью. Таким образом, может потребоваться оптимизация пегРНК для каждого экспериментального применения. Поскольку было показано, что эффективность первичного редактирования широко варьируется в зависимости от длины и / или основного состава шаблона RT и PBS 2-4 , pegFinder сообщает о шаблонах RT и последовательностях PBS различной длины (дополнительная рис.
 2). pegFinder также создает загружаемую таблицу, содержащую исчерпывающий каталог кандидатов пегРНК для каждого из спейсеров sgRNA с самым высоким рейтингом, а также соответствующие клонирующие олигопоследовательности (дополнительная таблица 2). Эта таблица может быть непосредственно использована для создания библиотек пегРНК, которые исчерпывающе тестируют различные комбинации спейсеров sgRNA, последовательностей PBS и шаблонов RT для желаемого редактирования. Таким образом, pegFinder может облегчить дальнейшую оптимизацию экспериментов по первичному редактированию.
2). pegFinder также создает загружаемую таблицу, содержащую исчерпывающий каталог кандидатов пегРНК для каждого из спейсеров sgRNA с самым высоким рейтингом, а также соответствующие клонирующие олигопоследовательности (дополнительная таблица 2). Эта таблица может быть непосредственно использована для создания библиотек пегРНК, которые исчерпывающе тестируют различные комбинации спейсеров sgRNA, последовательностей PBS и шаблонов RT для желаемого редактирования. Таким образом, pegFinder может облегчить дальнейшую оптимизацию экспериментов по первичному редактированию.Для проверки алгоритма проектирования мы сначала сопоставили конструкции pegRNA, рекомендованные pegFinder, с экспериментальными данными, используя первичное редактирование в клетках человека 2 , клетках мыши 3 и растениях 4 , обнаружив, что pegFinder успешно идентифицировал функциональные pegRNAs в этих системах (дополнительное примечание). Затем мы использовали pegFinder для разработки двух разных пегРНК, нацеленных на человеческий локус HEK3 (также известный как LINC01509 ).
 Первая пегРНК была разработана для вставки «СТТ» в ту же геномную позицию, что и одна из пегРНК, описанных в исходном исследовании первичного редактирования 9.0235 2 и, таким образом, служил эталоном положительного контроля. При минимальном участии пользователя pegFinder разработал пегРНК-кандидат CTTins, которая была в значительной степени идентична последовательности пегРНК CTTins, описанной ранее 2 , что демонстрирует точность алгоритма. Олигонуклеотидные последовательности, созданные с помощью pegFinder, затем непосредственно использовали для лигирования клонирования (Методы). По сравнению с контрольными клетками, совместно трансфицированными PE2 и пустым вектором (), клетки, котрансфицированные PE2 и пегРНК CTTins, сконструированной с помощью pegFinder, демонстрировали признаки первичного редактирования, что определялось анализом второстепенных пиков на хроматограммах секвенирования (). Аналогичным образом мы использовали pegFinder для разработки pegRNA для вставки «CT» в HEK3 локус; это конкретное редактирование (CTins) не выполнялось в исходном исследовании 2 .
Первая пегРНК была разработана для вставки «СТТ» в ту же геномную позицию, что и одна из пегРНК, описанных в исходном исследовании первичного редактирования 9.0235 2 и, таким образом, служил эталоном положительного контроля. При минимальном участии пользователя pegFinder разработал пегРНК-кандидат CTTins, которая была в значительной степени идентична последовательности пегРНК CTTins, описанной ранее 2 , что демонстрирует точность алгоритма. Олигонуклеотидные последовательности, созданные с помощью pegFinder, затем непосредственно использовали для лигирования клонирования (Методы). По сравнению с контрольными клетками, совместно трансфицированными PE2 и пустым вектором (), клетки, котрансфицированные PE2 и пегРНК CTTins, сконструированной с помощью pegFinder, демонстрировали признаки первичного редактирования, что определялось анализом второстепенных пиков на хроматограммах секвенирования (). Аналогичным образом мы использовали pegFinder для разработки pegRNA для вставки «CT» в HEK3 локус; это конкретное редактирование (CTins) не выполнялось в исходном исследовании 2 .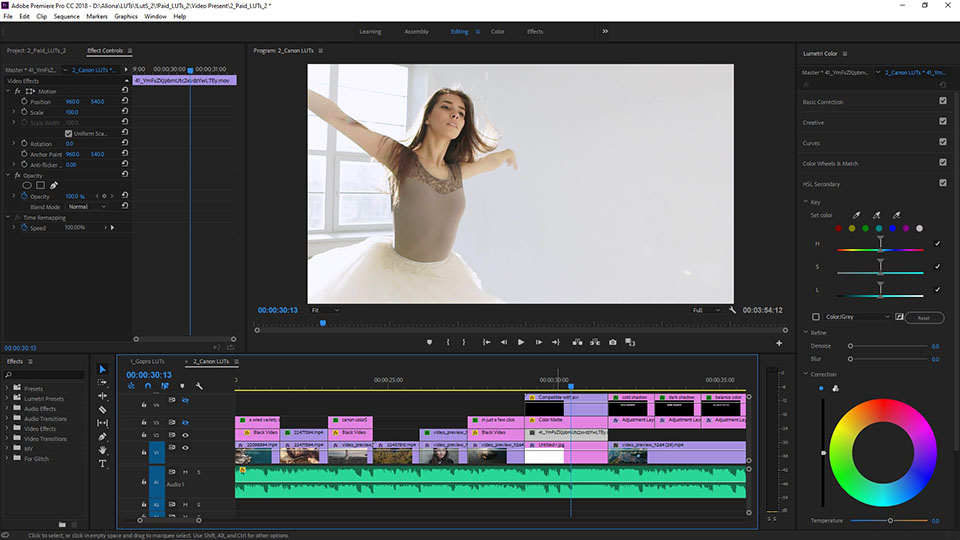 Используя конструкции, полученные с помощью pegFinder, мы наблюдали признаки первичного редактирования в клетках, трансфицированных pegRNA CTins (1), экспериментально демонстрируя функциональность pegRNAs, сконструированных pegFinder.
Используя конструкции, полученные с помощью pegFinder, мы наблюдали признаки первичного редактирования в клетках, трансфицированных pegRNA CTins (1), экспериментально демонстрируя функциональность pegRNAs, сконструированных pegFinder.Открыть в отдельном окне
Экспериментальная проверка пегРНК, разработанных pegFinder.
a , Хроматограмма секвенирования Сайт-мишень HEK3 в клетках, трансфицированных контрольным вектором. b , Хроматограмма секвенирования сайта-мишени HEK3 в клетках, трансфицированных пегРНК, предназначенной для вставки «СТТ» в положение +1 вместе с вторичной никсинговой sgRNA. c . Хроматограмма секвенирования сайта-мишени HEK3 в клетках, трансфицированных пегРНК, предназначенной для вставки «CT» в положение +1 вместе с вторичной никирующей sgRNA. Показаны репрезентативные данные из трех независимых экспериментов.

Вместе эти данные демонстрируют простоту и полезность pegFinder для дизайна pegRNA. pegFinder — это удобный инструмент для исследователей в различных областях, позволяющий быстро освоить универсальность основного редактирования.
Разработка алгоритма pegFinder и веб-сервера
Основной алгоритм pegFinder был разработан на Perl. Веб-портал был реализован в веб-фреймворке реального времени Mojolicious на Perl.
Входы для pegFinder
Для pegFinder требуется как минимум два входа. Во-первых, необходима последовательность ДНК дикого типа интересующей области. Поскольку sgRNAs-кандидаты должны быть найдены в этой последовательности дикого типа, мы рекомендуем > 100 nt флангов вокруг желаемого сайта редактирования. Эти последовательности можно легко найти с помощью браузеров генома, таких как UCSC, IGV или Ensembl BioMart. Во-вторых, отредактированная последовательность ДНК должна быть получена путем модификации последовательности дикого типа для включения желаемых изменений.
 Обратите внимание, что pegFinder ожидает, что последовательности дикого типа и отредактированные последовательности будут иметь одинаковые 5- и 3-концы, и уведомит пользователя, если это не так. В качестве примера рассмотрим последовательность ДНК дикого типа из 200 нуклеотидов, в которую пользователь хочет вставить последовательность из 10 нуклеотидов после 100 -й нуклеотид. Затем отредактированная последовательность должна быть следующей: 100 нуклеотидов — вставка 10 нуклеотидов — 100 нуклеотидов фланк, где 5’ и 3’ 100 нуклеотидов вокруг вставки точно соответствуют 200-нуклеотидной последовательности дикого типа. Таким образом, мы рекомендуем генерировать отредактированную последовательность ДНК, напрямую изменяя ввод ДНК дикого типа, поскольку это гарантирует, что 5 ‘и 3’ фланги останутся идентичными.
Обратите внимание, что pegFinder ожидает, что последовательности дикого типа и отредактированные последовательности будут иметь одинаковые 5- и 3-концы, и уведомит пользователя, если это не так. В качестве примера рассмотрим последовательность ДНК дикого типа из 200 нуклеотидов, в которую пользователь хочет вставить последовательность из 10 нуклеотидов после 100 -й нуклеотид. Затем отредактированная последовательность должна быть следующей: 100 нуклеотидов — вставка 10 нуклеотидов — 100 нуклеотидов фланк, где 5’ и 3’ 100 нуклеотидов вокруг вставки точно соответствуют 200-нуклеотидной последовательности дикого типа. Таким образом, мы рекомендуем генерировать отредактированную последовательность ДНК, напрямую изменяя ввод ДНК дикого типа, поскольку это гарантирует, что 5 ‘и 3’ фланги останутся идентичными.По умолчанию pegFinder основывает свои проекты pegRNA на использовании Cas9 дикого типа с мотивом, прилегающим к протоспейсеру NGG (PAM). pegFinder также поддерживает использование Cas9.
 Варианты -NG или Cas9-SpRY 13 , которые расширяют потенциальный диапазон первичных редакторов, хотя такие конструкции еще не тестировались экспериментально. При использовании Cas9-NGG в качестве фермента CRISPR pegFinder может дополнительно включать предсказанные оценки соответствия/несоответствия цели с помощью инструментов разработки sgRNA с оговоркой, что эти алгоритмы оценки были обучены на данных нокаута гена и, следовательно, могут не иметь отношения к первичным монтажные эксперименты. pegFinder может использовать результаты инструмента разработки Broad sgRNA 6,7 (https://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design) или CRISPRscan 8 (https://www.crisprscan.org/?page=sequence), с последовательностью ДНК дикого типа, описанной выше, в качестве входного запроса. Для дизайнера Broad следует выбрать фермент CRISPR SpyoCas9 (NGG) и соответствующий целевой геном. Также обратите внимание, что следует сообщать обо всех невыбранных последовательностях.
Варианты -NG или Cas9-SpRY 13 , которые расширяют потенциальный диапазон первичных редакторов, хотя такие конструкции еще не тестировались экспериментально. При использовании Cas9-NGG в качестве фермента CRISPR pegFinder может дополнительно включать предсказанные оценки соответствия/несоответствия цели с помощью инструментов разработки sgRNA с оговоркой, что эти алгоритмы оценки были обучены на данных нокаута гена и, следовательно, могут не иметь отношения к первичным монтажные эксперименты. pegFinder может использовать результаты инструмента разработки Broad sgRNA 6,7 (https://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design) или CRISPRscan 8 (https://www.crisprscan.org/?page=sequence), с последовательностью ДНК дикого типа, описанной выше, в качестве входного запроса. Для дизайнера Broad следует выбрать фермент CRISPR SpyoCas9 (NGG) и соответствующий целевой геном. Также обратите внимание, что следует сообщать обо всех невыбранных последовательностях. Полученный файл результатов с разделителями табуляции («Результаты выбора sgRNA») можно сохранить и загрузить в pegFinder. Для CRISPRscan следует выбрать правильный целевой вид, фермент должен быть установлен как Cas9.-NGG, и следует выбрать вариант поиска sgRNAs как с промоторов T7, так и с промоторов Sp6. Созданный файл с разделителями табуляции можно сохранить и загрузить в pegFinder. В качестве другой альтернативы, если пользователь желает указать предварительно выбранную sgRNA, последовательность спейсера из 20 нуклеотидов может быть введена непосредственно в pegFinder. Если была указана предварительно выбранная sgRNA, pegFinder проверит, правильно ли расположена выбранная sgRNA для получения желаемых изменений.
Полученный файл результатов с разделителями табуляции («Результаты выбора sgRNA») можно сохранить и загрузить в pegFinder. Для CRISPRscan следует выбрать правильный целевой вид, фермент должен быть установлен как Cas9.-NGG, и следует выбрать вариант поиска sgRNAs как с промоторов T7, так и с промоторов Sp6. Созданный файл с разделителями табуляции можно сохранить и загрузить в pegFinder. В качестве другой альтернативы, если пользователь желает указать предварительно выбранную sgRNA, последовательность спейсера из 20 нуклеотидов может быть введена непосредственно в pegFinder. Если была указана предварительно выбранная sgRNA, pegFinder проверит, правильно ли расположена выбранная sgRNA для получения желаемых изменений.Выравнивание последовательностей ДНК дикого типа и отредактированных последовательностей ДНК
Первым шагом pegFinder является выравнивание последовательностей ДНК дикого типа и отредактированных последовательностей с использованием алгоритма Нидлмана-Вунша с функцией штрафа за аффинные пробелы 12 .
 Отмечаются положения пробелов и несовпадающих оснований, которые затем используются для выбора sgRNA-кандидатов.
Отмечаются положения пробелов и несовпадающих оснований, которые затем используются для выбора sgRNA-кандидатов.Выбор первичных разрывных спейсеров sgRNA для pegRNAs
Если предварительно выбранный спейсер sgRNA не был указан, pegFinder идентифицирует спейсеры-кандидаты sgRNA de novo . После определения диапазона позиций желаемых изменений pegFinder ищет разделители, которые потенциально могут опосредовать желаемый основной результат редактирования. pegFinder определяет спейсеры sgRNA на «смысловой» цепи в качестве потенциальных кандидатов, если выполняются следующие условия: 1) положение разреза sgRNA выше по течению от 5′-наиболее редактируемого основания, 2) 3′-наиболее редактируемое основание находится в пределах 150 н. положение разреза (50 нт для Cas9-NG и 20 нуклеотидов для Cas9-SpRY, учитывая значительно возросшее количество спейсеров-кандидатов для этих вариантов), 3) спейсер не содержит 5 или более последовательных тимидинов, которые могли бы терминировать транскрипцию U6, и, если применимо, 4) спейсер sgRNA имеет общая «нецелевая» оценка уровня I Bin I + Bin II ≤ 1 (согласно определению дизайнера Broad sgRNA) или количество «исходных нецелевых значений» равно 0 (согласно определению CRISPRscan).
 В то время как оценка Уровня I, равная 1 в конструкторе Broad, обычно указывает на идеальное соответствие нецелевому сайту, в этом контексте она будет просто соответствовать геномному локусу входной последовательности и, следовательно, может быть проигнорирована. Точно так же для спейсеров sgRNA на «антисмысловой» цепи pegFinder определяет спейсер как кандидата, если: 1) положение разреза sgRNA находится ниже 3′-наиболее редактируемого основания (в смысловой ориентации), 2) 5′- наиболее редактируемая база (смысловая ориентация) находится в пределах 150 нт от положения разреза (50 нт для Cas9-NG и 20 нуклеотидов для Cas9-SpRY), 3) нет поли(Т)-трактов, и, если применимо, 4) sgRNA имеет общую «нецелевую» оценку уровня I Bin I + Bin II ≤ 1 (широкий конструктор sgRNA) или количество «начальных отклонений от целей», равное 0 (CRISPRscan).
В то время как оценка Уровня I, равная 1 в конструкторе Broad, обычно указывает на идеальное соответствие нецелевому сайту, в этом контексте она будет просто соответствовать геномному локусу входной последовательности и, следовательно, может быть проигнорирована. Точно так же для спейсеров sgRNA на «антисмысловой» цепи pegFinder определяет спейсер как кандидата, если: 1) положение разреза sgRNA находится ниже 3′-наиболее редактируемого основания (в смысловой ориентации), 2) 5′- наиболее редактируемая база (смысловая ориентация) находится в пределах 150 нт от положения разреза (50 нт для Cas9-NG и 20 нуклеотидов для Cas9-SpRY), 3) нет поли(Т)-трактов, и, если применимо, 4) sgRNA имеет общую «нецелевую» оценку уровня I Bin I + Bin II ≤ 1 (широкий конструктор sgRNA) или количество «начальных отклонений от целей», равное 0 (CRISPRscan).pegFinder затем ранжирует спейсеры-кандидаты sgRNA на основе их расстояния до первого отредактированного основания, отдавая приоритет спейсерам, сайты-мишени которых будут нарушены при успешном прайм-редактировании (т.
 е. мутациях в исходной области и/или PAM). Поскольку нарушение сайта-мишени pegRNA уменьшит вероятность повторного редактирования, предполагается, что такие спейсеры будут иметь более высокую эффективность первичного редактирования. При включении показателей прогнозирования целевых показателей из инструментов дизайнера sgRNA pegFinder дополнительно отдает приоритет спейсерам sgRNA с более высокой эффективностью при попадании в цель. Если есть спейсеры-кандидаты с высокими целевыми баллами ≥ 0,5 (широкий дизайнер) или ≥ 25 (CRISPRscan), pegFinder выберет спейсер с кратчайшим расстоянием до ближайшей позиции редактирования, снова отдавая приоритет спейсерам, которые больше не будут работать после успешного завершения. первичное редактирование. В случае совпадения pegFinder выбирает sgRNA с более высоким показателем попадания в цель, если он предоставлен.
е. мутациях в исходной области и/или PAM). Поскольку нарушение сайта-мишени pegRNA уменьшит вероятность повторного редактирования, предполагается, что такие спейсеры будут иметь более высокую эффективность первичного редактирования. При включении показателей прогнозирования целевых показателей из инструментов дизайнера sgRNA pegFinder дополнительно отдает приоритет спейсерам sgRNA с более высокой эффективностью при попадании в цель. Если есть спейсеры-кандидаты с высокими целевыми баллами ≥ 0,5 (широкий дизайнер) или ≥ 25 (CRISPRscan), pegFinder выберет спейсер с кратчайшим расстоянием до ближайшей позиции редактирования, снова отдавая приоритет спейсерам, которые больше не будут работать после успешного завершения. первичное редактирование. В случае совпадения pegFinder выбирает sgRNA с более высоким показателем попадания в цель, если он предоставлен.Выбор RT-матриц и последовательностей PBS
После выбора первичной sgRNA (или непосредственного использования предварительно выбранной sgRNA, указанной пользователем) pegFinder затем извлекает кандидатные RT-матрицы и последовательности PBS, которые могут быть включены в 3’-удлинение pegRNAs.
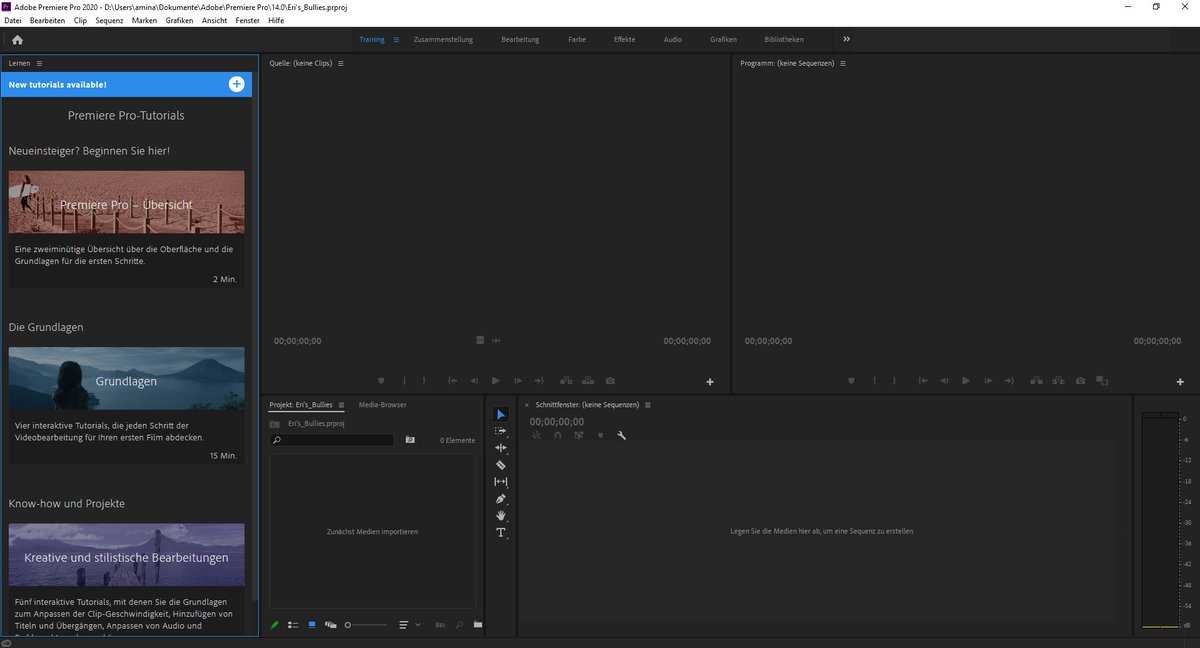 Для разработки шаблонов RT pegFinder использует отредактированную/нужную последовательность и извлекает ДНК между основным ник-сайтом и самым дальним отредактированным основанием (самое 3′-основание при использовании смысловой цепи sgRNA или 5′-самое основание, если антисмысловая) , плюс дополнительный 1нт. Если результирующая последовательность < 10 нуклеотидов, pegFinder сообщит о шаблонах-кандидатах RT длиной от 10 до 17 нуклеотидов. Если расстояние между основным сайтом ника и самой дальней редактируемой базой составляет ≥ 10 нт, pegFinder сообщит шаблоны RT в диапазоне от +1 до +7 нт от ника до расстояния редактирования. Во всех случаях pegFinder будет помечать RT-шаблоны, которые имеют «C» в качестве своего 5-футового основания (соответствующее «G» в качестве окончательного основания шаблона), поскольку ранее было продемонстрировано, что такие RT-шаблоны демонстрируют более низкую эффективность для основного редактирования. , потенциально из-за взаимодействий спаривания оснований с каркасом sgRNA 2 .
Для разработки шаблонов RT pegFinder использует отредактированную/нужную последовательность и извлекает ДНК между основным ник-сайтом и самым дальним отредактированным основанием (самое 3′-основание при использовании смысловой цепи sgRNA или 5′-самое основание, если антисмысловая) , плюс дополнительный 1нт. Если результирующая последовательность < 10 нуклеотидов, pegFinder сообщит о шаблонах-кандидатах RT длиной от 10 до 17 нуклеотидов. Если расстояние между основным сайтом ника и самой дальней редактируемой базой составляет ≥ 10 нт, pegFinder сообщит шаблоны RT в диапазоне от +1 до +7 нт от ника до расстояния редактирования. Во всех случаях pegFinder будет помечать RT-шаблоны, которые имеют «C» в качестве своего 5-футового основания (соответствующее «G» в качестве окончательного основания шаблона), поскольку ранее было продемонстрировано, что такие RT-шаблоны демонстрируют более низкую эффективность для основного редактирования. , потенциально из-за взаимодействий спаривания оснований с каркасом sgRNA 2 . По умолчанию pegFinder затем выбирает один шаблон RT, выбирая шаблон, представляющий медианную длину среди кандидатов, которые не начинаются с «C», выбирая более короткий шаблон, если имеется четное количество кандидатов. Если нет шаблонов RT, которые не начинаются с «C», pegFinder выберет шаблон средней длины среди всех кандидатов. Поскольку длина шаблона RT может потребовать дальнейшей оптимизации, обо всех шаблонах RT-кандидатах также сообщает pegFinder.
По умолчанию pegFinder затем выбирает один шаблон RT, выбирая шаблон, представляющий медианную длину среди кандидатов, которые не начинаются с «C», выбирая более короткий шаблон, если имеется четное количество кандидатов. Если нет шаблонов RT, которые не начинаются с «C», pegFinder выберет шаблон средней длины среди всех кандидатов. Поскольку длина шаблона RT может потребовать дальнейшей оптимизации, обо всех шаблонах RT-кандидатах также сообщает pegFinder.Для конструирования последовательностей PBS pegFinder извлекает последовательности длиной 8–17 нуклеотидов из обратной комплементарности первичной последовательности sgRNA, двигаясь назад от положения -1 (положения перед участком разреза). Следуя рекомендациям исходного исследования 2 , pegFinder выбирает одну длину PBS на основе содержания GC в спейсере. В частности, pegFinder использует следующую формулу: рекомендуемая длина PBS = 24 — (GC% / 5) с минимальным и максимальным значением 8–17 нт. Все последовательности PBS 8-17nt сообщаются pegFinder для облегчения экспериментальной оптимизации.
Дизайн последовательностей олигонуклеотидов для клонирования
После выбора первичного спейсера sgRNA, вторичного спейсера sgRNA, матрицы RT и последовательности PBS, pegFinder дополнительно генерирует последовательности олигонуклеотидов, которые можно непосредственно использовать для лигирования клонирования сконструированных pegRNAs и/или sgRNA. Олигонуклеотиды, разработанные pegFinder, предназначены для лигирования путем адаптации протокола lentiGuide-Puro 14 (см. раздел ниже). Следует отметить, что последовательности pegRNA/sgRNA, которые не начинаются с буквы «G» на 5’-конце, автоматически будут иметь добавленную букву «G» в клонирующих олигонуклеотидных последовательностях для облегчения транскрипции со стандартного промотора U6.
Выходная таблица pegFinder для экспериментальной оптимизации
В то время как pegFinder автоматически рекомендует один дизайн pegRNA для указанного редактирования, pegFinder также создает загружаемую таблицу, содержащую библиотеку дизайнов pegRNA для каждого из спейсеров с самым высоким рейтингом.
/cc0076d6e2a8d21.s.siteapi.org/img/2cee63a951aece7044b09d309d0682c02d3153e7.png) Пользователь может дополнительно указать, сколько распорок-кандидатов будет включено в таблицу результатов (по умолчанию установлено 3 распорки, каждая с полным набором 3-дюймовых распорок-кандидатов). Для каждой из этих конструкций также предоставляются готовые к клонированию олигонуклеотидные последовательности, что облегчает экспериментальную оптимизацию. Если, например, последующие эксперименты покажут, что спейсер sgRNA, первоначально выбранный pegFinder, неэффективен для индукции первичного редактирования, пользователь может легко обратиться к таблице результатов pegFinder и протестировать альтернативные конструкции sgRNA/pegRNA.
Пользователь может дополнительно указать, сколько распорок-кандидатов будет включено в таблицу результатов (по умолчанию установлено 3 распорки, каждая с полным набором 3-дюймовых распорок-кандидатов). Для каждой из этих конструкций также предоставляются готовые к клонированию олигонуклеотидные последовательности, что облегчает экспериментальную оптимизацию. Если, например, последующие эксперименты покажут, что спейсер sgRNA, первоначально выбранный pegFinder, неэффективен для индукции первичного редактирования, пользователь может легко обратиться к таблице результатов pegFinder и протестировать альтернативные конструкции sgRNA/pegRNA.Выбор спейсеров sgRNA для вторичного разрыва
Разрыв противоположной цепи может повысить эффективность первичного редактирования (PE3) 2 . Для разработки «вторичных» разрывных sgRNA, которые будут использоваться для PE3, pegFinder идентифицирует спейсеры sgRNA de novo на основе выбранного фермента CRISPR. pegFinder считает sgRNA кандидатом на вторичный разрыв, если: 1) sgRNA нацелена на нить, противоположную первичной sgRNA, 2) вторичный разрыв происходит на расстоянии 40-150 нуклеотидов (диапазон по умолчанию, может быть указан пользователем) от первичного ник, 3) нет поли(Т)-трактов, которые могли бы терминировать транскрипцию U6, и, если применимо, 4) sgRNA имеет сумму «нецелевых» баллов уровня I Bin I + Bin II ≤ 1 (широкий дизайнер) или количество «посев вне цели» равен 0 (CRISPRscan).
 Затем pegFinder выбирает спейсер sgRNA, который разрывается ближе всего к ± 50 нт от первичной sgRNA с разрывом. При включении прогнозов эффективности на мишени pegFinder выбирает спейсер sgRNA с наивысшим показателем на мишени среди кандидатов на вторичные разрывы sgRNA. Все спейсеры-кандидаты вторичного разрыва sgRNA возвращаются pegFinder для простоты экспериментальной оптимизации.
Затем pegFinder выбирает спейсер sgRNA, который разрывается ближе всего к ± 50 нт от первичной sgRNA с разрывом. При включении прогнозов эффективности на мишени pegFinder выбирает спейсер sgRNA с наивысшим показателем на мишени среди кандидатов на вторичные разрывы sgRNA. Все спейсеры-кандидаты вторичного разрыва sgRNA возвращаются pegFinder для простоты экспериментальной оптимизации.Для повышения специфичности первичного редактирования Anzalone et al. описали разновидность системы PE3, в которой используются sgRNA со вторичными разрывами, которые активны только после первичного редактирования (PE3b). Когда это возможно, pegFinder также разрабатывает специфические для редактирования вторичные никеры PE3b sgRNA.
Протокол клонирования pegRNA/sgRNA
Используя олигонуклеотидные последовательности, полученные с помощью pegFinder, каждую прямую/обратную пару олигонуклеотидов отжигали в буфере для лигирования T4 (NEB) с T4 PNK для фосфорилирования олигонуклеотидов.
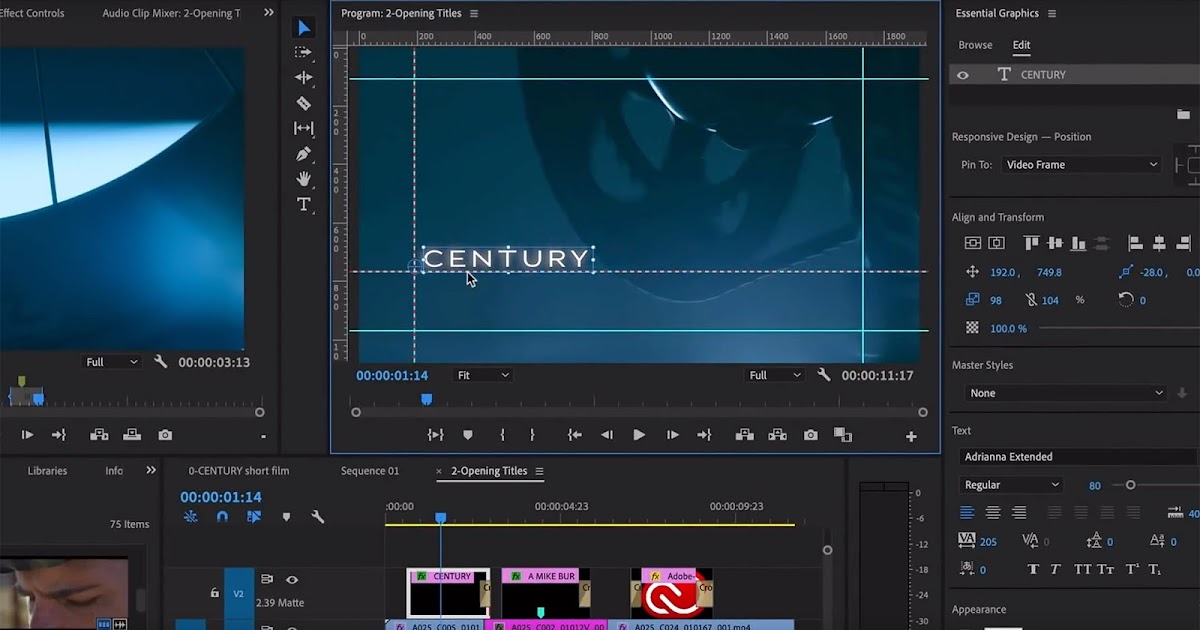 Вектор экспрессии пегРНК-реципиента (вектор pU6-pegRNA-GG-акцептор; Addgene #132777, подарок от David Liu) расщепляли с помощью BsaI (NEB). После разбавления олигодуплексов 1:100 первичную разрывную sgRNA, инвариантный каркас и 3′-удлинение лигировали вместе в расщепленный вектор с использованием Quick Ligase (NEB) для получения полной плазмиды pegRNA. Аналогичным образом, разбавленный дуплекс вторичной никсинговой sgRNA лигировали в стандартный вектор sgRNA U6 (такой как вектор lentiGuide-Puro; Addgene #529).63). Обратите внимание, что при использовании альтернативных плазмид для выполнения клонирования pegRNA/sgRNA выступы, полученные с помощью pegFinder, возможно, потребуется настроить, чтобы они соответствовали липким концам после расщепления плазмиды.
Вектор экспрессии пегРНК-реципиента (вектор pU6-pegRNA-GG-акцептор; Addgene #132777, подарок от David Liu) расщепляли с помощью BsaI (NEB). После разбавления олигодуплексов 1:100 первичную разрывную sgRNA, инвариантный каркас и 3′-удлинение лигировали вместе в расщепленный вектор с использованием Quick Ligase (NEB) для получения полной плазмиды pegRNA. Аналогичным образом, разбавленный дуплекс вторичной никсинговой sgRNA лигировали в стандартный вектор sgRNA U6 (такой как вектор lentiGuide-Puro; Addgene #529).63). Обратите внимание, что при использовании альтернативных плазмид для выполнения клонирования pegRNA/sgRNA выступы, полученные с помощью pegFinder, возможно, потребуется настроить, чтобы они соответствовали липким концам после расщепления плазмиды.Экспериментальная проверка пегРНК
Для вставки +1 СТТ в HEK3 использовалась следующая пегРНК: 5’ GGCCCAGACTGAGCACGTGAGTTTTAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTGGCCCAAGTCAAAGAGCTTGGCCCAAGTCAAAG
Для вставки +1 ТТ на HEK3 использовалась следующая пегРНК: 5’ GGCCCAGACTGAGCACGTGAGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTCTGCCATCAAGCGTGCTCAGTCTG 3’.

Олигопоследовательности, предоставленные pegFinder, затем клонировали в соответствующие векторы экспрессии, как описано выше. Экспериментальную проверку пегРНК проводили, как описано ранее, с небольшими модификациями 2 . Клетки HEK293T (ATCC) высевали на 24-луночные планшеты и через 16 часов трансфицировали при 60-80% конфлюэнтности 2 мкл липофектамина 2000 (ThermoFisher), 1,5 мкг плазмиды pCMV-PE2 (Addgene #132775), 500 нг плазмиды pegRNA (клонированные в Addgene #132777) и 200 нг вторичных разрывных плазмид sgRNA. 50 нг sfGFP-N1 (Addgene #54737) также были включены для оценки эффективности трансфекции. Клетки собирали через 2 дня после трансфекции и геномную ДНК (гДНК) очищали с помощью набора QIAamp DNA Blood Mini Kit (Qiagen). Затем геномную область, окружающую целевой сайт пегРНК, амплифицировали с помощью ПЦР со следующими праймерами, используя 200 нг исходной гДНК: Forward, AGGGAAACGCCCATGCAATTAGTCT; Реверс, CTAGCCCCTGTCTAGGAAAAGCTGTC.
ПЦР проводили с использованием полимеразы Phusion Flash High-Fidelity (ThermoFisher) со следующими параметрами: 98°C в течение 2 мин, затем 35 циклов [98°C в течение 1 с, 60°C в течение 5 с и 72°C в течение 5 с], с последующим удлинением при 72°С в течение 2 мин.
 Полученные ПЦР-ампликоны очищали в геле (Qiagen) и обрабатывали для секвенирования по Сэнгеру (анализатор ДНК Applied Biosystems 3730xL).
Полученные ПЦР-ампликоны очищали в геле (Qiagen) и обрабатывали для секвенирования по Сэнгеру (анализатор ДНК Applied Biosystems 3730xL).Сводка отчета
Дополнительная информация о дизайне исследования доступна в Сводке отчета по исследованию природы, связанной с этой статьей.
Доступность данных
Основные данные, подтверждающие результаты этого исследования, доступны в документе и его дополнительной информации. Для pegRNAs, которые были экспериментально протестированы в этом исследовании, вся соответствующая информация представлена в качестве дополнительной информации. Эта информация может быть использована для воссоздания конструкций pegRNA, описанных здесь, через веб-портал pegFinder (http://pegfinder.sidichenlab.org).
Доступность кода
Пользовательский код доступен на GitHub (https://github.com/rdchow/pegfinder). Веб-портал доступен по адресу http://pegfinder.sidichenlab.org.
1625906_SuppTable1
Нажмите здесь для просмотра.
 (8.9K, xlsx)
(8.9K, xlsx) 1625906_SuppTable2
Щелкните здесь для просмотра. (20K, xlsx)
1625906_SuppInfo
Щелкните здесь для просмотра. (770K, pdf)
Благодарим С. Эйзенбарта за поддержку. RDC поддерживается учебным грантом Yale NIH MSTP (T32GM136651) и стипендией NIH NRSA от NCI (F30CA250249). АО поддерживается учебным грантом Yale MSTP от NIH (T32GM136651) и стипендией NIH NSRA от NHLBI (F30HL149).151). SC поддерживается Yale SBI/Genetics Startup Fund, NIH/NCI/NIDA (DP2CA238295, 1R01CA231112, U54CA209992-8697, R33CA225498, RF1DA048811), DoD (W81XWH-20-1-0072 / BC1
), AACR (4
5, -01-CHEN), Институт исследования рака (CLIP), Фонд V, Фонд семьи Людвиг, Фонд Зонтаг (DSA), Фонд семьи Блаватник и Фонд семьи Шеневерт.Конкурирующие интересы
Авторы заявляют об отсутствии конкурирующих интересов. Для полного раскрытия: SC является соучредителем, получателем финансирования и научным консультантом EvolveImmune Therapeutics; компания не имеет никакого отношения к этому исследованию.
/cc0076d6e2a8d21.s.siteapi.org/img/be6245400a27d34d8bff839e3e1e8c8500c85abf.jpg)
Дополнительная информация доступна для этого документа по адресу https://doi.org/10.1038/s41551-020-00622-8.
Информация о переизданиях и разрешениях доступна на сайте www.nature.com/reprints.
Примечание издателя : Springer Nature остается нейтральной в отношении юрисдикционных претензий в опубликованных картах и институциональной принадлежности.
1. Пикар-Оливер А. и Герсбах К.А. Следующее поколение технологий и приложений CRISPR-Cas. Nat Rev Mol Cell Biol 20, 490–507 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
2. Anzalone AV и другие. Редактирование генома с помощью поиска и замены без двухцепочечных разрывов или донорской ДНК. Природа 576, 149–157 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
3. Liu Y и другие. Эффективное создание моделей мышей с помощью основной системы редактирования. Обнаружение клеток 6, 1–4 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
4.
 Lin Q
и другие.
Основное редактирование генома риса и пшеницы. Природная биотехнология
38, 582–585 (2020). [PubMed] [Академия Google]
Lin Q
и другие.
Основное редактирование генома риса и пшеницы. Природная биотехнология
38, 582–585 (2020). [PubMed] [Академия Google]5. Мейер Дж. А., Чжан Ф. и Санджана Н.Э. РУКОВОДСТВА: дизайн sgRNA для скрининга потери функции. Природные методы 14, 831–832 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
6. Doench JG и другие. Оптимизированный дизайн sgRNA для максимальной активности и минимизации нецелевых эффектов CRISPR-Cas9. Нац. Биотехнолог. 34, 184–191 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
7. Listgarten J и другие. Прогнозирование нецелевой активности для сквозного дизайна направляющих РНК CRISPR. Природная биомедицинская инженерия 2, 38 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
8. Морено-Матеос М.А. и другие. CRISPRscan: разработка высокоэффективных sgRNA для нацеливания CRISPR-Cas9 in vivo. Нац. Методы 12, 982–988 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
9. Haeussler M и другие.
 Оценка нецелевых и целевых алгоритмов оценки и интеграция в инструмент выбора направляющей РНК CRISPOR. Геномная биология
17, 148 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Оценка нецелевых и целевых алгоритмов оценки и интеграция в инструмент выбора направляющей РНК CRISPOR. Геномная биология
17, 148 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]10. Лабун К. и другие. CHOPCHOP v3: расширение веб-инструментов CRISPR за пределы редактирования генома. Нуклеиновые Кислоты Res 47, W171–W174 (2019 г.). [Бесплатная статья PMC] [PubMed] [Google Scholar]
11. Park J, Bae S & Kim J-S Cas-Designer: веб-инструмент для выбора целевых сайтов CRISPR-Cas9. Биоинформатика 31, 4014–4016 (2015). [PubMed] [Google Scholar]
12. Needleman SB & Wunsch CD Общий метод, применимый для поиска сходства в аминокислотной последовательности двух белков. Журнал молекулярной биологии 48, 443–453 (1970). [PubMed] [Google Scholar]
13. Уолтон Р.Т., Кристи К.А., Уиттакер М.Н. и Кляйнстивер Б.П. Неограниченное нацеливание на геном с помощью сконструированного CRISPR-Cas9, почти не содержащего PAMварианты. Наука 368, 290–296 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
14.
 Санджана Н.Е., Шалем О. и Чжан Ф.
Улучшенные векторы и полногеномные библиотеки для скрининга CRISPR. Нац. Методы
11, 783–784 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Санджана Н.Е., Шалем О. и Чжан Ф.
Улучшенные векторы и полногеномные библиотеки для скрининга CRISPR. Нац. Методы
11, 783–784 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]Сконструированные пегРНК повышают эффективность первичного редактирования
- Список журналов
- Рукописи авторов HHS
- PMC88
Нац биотехнолог. Авторская рукопись; доступно в PMC 2022 4 апреля. 2022 март; 40(3): 402–410.
Опубликовано в Интернете 4 октября 2021 г. doi: 10.1038/s41587-021-01039-7
PMCID: PMC8
8NIHMSID: NIHMS1729773
608 PMID: 30032, 1, 2, 3, * , 1, 2, 3, * , 1, 2, 3 , 1, 2, 3 , 1, 2, 3 , 1, 2, 3 , 1, 2, 3 , 1, 2, 3 , 1, 2, 3 , 1, 2, 3 и 1, 2, 3, ‡
. практически любую комбинацию точечных мутаций, небольших вставок или небольших делеций в ДНК живых клеток. Основная направляющая РНК для редактирования (pegRNA) направляет белок основного редактора в целевой локус, а также кодирует желаемое редактирование. Здесь мы демонстрируем, что деградация 3′-области пегРНК, которая содержит матрицу обратной транскриптазы и сайт связывания праймеров, может отравить активность основных систем редактирования, препятствуя эффективности редактирования. Мы включили мотивы структурированной РНК в 3′-конец пегРНК, которые повышают их стабильность и предотвращают деградацию 3′-удлинения. Полученные сконструированные pegRNAs (epegRNAs) повышают эффективность первичного редактирования в 3-4 раза в клетках HeLa, U2OS и K562 и в первичных фибробластах человека без увеличения активности нецелевого редактирования. Мы оптимизировали выбор 3′-структурного мотива и разработали pegLIT, вычислительный инструмент для идентификации неинтерферирующих нуклеотидных линкеров между пегРНК и 3′-мотивами.
практически любую комбинацию точечных мутаций, небольших вставок или небольших делеций в ДНК живых клеток. Основная направляющая РНК для редактирования (pegRNA) направляет белок основного редактора в целевой локус, а также кодирует желаемое редактирование. Здесь мы демонстрируем, что деградация 3′-области пегРНК, которая содержит матрицу обратной транскриптазы и сайт связывания праймеров, может отравить активность основных систем редактирования, препятствуя эффективности редактирования. Мы включили мотивы структурированной РНК в 3′-конец пегРНК, которые повышают их стабильность и предотвращают деградацию 3′-удлинения. Полученные сконструированные pegRNAs (epegRNAs) повышают эффективность первичного редактирования в 3-4 раза в клетках HeLa, U2OS и K562 и в первичных фибробластах человека без увеличения активности нецелевого редактирования. Мы оптимизировали выбор 3′-структурного мотива и разработали pegLIT, вычислительный инструмент для идентификации неинтерферирующих нуклеотидных линкеров между пегРНК и 3′-мотивами. Наконец, мы продемонстрировали, что epegRNAs повышают эффективность установки или коррекции мутаций, связанных с заболеванием.
Наконец, мы продемонстрировали, что epegRNAs повышают эффективность установки или коррекции мутаций, связанных с заболеванием.Nelson Randolph Сводка отчета по рукописи пересмотрена.pdf
Способность вносить целенаправленные изменения в геном живых систем продолжает совершенствовать науки о жизни и медицину. Стратегии редактирования ДНК, опосредованные двухцепочечным разрывом (DSB), в которых используются программируемые нуклеазы, такие как нуклеазы ZFN, TALEN или CRISPR-Cas, могут эффективно разрушать гены, вызывая вставки или делеции (indels) в целевом сайте, но DSB также приводят к результатам. которые часто нежелательны, включая неконтролируемое смешение результатов редактирования 1,2 , более крупные перестройки ДНК 3–5 , активация p53 6–8 и хромотрипсис 9,10 . Хотя таргетные DSB могут стимулировать точную коррекцию генов посредством репарации, направленной на гомологию, этот процесс неэффективен в большинстве терапевтически релевантных типов клеток -11.
 Напротив, базовые редакторы 12,13 и первичные редакторы 14 могут эффективно вносить точные изменения в терапевтически значимые клетки, не требуя DSB. Редакторы оснований цитозина и аденозина позволяют преобразовывать C•G в T•A и A•T в G•C соответственно, в то время как первичные редакторы позволяют устанавливать практически любую локальную мутацию, включая замену, вставку и/или делецию. до десятков пар оснований на целевых участках ДНК.
Напротив, базовые редакторы 12,13 и первичные редакторы 14 могут эффективно вносить точные изменения в терапевтически значимые клетки, не требуя DSB. Редакторы оснований цитозина и аденозина позволяют преобразовывать C•G в T•A и A•T в G•C соответственно, в то время как первичные редакторы позволяют устанавливать практически любую локальную мутацию, включая замену, вставку и/или делецию. до десятков пар оснований на целевых участках ДНК.Системы первичного редактирования (PE) как минимум состоят из двух компонентов: белка, содержащего программируемую ДНК-никазу, слитую со сконструированной обратной транскриптазой (RT), и направляющей РНК первичного редактирования, или pegRNA () 14 . ПегРНК содержит спейсер, который указывает целевой сайт, каркас sgRNA и 3′-удлинение, которое кодирует желаемое редактирование. Это удлинение содержит сайт связывания праймеров (PBS), который комплементарен части протоспейсера ДНК, и матрицу RT, которая кодирует желаемое редактирование и нижестоящую геномную последовательность.
 После того, как рибонуклеопротеин PE (RNP) связывается с целевым сайтом и разрывает цепь ДНК, содержащую PAM, образующаяся в результате разрыв цепи ДНК соединяется с PBS в pegRNA, запуская обратную транскрипцию матрицы RT непосредственно в целевой участок ДНК 9.0235 14 . Затем вновь синтезированный 3′-лоскут отредактированной ДНК разрешается путями репарации клеточной ДНК, что приводит к установке желаемого редактирования в целевом сайте.
После того, как рибонуклеопротеин PE (RNP) связывается с целевым сайтом и разрывает цепь ДНК, содержащую PAM, образующаяся в результате разрыв цепи ДНК соединяется с PBS в pegRNA, запуская обратную транскрипцию матрицы RT непосредственно в целевой участок ДНК 9.0235 14 . Затем вновь синтезированный 3′-лоскут отредактированной ДНК разрешается путями репарации клеточной ДНК, что приводит к установке желаемого редактирования в целевом сайте.Открыть в отдельном окне
Укороченные пегРНК ограничивают эффективность первичного редактирования.
( a ) (слева) Схема комплекса первичного редактирования, состоящего из белка первичного редактора (PE), который состоит из никазы Cas9 (nCas9), слитой с модифицированной обратной транскриптазой посредством гибкого линкера и направляющей первичного редактирования. РНК (пэгРНК). (справа) Деградация 3′-удлинения пегРНК экзонуклеазами может снизить эффективность редактирования из-за потери PBS.
 ( b ) Эффективность редактирования, опосредованная PE3, с добавлением плазмид, экспрессирующих sgRNA, укороченных pegRNA, нацеленных на один и тот же геномный локус ( HEK3 ), нецелевых pegRNA или пегРНК SaCas9. Все пегРНК экспрессируются с промотора U6. Планки данных и погрешностей отражают среднее значение и стандартное отклонение трех независимых биологических повторов. ( c ) Дизайн сконструированных пегРНК (эпегРНК), которые содержат псевдоузел структурированной РНК, защищающий 3′-удлинение от деградации экзонуклеазами.
( b ) Эффективность редактирования, опосредованная PE3, с добавлением плазмид, экспрессирующих sgRNA, укороченных pegRNA, нацеленных на один и тот же геномный локус ( HEK3 ), нецелевых pegRNA или пегРНК SaCas9. Все пегРНК экспрессируются с промотора U6. Планки данных и погрешностей отражают среднее значение и стандартное отклонение трех независимых биологических повторов. ( c ) Дизайн сконструированных пегРНК (эпегРНК), которые содержат псевдоузел структурированной РНК, защищающий 3′-удлинение от деградации экзонуклеазами.Универсальность первичного редактирования обусловлена способностью 3′-удлинения пегРНК кодировать широкий спектр редактируемых последовательностей. Несмотря на свою универсальность, эффективность современных основных редакторов существенно различается в зависимости от целевых сайтов и типов ячеек 14 . В этом исследовании мы сообщаем, что предполагаемая деградация 3′-удлинения pegRNAs может снизить эффективность первичного редактирования.
 Хотя полученные в результате укороченные pegRNAs конкурируют за вовлечение сайта-мишени, они неспособны к первичному редактированию. Чтобы устранить эту уязвимость, мы определили мотивы РНК, которые защищают целостность pegRNA и в целом улучшают эффективность основного редактирования в различных целевых сайтах в нескольких клеточных линиях и с помощью нескольких способов доставки. Полученные сконструированные pegRNAs (epegRNAs) существенно повышают эффективность и расширяют область применения первичного редактирования.
Хотя полученные в результате укороченные pegRNAs конкурируют за вовлечение сайта-мишени, они неспособны к первичному редактированию. Чтобы устранить эту уязвимость, мы определили мотивы РНК, которые защищают целостность pegRNA и в целом улучшают эффективность основного редактирования в различных целевых сайтах в нескольких клеточных линиях и с помощью нескольких способов доставки. Полученные сконструированные pegRNAs (epegRNAs) существенно повышают эффективность и расширяют область применения первичного редактирования.Стабильность РНК ограничивает эффективность пегРНК
Незащищенные ядерные РНК подвержены деградации как с 5′-, так и с 3′-концов экзонуклеазами 15 . В отличие от sgRNAs, в которых вся направляющая РНК защищена ассоциированным белком Cas9 16 , 3′-удлинение pegRNAs, вероятно, экспонируется в клетках и, таким образом, более восприимчиво к экзонуклеолитической деградации. Мы предположили, что хотя частично деградированные пегРНК могут сохранять свою способность связывать Cas9.
 и задействовать сайт-мишень ДНК, потеря или укорочение PBS может помешать их способности установить желаемое редактирование, тем самым занимая белки PE и сайты-мишени направляющими РНК, которые не могут опосредовать первичное редактирование.
и задействовать сайт-мишень ДНК, потеря или укорочение PBS может помешать их способности установить желаемое редактирование, тем самым занимая белки PE и сайты-мишени направляющими РНК, которые не могут опосредовать первичное редактирование.Чтобы проверить эту гипотезу, мы трансфицировали клетки HEK293T смесями двух плазмид в различных соотношениях, которые генерируют либо полноразмерную пегРНК, содержащую матрицу RT, кодирующую трансверсию T•A-to-A•T, либо укороченную пегРНК, содержащую Матрица RT, кодирующая трансверсию T•A-G•C, но лишенная PBS на 3′-конце. Две пегРНК нацелены либо на одни и те же, либо на разные геномные локусы в клетках человека. Мы также проверили эффект добавления плазмиды, которая генерировала невзаимодействующий SaCas9.pegRNA, которая должна конкурировать за транскрипцию с плазмидами, кодирующими pegRNA SpCas9, но не взаимодействовать с белком первичного редактора. Увеличение продукции укороченной pegRNA приводило к ингибированию активности PE, когда полноразмерная и укороченная pegRNA были нацелены на один и тот же сайт (12).
 Напротив, ни укороченная pegRNA, нацеленная на другой геномный сайт, ни ненаправленная SpCas9 sgRNA не препятствуют активности PE больше, чем pegRNA SaCas9 (10). Эти данные свидетельствуют о том, что деградированные pegRNAs с укороченными 3′-удлинениями ингибируют активность PE, позволяя некомпетентным к редактированию рибонуклеопротеинам первичного редактора (RNP) конкурировать за целевой геномный локус.
Напротив, ни укороченная pegRNA, нацеленная на другой геномный сайт, ни ненаправленная SpCas9 sgRNA не препятствуют активности PE больше, чем pegRNA SaCas9 (10). Эти данные свидетельствуют о том, что деградированные pegRNAs с укороченными 3′-удлинениями ингибируют активность PE, позволяя некомпетентным к редактированию рибонуклеопротеинам первичного редактора (RNP) конкурировать за целевой геномный локус.Дизайн сконструированных pegRNAs (epegRNAs), повышающих эффективность первичного редактирования
Определив укороченные pegRNAs как мощный ингибитор первичного редактирования, мы затем попытались свести к минимуму деградацию pegRNA. Мы предположили, что структурированные РНК-мотивы на 3′-конце пегРНК () могут улучшить стабильность пегРНК, что согласуется со способностью структур РНК на 5′- или 3′-концах повышать стабильность мРНК в клетках человека и дрожжей 17,18 . Например, длинная некодирующая РНК MALAT1 стабилизирована тройной спиралью, которая изолирует ее поли(А)-хвост, ограничивая как деградацию, так и ядерный экспорт 19 .

Сначала мы проверили, можно ли повысить эффективность первичного редактирования путем включения одного из двух стабильных псевдоузлов на 3′-конце пегРНК: либо модифицированного прекеозина 1 -1 рибопереключающего аптамера 20,21 , (evopreQ 1 ) или псевдоузел со сдвигом рамки от вируса мышиного лейкоза Молони (MMLV) 22 , далее именуемый «mpknot» (дополнительная рис. 1). Мы выбрали evopreQ 1 , потому что это один из самых маленьких природных структурных мотивов РНК с определенной третичной структурой (длиной 42 нуклеотида, н.) 20,21 . Мы пришли к выводу, что меньшие мотивы минимизируют образование вторичных структур, которые могут мешать функции pegRNA. Кроме того, более короткие pegRNAs могут быть легче получены путем химического синтеза. Мы выбрали mpknot из-за его третичной структуры и потому, что это эндогенный шаблон для MMLV RT, на основе которого был создан RT в канонических основных редакторах, что повышает вероятность того, что mpknot может помочь рекрутировать RT.
 Мы проверили, могут ли эти epegRNAs вставлять последовательность метки эпитопа FLAG с использованием PE3 в пяти геномных локусах в HEK29.3Т-клетки (). Чтобы уменьшить вероятность того, что мотив будет мешать функции пегРНК во время первичного редактирования, мы включили 8-нуклеотидный линкер для соединения либо evopreQ 1 , либо mpknot с 3′-концом PBS epegRNA. Последовательности линкера были сконструированы с использованием ViennaRNA 23 , чтобы избежать потенциальных взаимодействий спаривания оснований между линкером и PBS или между линкером и спейсером пегРНК 14 . Мы наблюдали в среднем 2,1-кратное повышение эффективности вставки метки FLAG при использовании epegRNAs по сравнению с каноническими pegRNAs во всех пяти протестированных геномных сайтах без видимых изменений в соотношении edit:indel (дополнительная рис. 2), предполагая, что 3′-концевой псевдоузел мотивы могут улучшить эффективность PE.
Мы проверили, могут ли эти epegRNAs вставлять последовательность метки эпитопа FLAG с использованием PE3 в пяти геномных локусах в HEK29.3Т-клетки (). Чтобы уменьшить вероятность того, что мотив будет мешать функции пегРНК во время первичного редактирования, мы включили 8-нуклеотидный линкер для соединения либо evopreQ 1 , либо mpknot с 3′-концом PBS epegRNA. Последовательности линкера были сконструированы с использованием ViennaRNA 23 , чтобы избежать потенциальных взаимодействий спаривания оснований между линкером и PBS или между линкером и спейсером пегРНК 14 . Мы наблюдали в среднем 2,1-кратное повышение эффективности вставки метки FLAG при использовании epegRNAs по сравнению с каноническими pegRNAs во всех пяти протестированных геномных сайтах без видимых изменений в соотношении edit:indel (дополнительная рис. 2), предполагая, что 3′-концевой псевдоузел мотивы могут улучшить эффективность PE.Открыть в отдельном окне
Эффективность редактирования PE повышается за счет добавления мотивов структурированной РНК к 3′-концу пегРНК.
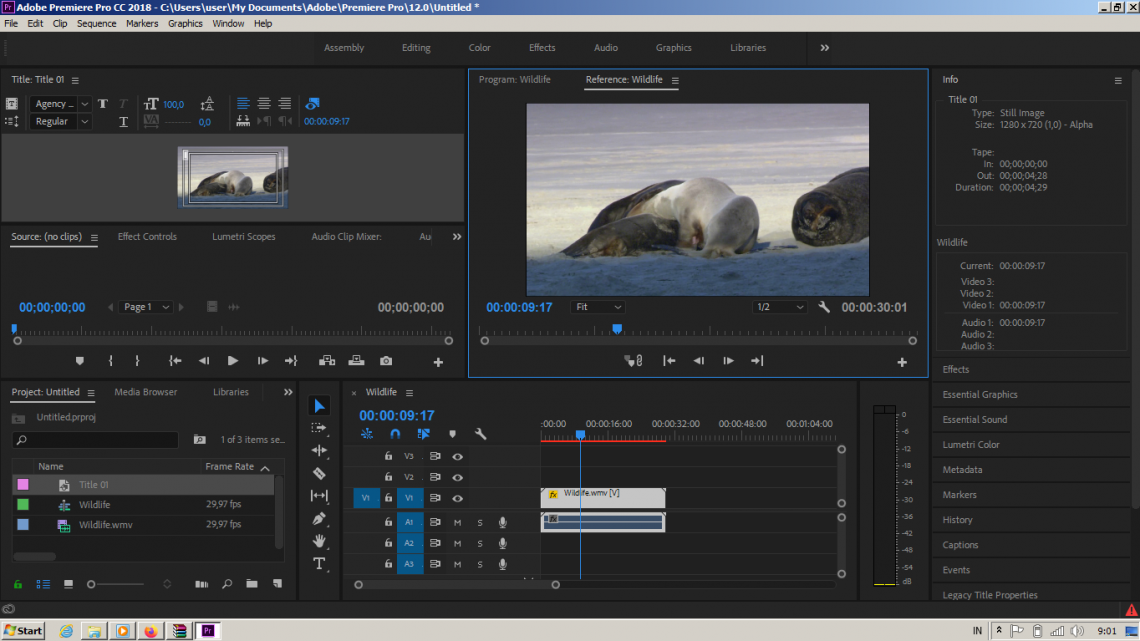
( a ) Эффективность PE3-опосредованных вставок эпитопной метки FLAG в позиции редактирования +1 (вставка непосредственно в pegRNA-индуцированный nick-сайт) во множестве геномных локусов в клетках HEK293T с использованием канонических pegRNAs («немодифицированные» ), pegRNAs либо с evopreQ 1 , либо с mpknot, присоединенными к 3′-концу PBS через 8-нуклеотидный линкер, или pegRNAs, присоединенные только с 8-nt линкерной последовательностью. ( b ) Резюме кратности изменения эффективности редактирования PE по сравнению с каноническими пегРНК указанного редактирования в различных геномных локусах при добавлении указанного 3′-мотива через 8-нуклеотидный линкер или при добавлении только линкера. «Трансверсия» означает мутацию +5 G•C в T•A по RUNX1 , EMX1 , VEGFA и DNMT1 , +1 C•G по T•A по RNF2 , и от +1 T•A до A•T по адресу HEK3 , где положительное целое число указывает расстояние от Cas9сайт ник.
 «Делегация» означает делецию 15 п.н. в ник-сайте Cas9. Обобщенные здесь данные представлены в (c) и на дополнительном рисунке 2. Горизонтальные полосы показывают средние значения. ( c ) Репрезентативное улучшение эффективности редактирования PE за счет добавления evopreQ 1 (p) или mpknot (m) через 8-нуклеотидный линкер к пегРНК с различной длиной матрицы (в нуклеотидах, указано). ( d ) Редактирование активности канонических пегРНК и модифицированных пегРНК в трех геномных локусах в клетках HeLa, клетках U2OS и клетках K562. Данные и планки погрешностей в a, c и d отражают среднее значение и стандартное отклонение трех независимых биологических повторов.
«Делегация» означает делецию 15 п.н. в ник-сайте Cas9. Обобщенные здесь данные представлены в (c) и на дополнительном рисунке 2. Горизонтальные полосы показывают средние значения. ( c ) Репрезентативное улучшение эффективности редактирования PE за счет добавления evopreQ 1 (p) или mpknot (m) через 8-нуклеотидный линкер к пегРНК с различной длиной матрицы (в нуклеотидах, указано). ( d ) Редактирование активности канонических пегРНК и модифицированных пегРНК в трех геномных локусах в клетках HeLa, клетках U2OS и клетках K562. Данные и планки погрешностей в a, c и d отражают среднее значение и стандартное отклонение трех независимых биологических повторов.Мы охарактеризовали необходимость линкерной последовательности, сравнив способность эпигРНК с 8-нуклеотидными линкерами или без них опосредовать трансверсии или вставки метки FLAG. Мы наблюдали значительное снижение эффективности редактирования PE3 при удалении линкера для epegRNAs, содержащих mpknot (p = 0,022), но без существенной разницы для epegRNAs, содержащих evopreQ 1 (дополнительная рис.
 3), возможно, потому, что evopreQ 1 меньше чем mpknot, и менее склонен к стерическим столкновениям с RT. В то время как общая средняя эффективность редактирования epegRNAs с помощью evopreQ 1 были похожи (с линкером или без него), мы отмечали случайное снижение производительности для epegRNAs без линкера (дополнительная рис. 3). Поэтому мы решили включить 8-нуклеотидный линкер, если не указано иное для последующих конструкций epegRNA.
3), возможно, потому, что evopreQ 1 меньше чем mpknot, и менее склонен к стерическим столкновениям с RT. В то время как общая средняя эффективность редактирования epegRNAs с помощью evopreQ 1 были похожи (с линкером или без него), мы отмечали случайное снижение производительности для epegRNAs без линкера (дополнительная рис. 3). Поэтому мы решили включить 8-нуклеотидный линкер, если не указано иное для последующих конструкций epegRNA.Чтобы гарантировать, что это улучшение эффективности PE не ограничивается epegRNAs с более длинными удлинениями, мы протестировали 148 дополнительных epegRNAs, кодирующих различные точечные мутации или делеции с различной длиной матрицы RT в семи различных геномных сайтах в HEK29.3T-клетки с использованием PE3. Использование любого мотива привело к среднему 1,5-кратному повышению эффективности первичного редактирования по сравнению с эффективностью канонических пегРНК во всех протестированных сайтах и пегРНК в клетках HEK293T без явного изменения соотношения редактирования:индел (- и дополнительные рисунки 4 и 5).
 ). Вместе эти результаты показывают, что epegRNAs в целом улучшают эффективность PE в клетках HEK293T.
). Вместе эти результаты показывают, что epegRNAs в целом улучшают эффективность PE в клетках HEK293T.Сконструированные пегРНК улучшают первичное редактирование в нескольких клеточных линиях млекопитающих
Ранее мы наблюдали, что эффективность PE существенно различается между типами клеток млекопитающих 14 , подчеркивая необходимость тестирования улучшенных систем PE в различных камерах. Мы проверили способность epegRNAs, содержащих мотив 3′ evopreQ 1 или mpknot, вставлять эпитопную метку FLAG из 24 п.н. по адресу HEK3 , удалять 15 п.н. Т-трансверсия RNF2 через PE3 в клетках K562, U2OS и HeLa. В каждой из этих клеточных линий epegRNAs привели к значительному повышению эффективности редактирования по сравнению с pegRNAs, в среднем в 2,4 раза выше редактирование в клетках K562, в 3,1 раза выше редактирование в клетках HeLa и в 5,6 раза выше редактирование в клетках U2OS во всех протестированных клетках. редактирования () без уменьшения отношения редактирования: вставки (дополнительный рис.
 2). Эти результаты показывают, что epegRNAs могут усиливать первичное редактирование во многих клеточных линиях млекопитающих. Кроме того, epegRNAs в большей степени улучшали эффективность редактирования в не-HEK29.3T, чем в клетках HEK293T (и дополнительный рисунок 4 по сравнению с), предполагая, что epegRNAs особенно полезны в клеточных линиях, которые менее эффективно трансфицируются или редактируются исходными системами PE.
2). Эти результаты показывают, что epegRNAs могут усиливать первичное редактирование во многих клеточных линиях млекопитающих. Кроме того, epegRNAs в большей степени улучшали эффективность редактирования в не-HEK29.3T, чем в клетках HEK293T (и дополнительный рисунок 4 по сравнению с), предполагая, что epegRNAs особенно полезны в клеточных линиях, которые менее эффективно трансфицируются или редактируются исходными системами PE.Влияние сконструированных пегРНК на нецелевое основное редактирование
Предыдущие исследования показали, что основное редактирование приводит к значительно меньшему нецелевому редактированию, чем другие стратегии редактирования генов CRISPR 14,24–27 . Чтобы определить, будет ли добавление evopreQ 1 или mpknot изменили степень нецелевого редактирования, мы обрабатывали клетки HEK293T пегРНК или эпегРНК, нацеленными на HEK3 , EMX1 или FANCF , которые шаблонируют либо трансверсию (T•A-to-A•T по адресу HEK3 или G•C-to-T•A по адресу EMX1 и FANCF ) или удаление 15 п.
 н. с использованием PE3. Мы измерили степень образования инделей и любых нуклеотидных изменений, которые могли бы возникнуть в результате первичного редактирования в четырех верхних экспериментально подтвержденных нецелевых сайтах 9.0235 28 для каждого целевого локуса и сравнили степень нецелевого редактирования между эпигРНК и немодифицированными пегРНК после обработки PE3. Во всех случаях epegRNAs и pegRNAs демонстрировали ≤0,1% нецелевого первичного редактирования и вставок в исследованных сайтах (дополнительная рис. 6), что позволяет предположить, что epegRNAs и pegRNAs демонстрируют одинаковые уровни нецелевого редактирования.
н. с использованием PE3. Мы измерили степень образования инделей и любых нуклеотидных изменений, которые могли бы возникнуть в результате первичного редактирования в четырех верхних экспериментально подтвержденных нецелевых сайтах 9.0235 28 для каждого целевого локуса и сравнили степень нецелевого редактирования между эпигРНК и немодифицированными пегРНК после обработки PE3. Во всех случаях epegRNAs и pegRNAs демонстрировали ≤0,1% нецелевого первичного редактирования и вставок в исследованных сайтах (дополнительная рис. 6), что позволяет предположить, что epegRNAs и pegRNAs демонстрируют одинаковые уровни нецелевого редактирования.Основа улучшенного первичного редактирования с помощью сконструированных пегРНК
ЭпегРНК могут улучшать результаты первичного редактирования с помощью различных механизмов, включая устойчивость к деградации, более высокие уровни экспрессии, более эффективный Cas9связывание и/или вовлечение ДНК-мишени в комплексе с Cas9; мы исследовали каждую из этих возможностей.
Чтобы определить, препятствуют ли evopreQ 1 или mpknot деградации 3′-участка пегРНК, мы сравнили стабильность эпегРНК и пегРНК после инкубации in vitro с ядерными лизатами HEK293T, содержащими эндогенные экзонуклеазы. Мы обнаружили, что пегРНК деградировали в большей степени при таком лечении по сравнению с эпегРНК (в 1,9 раза по сравнению с evopreQ 9).0329 1 и в 1,8 раза по сравнению с mpknot, p<0,005, ). Наоборот, добавление Cas9, который связывает каркас направляющей РНК и, вероятно, защищает основную sgRNA от деградации, восстанавливает количество pegRNA по сравнению с любой из epegRNA, что определяется количественной оценкой RT-qPCR каркаса направляющей РНК (10).
Открыть в отдельном окне
Структурные мотивы повышают стабильность РНК и эффективность обратной транскрипции, но снижают аффинность связывания Cas9.
( a ) Устойчивость немодифицированной пегРНК или эпиРНК, содержащих evopreQ 1 или mpknot, к деградации при воздействии ядерных лизатов HEK293T.
 Показанный агарозный гель является репрезентативным для трех экспериментов. Необработанные транскрибированные in vitro pegRNAs или epegRNAs служили в качестве стандартов. Процент оставшейся РНК рассчитывали с помощью денситометрии. Значимость анализировали с использованием двустороннего непарного критерия Стьюдента (p=0,0028 для mpknot и 0,0022 для evopreQ 1 ). ( b ) Кратность изменения количества каркаса пегРНК по сравнению с немодифицированной пегРНК при воздействии ядерных лизатов HEK293T в отсутствие и в присутствии nCas9, как определено с помощью RT-qPCR каркаса sgRNA. ( c ) Сравнение промежуточных продуктов первичного редактирования, генерируемых PE2, с пегРНК или эпегРНК в RNF2 . Пунктирные линии указывают на полноразмерный продукт обратной транскриптазы, матрицированный пегРНК или эпегРНК, тестируемой в указанном локусе. Ось X относится к положению PE2-индуцированного разрыва с первым основанием 3′ ниже по течению, представленным как положение +1.
Показанный агарозный гель является репрезентативным для трех экспериментов. Необработанные транскрибированные in vitro pegRNAs или epegRNAs служили в качестве стандартов. Процент оставшейся РНК рассчитывали с помощью денситометрии. Значимость анализировали с использованием двустороннего непарного критерия Стьюдента (p=0,0028 для mpknot и 0,0022 для evopreQ 1 ). ( b ) Кратность изменения количества каркаса пегРНК по сравнению с немодифицированной пегРНК при воздействии ядерных лизатов HEK293T в отсутствие и в присутствии nCas9, как определено с помощью RT-qPCR каркаса sgRNA. ( c ) Сравнение промежуточных продуктов первичного редактирования, генерируемых PE2, с пегРНК или эпегРНК в RNF2 . Пунктирные линии указывают на полноразмерный продукт обратной транскриптазы, матрицированный пегРНК или эпегРНК, тестируемой в указанном локусе. Ось X относится к положению PE2-индуцированного разрыва с первым основанием 3′ ниже по течению, представленным как положение +1. Гистограммы и круговые диаграммы генерируются из среднего значения трех независимых биологических повторов. ( d ) Эффективность редактирования PE3 в клетках HEK293T с использованием немодифицированных пегРНК, пегРНК, содержащих мотив evopreQ1, или пегРНК, содержащих точечный мутант G15C evopreQ 1 (M1), который разрушает структуру мотива псевдоузла. ( e ) Фракция RNP Cas9, состоящая из dCas9 и либо немодифицированной пегРНК, либо эпегРНК, содержащая либо evopreQ 1 , либо mpknot и имеющая шаблонную вставку метки +1 FLAG в HEK3 , связанную с двухцепочечной ДНК, как определено с помощью MST. ( f ) Активация транскрипции CRISPRa с помощью pegRNAs, epegRNAs и sgRNAs. Сообщаемая флуоресценция GFP нормализована по флуоресценции iRFP, экспрессируемой совместно трансфицированной плазмидой. AU, условные единицы. ( г ) Фракция немодифицированной пегРНК или эпегРНК (шаблон +1 FLAG вставки в HEK3 ), содержащая либо evopreQ 1 , либо mpk, не связанную с H840A nCas9, как определено с помощью микромасштабного термофореза (MST).
Гистограммы и круговые диаграммы генерируются из среднего значения трех независимых биологических повторов. ( d ) Эффективность редактирования PE3 в клетках HEK293T с использованием немодифицированных пегРНК, пегРНК, содержащих мотив evopreQ1, или пегРНК, содержащих точечный мутант G15C evopreQ 1 (M1), который разрушает структуру мотива псевдоузла. ( e ) Фракция RNP Cas9, состоящая из dCas9 и либо немодифицированной пегРНК, либо эпегРНК, содержащая либо evopreQ 1 , либо mpknot и имеющая шаблонную вставку метки +1 FLAG в HEK3 , связанную с двухцепочечной ДНК, как определено с помощью MST. ( f ) Активация транскрипции CRISPRa с помощью pegRNAs, epegRNAs и sgRNAs. Сообщаемая флуоресценция GFP нормализована по флуоресценции iRFP, экспрессируемой совместно трансфицированной плазмидой. AU, условные единицы. ( г ) Фракция немодифицированной пегРНК или эпегРНК (шаблон +1 FLAG вставки в HEK3 ), содержащая либо evopreQ 1 , либо mpk, не связанную с H840A nCas9, как определено с помощью микромасштабного термофореза (MST). Планки данных и погрешностей отражают среднее значение и стандартное отклонение трех независимых биологических повторов.
Планки данных и погрешностей отражают среднее значение и стандартное отклонение трех независимых биологических повторов.Способность 3′-структурных мотивов увеличивать обилие вышележащей каркасной области () указывает на то, что в деградации pegRNA в ядре преобладает 3′-направленная деградация. Эта модель согласуется с характерным поведением ядерной экзосомы, основного источника оборота РНК в ядре 29 . Однако частично деградированные pegRNAs будут генерировать некомпетентные к редактированию RNP, которые, как ранее было показано, ингибируют прайм-редактирование (4). Чтобы обнаружить частично деградировавшие РНК в клетках, мы проанализировали лизаты клеток HEK293T, трансфицированных плазмидами, кодирующими PE2, и либо пегРНК, либо эпегРНК, матрицирующих либо вставку тега +1 FLAG в HEK3 , либо трансверсию нуклеотидов в EMX1 с помощью нозерн-блоттинга. Мы наблюдали виды РНК, содержащие каркас sgRNA и эквивалентные по размеру sgRNA, что согласуется с нашим предыдущим выводом (1), что Cas9связывание защищает каркас от 3′-направленной деградации (дополнительная рис.
 7). Однако лизаты с разными общими уровнями pegRNA или epegRNA имели аналогичные уровни sgRNA-подобных укороченных видов, которые представляли лишь меньшую часть содержания направляющей РНК в лизате (дополнительная рис. 7). Поскольку мы наблюдали сильную деградацию пегРНК, подвергшихся воздействию ядерного лизата in vitro (и ), а пегРНК присутствует на уровнях выше, чем PE2 в клетках HEK293T (), мы подозреваем, что частично деградированные виды пегРНК не накапливаются на уровнях, поддающихся нозерн-блоттингу. обнаружение.
7). Однако лизаты с разными общими уровнями pegRNA или epegRNA имели аналогичные уровни sgRNA-подобных укороченных видов, которые представляли лишь меньшую часть содержания направляющей РНК в лизате (дополнительная рис. 7). Поскольку мы наблюдали сильную деградацию пегРНК, подвергшихся воздействию ядерного лизата in vitro (и ), а пегРНК присутствует на уровнях выше, чем PE2 в клетках HEK293T (), мы подозреваем, что частично деградированные виды пегРНК не накапливаются на уровнях, поддающихся нозерн-блоттингу. обнаружение.Затем мы исследовали промежуточные продукты редактирования геномных праймов, чтобы лучше понять, как epegRNAs могут обеспечивать повышение эффективности редактирования. В нашей текущей модели 3′-промежуточный лоскут, генерируемый RT-удлинением сайта-мишени с разрывом, превращается в 5′-промежуточный лоскут, заменяя исходную геномную последовательность на вновь синтезированную 14 . Затем этот 5′-лоскут удаляется экзонуклеазами 5′-3′, и полученный геномный разрыв подвергается лигированию для установки первичного редактирования 9.
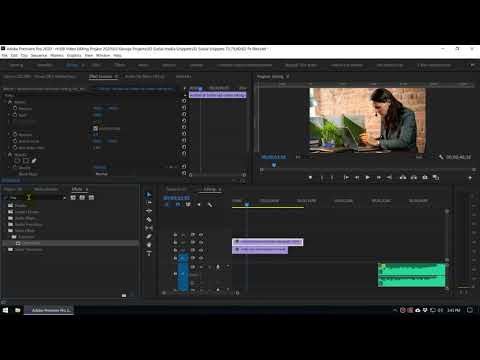 0235 14 . В то время как полноразмерные пегРНК, как ожидается, эффективно шаблонируют удлинение ОТ с разрывом геномной цепи, укороченные пегРНК без PBS должны быть неспособны сделать это, вместо этого приводя к надрезу целевой цепи с последующим пережевыванием или удлинением цепи с помощью Ферменты репарации ДНК (в любом случае без шаблонного редактирования). Если с epegRNAs наблюдается большая доля RT-удлиненных промежуточных соединений первичного редактирования, чем с pegRNAs, это может свидетельствовать о том, что добавление мотивов 3′ РНК улучшает целостность PBS.
0235 14 . В то время как полноразмерные пегРНК, как ожидается, эффективно шаблонируют удлинение ОТ с разрывом геномной цепи, укороченные пегРНК без PBS должны быть неспособны сделать это, вместо этого приводя к надрезу целевой цепи с последующим пережевыванием или удлинением цепи с помощью Ферменты репарации ДНК (в любом случае без шаблонного редактирования). Если с epegRNAs наблюдается большая доля RT-удлиненных промежуточных соединений первичного редактирования, чем с pegRNAs, это может свидетельствовать о том, что добавление мотивов 3′ РНК улучшает целостность PBS.To capture these intermediates, we transfected HEK293T cells with plasmids encoding PE2 and either unmodified pegRNAs or epegRNAs containing evopreQ 1 or mpknot that template transversions at HEK3 , DNMT1 , EMX1 , or RNF2 . Затем мы использовали терминальную трансферазу, чтобы пометить олиго-dG 3′-концы геномной ДНК, которые должны включать промежуточные продукты первичного редактирования, которые еще не подверглись лигированию.
 В каждом случае epegRNAs уменьшали количество некомпетентных к редактированию промежуточных продуктов в целевом сайте в среднем в 2,2 раза по четырем сайтам (и дополнительная рис. 8). Доминантный продукт обратной транскрипции содержал полную последовательность, матрицированную 3′-удлинением, и два нуклеотида, матрицированные двумя последними нуклеотидами каркаса пегРНК, что согласуется с предыдущими in vitro характеристика промежуточных продуктов ПЭ 14 . Нуклеотиды шаблонного каркаса предположительно удаляются во время репарации ДНК локуса-мишени для получения чисто отредактированных аллелей, которые представляют собой доминантный продукт PE. Эти данные согласуются с моделью, в которой epegRNAs улучшают обратную транскрипцию удлинения pegRNA в сайт-мишень за счет снижения частоты непродуктивного разрыва сайта-мишени от основных редакторов, связанных с укороченными pegRNAs.
В каждом случае epegRNAs уменьшали количество некомпетентных к редактированию промежуточных продуктов в целевом сайте в среднем в 2,2 раза по четырем сайтам (и дополнительная рис. 8). Доминантный продукт обратной транскрипции содержал полную последовательность, матрицированную 3′-удлинением, и два нуклеотида, матрицированные двумя последними нуклеотидами каркаса пегРНК, что согласуется с предыдущими in vitro характеристика промежуточных продуктов ПЭ 14 . Нуклеотиды шаблонного каркаса предположительно удаляются во время репарации ДНК локуса-мишени для получения чисто отредактированных аллелей, которые представляют собой доминантный продукт PE. Эти данные согласуются с моделью, в которой epegRNAs улучшают обратную транскрипцию удлинения pegRNA в сайт-мишень за счет снижения частоты непродуктивного разрыва сайта-мишени от основных редакторов, связанных с укороченными pegRNAs.Поскольку одноцепочечные 3′-концы являются общей чертой субстратов 3′-экзонуклеаз 30 , мы затем проверили, можно ли объяснить устойчивость к деградации, придаваемую этими мотивами, более механически стабильными третичными структурами псевдоузлов.
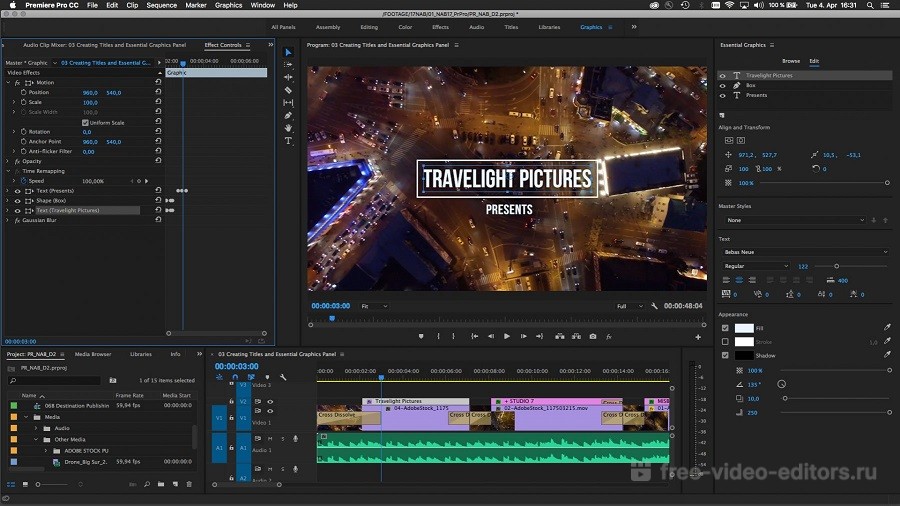 Примечательно, что добавление шпилек из 15 п.н. (34 нуклеотида) к 3′-концу привело к непоследовательному повышению эффективности PE по сравнению с добавлением псевдоузлов (дополнительная рис. 9), что позволяет предположить, что третичная структура действительно является важной особенностью epegRNAs.
Примечательно, что добавление шпилек из 15 п.н. (34 нуклеотида) к 3′-концу привело к непоследовательному повышению эффективности PE по сравнению с добавлением псевдоузлов (дополнительная рис. 9), что позволяет предположить, что третичная структура действительно является важной особенностью epegRNAs.Чтобы проверить, требуется ли третичная структура псевдоузла для опосредованного epegRNA улучшения эффективности PE, мы исследовали эффективность редактирования epegRNAs, содержащих точечную мутацию G15C в evopreQ 1 , мутацию, которая, как известно, нарушает формирование псевдоузла (M1 на дополнительной рис. 1). ) 21 . Мы использовали epegRNAs для установки вставки метки эпитопа FLAG длиной 24 п.н., делеции длиной 15 п.н. или трансверсий в HEK3 или RNF2 в клетках HEK293T с использованием PE3. Действительно, включение мутации G15C в evopreQ 1 отменил повышение эффективности редактирования (). Эти результаты показывают, что вторичная или третичная структура мотивов имеет решающее значение для опосредованных epegRNA улучшений PE, вероятно, за счет стабилизации 3′-удлинения.
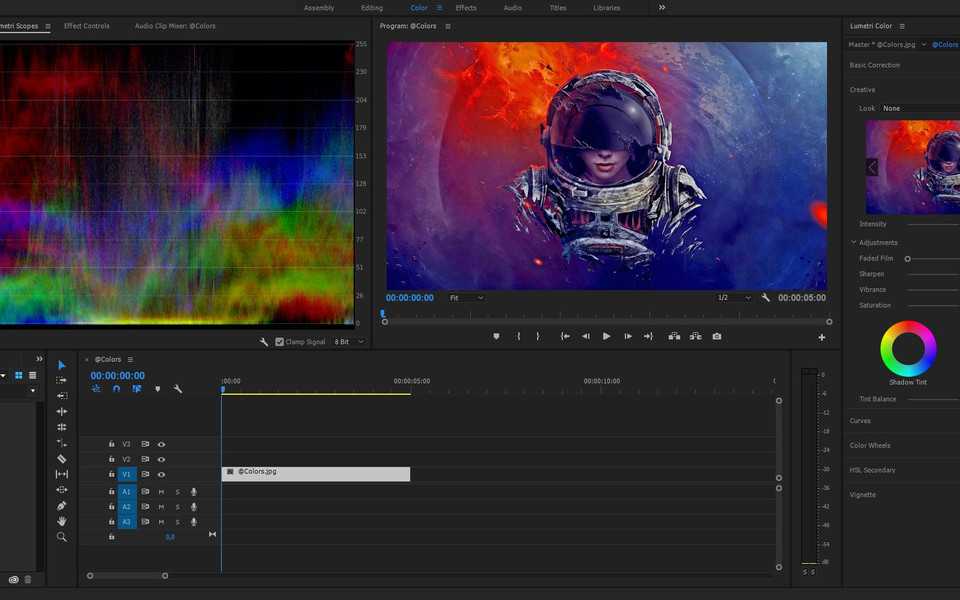
Затем мы проверили, увеличивают ли структурированные 3′-мотивы в epegRNAs уровень их экспрессии по сравнению с pegRNAs. Количественная оценка RT-qPCR каркаса пегРНК выявила зависящие от мишени различия в уровнях экспрессии эпигРНК по сравнению с немодифицированными пегРНК (дополнительная рис. 7).
Для пегРНК, которая является матрицей вставки тега +1 FLAG по адресу HEK3 , мы наблюдали, что добавление evopreQ 1 или mpknot снижало экспрессию пегРНК в 9,2-9,6 раза, несмотря на то, что приводило к 1,9-кратному повышению эффективности Вставка эпитопа метки FLAG по адресу HEK3 (). Сходным образом, epegRNAs, которые шаблонируют трансверсию DNMT1 , также обнаруживают пониженную экспрессию (в 1,6-2,1 раза). Тем не менее, epegRNAs, которые матричные трансверсии в RNF2 или EMX1 экспрессировались на более высоких уровнях, чем у немодифицированной пегРНК (в 2,2–2,4 раза и в 1,4–3,7 раза соответственно, дополнительная рис.
 7). Эти данные свидетельствуют о том, что 3′-мотивы непоследовательно влияют на экспрессию pegRNA, что согласуется с нашими более ранними выводами (1), что эффективность PE в этих условиях трансфекции не ограничивается экспрессией pegRNA в клетках HEK293T. Однако, когда экспрессия epegRNA более ограничена, улучшение экспрессии epegRNA может еще больше повысить эффективность редактирования.
7). Эти данные свидетельствуют о том, что 3′-мотивы непоследовательно влияют на экспрессию pegRNA, что согласуется с нашими более ранними выводами (1), что эффективность PE в этих условиях трансфекции не ограничивается экспрессией pegRNA в клетках HEK293T. Однако, когда экспрессия epegRNA более ограничена, улучшение экспрессии epegRNA может еще больше повысить эффективность редактирования.Затем мы проверили, снижает ли добавление структурного мотива 3′-РНК участие целевого участка ДНК, сравнив способность эпигРНК и пегРНК поддерживать активацию транскрипции слияниями dCas9–VP64–p65–Rta (dCas9–VPR) 32,33 . Клетки HEK293T трансфицировали плазмидами, кодирующими dCas9-VPR, GFP ниже целевого протоспейсера HEK3 , DNMT1 , RNF2 или EMX1 , а также пегРНК, эпиРНК или sgРНК, нацеленных на соответствующие сайты. Активацию транскрипции измеряли по флуоресценции клеточного GFP. В отличие от их способности усиливать активность PE (1), epegRNAs демонстрировали сходную активность Cas9.
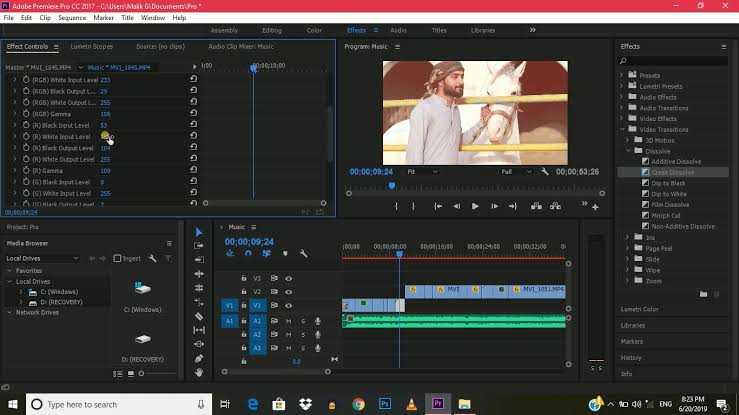 -зависимая активация транскрипции в клетках HEK293T в виде пегРНК (). ЭпегРНК и пегРНК приводили к меньшей активации транскрипции по сравнению с sgRNA, нацеленной на один и тот же сайт (в 3,0 раза для пегРНК, в 2,3 раза для evopreQ 1 epegRNA и в 1,9 раза для mpknot epegRNA по четырем сайтам), что позволяет предположить, что 3 ‘удлинение в pegRNAs и epegRNAs незначительно препятствует вовлечению сайта-мишени.
-зависимая активация транскрипции в клетках HEK293T в виде пегРНК (). ЭпегРНК и пегРНК приводили к меньшей активации транскрипции по сравнению с sgRNA, нацеленной на один и тот же сайт (в 3,0 раза для пегРНК, в 2,3 раза для evopreQ 1 epegRNA и в 1,9 раза для mpknot epegRNA по четырем сайтам), что позволяет предположить, что 3 ‘удлинение в pegRNAs и epegRNAs незначительно препятствует вовлечению сайта-мишени.Чтобы выявить потенциальные изменения в вовлечении целевого сайта и различия в экспрессии пегРНК и эпигРНК, мы провели микромасштабный термофорез (МСТ) для измерения аффинности предварительно инкубированных комплексов РНП каталитически инертного Cas9.(dCas9) и pegRNAs или epegRNAs для субстрата dsDNA. Мы обнаружили, что добавление mpknot или evopreQ 1 приводит к сравнимому или незначительному снижению аффинности связывания с двухцепочечной ДНК по сравнению с немодифицированной пегРНК соответственно (K D = 10 нМ для evopreQ 1 epegRNA и 21 нМ для mpknot pegRNA по сравнению с 8,1 нМ для немодифицированная пегРНК).
 Аффинность pegRNAs к никазе Cas9 H840A также была незначительно снижена для любого мотива (K D = 18 нМ для evopreQ 1 epegRNA, 11 нМ для mpknot pegRNA и 5 нМ для немодифицированной pegRNA; ). Эти данные свидетельствуют о том, что повышенная эффективность PE от epegRNAs не возникает из-за улучшенного связывания pegRNA с Cas9.или комплекса PE RNP к целевому сайту.
Аффинность pegRNAs к никазе Cas9 H840A также была незначительно снижена для любого мотива (K D = 18 нМ для evopreQ 1 epegRNA, 11 нМ для mpknot pegRNA и 5 нМ для немодифицированной pegRNA; ). Эти данные свидетельствуют о том, что повышенная эффективность PE от epegRNAs не возникает из-за улучшенного связывания pegRNA с Cas9.или комплекса PE RNP к целевому сайту.В совокупности эти результаты позволяют предположить, что epegRNAs более устойчивы к клеточной деградации, чем pegRNAs, и, таким образом, генерируют меньше видов усеченных pegRNA, которые снижают эффективность первичного редактирования. Нельзя исключать дополнительные механизмы, лежащие в основе улучшений от epegRNAs.
Оптимизация сконструированных 3′-мотивов pegRNA
Установив, что epegRNAs улучшают эффективность редактирования за счет устойчивости к экзонуклеолитической деградации, мы предположили, что более стабильные мотивы РНК могут дополнительно улучшать активность PE. Мы провели скрининг 25 дополнительных мотивов структурированной РНК на предмет их способности улучшать эффективность редактирования epegRNA среди эпэгРНК, кодирующих либо вставку эпитопной метки FLAG длиной 24 п.
 н., либо делецию длиной 15 п.н., либо трансверсию HEK3 или RNF2 (дополнительные рисунки 9 и 10). Эти мотивы включали дополнительные развившиеся прекеозины 1 -1 рибопереключающие аптамеры 21 , варианты mpknot с улучшенной стабильностью псевдоузлов 22 , G-квадруплексы с возрастающей стабильностью 34 , 15-п.н. Домен P4-P6 группы I интрона 36 . В то время как 123 из 137 протестированных epegRNA продемонстрировали улучшенное общее прайм-редактирование по сравнению с соответствующими pegRNA, ни одна из них не продемонстрировала устойчивых улучшений по сравнению с evopreQ 9.0329 1 или mpknot в большинстве проверенных правок (дополнительные рисунки 9 и 10).
н., либо делецию длиной 15 п.н., либо трансверсию HEK3 или RNF2 (дополнительные рисунки 9 и 10). Эти мотивы включали дополнительные развившиеся прекеозины 1 -1 рибопереключающие аптамеры 21 , варианты mpknot с улучшенной стабильностью псевдоузлов 22 , G-квадруплексы с возрастающей стабильностью 34 , 15-п.н. Домен P4-P6 группы I интрона 36 . В то время как 123 из 137 протестированных epegRNA продемонстрировали улучшенное общее прайм-редактирование по сравнению с соответствующими pegRNA, ни одна из них не продемонстрировала устойчивых улучшений по сравнению с evopreQ 9.0329 1 или mpknot в большинстве проверенных правок (дополнительные рисунки 9 и 10).Затем мы предположили, что удаление ненужной последовательности из добавленных мотивов evopreQ 1 и mpknot может еще больше улучшить дизайн epegRNA, поскольку удаление посторонних последовательностей в структурированной РНК может уменьшить склонность к неправильной укладке 37 .
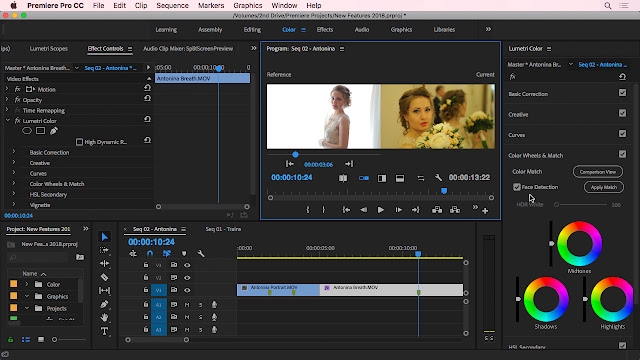 Мы обнаружили, что удаление 5 нуклеотидов избыточной последовательности из evopreQ 1 или mpknot привело к незначительному увеличению средней эффективности редактирования PE3 по сравнению с полноразмерными epegRNAs (дополнительная рис. 10). Поскольку обрезка этих РНК-мотивов не оказывает отрицательного влияния на эффективность редактирования, а более короткие epegRNAs легче получают путем химического синтеза, мы решили использовать урезанный evopreQ 9.0329 1 (tevopreQ 1 ) в epegRNAs при применении epegRNAs для установки терапевтически значимых мутаций (см. ниже).
Мы обнаружили, что удаление 5 нуклеотидов избыточной последовательности из evopreQ 1 или mpknot привело к незначительному увеличению средней эффективности редактирования PE3 по сравнению с полноразмерными epegRNAs (дополнительная рис. 10). Поскольку обрезка этих РНК-мотивов не оказывает отрицательного влияния на эффективность редактирования, а более короткие epegRNAs легче получают путем химического синтеза, мы решили использовать урезанный evopreQ 9.0329 1 (tevopreQ 1 ) в epegRNAs при применении epegRNAs для установки терапевтически значимых мутаций (см. ниже).Мы также исследовали, будет ли каркас sgRNA с «переворотом и удлинением» (F+E) 38 повышать эффективность редактирования epegRNA. Этот каркас направляющей РНК мутирует четвертую пару оснований прямого повтора с U•A на A•U для удаления потенциального терминатора pol III и удлиняет прямой повтор на пять пар оснований для улучшения связывания Cas9 38 . Мы преобразовали HEK293T-клетки с лентивирусами, кодирующими немодифицированную (F+E) пегРНК, (F+E) эпегРНК, содержащую tevopreQ 1 , или tevopreQ 1 epegRNA со стандартным каркасом, который матрицирует трансверсию по HEK3 или DNMT1 или вставка из 3 нт по адресу HEK3 .
 Использование tevopreQ 1 значительно повысило эффективность редактирования (в 3,8 раза для трансверсии нуклеотидов и в 2,6 раза для вставки 3 нуклеотидов в HEK3 и в 6,8 раза в DNMT1 ) (дополнительный рис. 11). Использование каркаса (F+E) в эпегРНК tevopreQ 1 дополнительно повысило эффективность редактирования (в 1,1 раза для трансверсии нуклеотидов, в 1,5 раза для вставки 3 нуклеотидов в HEK3 и в 2,5 раза в DNMT1). ). Мы также охарактеризовали варианты каркаса sgRNA, ранее показавшие, что они повышают активность Cas9-нуклеазы 39 в условиях трансфекции с уменьшенным количеством плазмиды, и наблюдали аналогичные общие преимущества, хотя и с большей вариабельностью (дополнительное обсуждение, дополнительная рис. 12). Эти находки также предполагают, что epegRNAs обеспечивают большее улучшение эффективности PE, когда экспрессия ограничена. Кроме того, эти данные подчеркивают потенциал модифицированных каркасов для повышения эффективности PE в сочетании с epegRNAs, хотя необходимо более глубокое исследование этой возможности.
Использование tevopreQ 1 значительно повысило эффективность редактирования (в 3,8 раза для трансверсии нуклеотидов и в 2,6 раза для вставки 3 нуклеотидов в HEK3 и в 6,8 раза в DNMT1 ) (дополнительный рис. 11). Использование каркаса (F+E) в эпегРНК tevopreQ 1 дополнительно повысило эффективность редактирования (в 1,1 раза для трансверсии нуклеотидов, в 1,5 раза для вставки 3 нуклеотидов в HEK3 и в 2,5 раза в DNMT1). ). Мы также охарактеризовали варианты каркаса sgRNA, ранее показавшие, что они повышают активность Cas9-нуклеазы 39 в условиях трансфекции с уменьшенным количеством плазмиды, и наблюдали аналогичные общие преимущества, хотя и с большей вариабельностью (дополнительное обсуждение, дополнительная рис. 12). Эти находки также предполагают, что epegRNAs обеспечивают большее улучшение эффективности PE, когда экспрессия ограничена. Кроме того, эти данные подчеркивают потенциал модифицированных каркасов для повышения эффективности PE в сочетании с epegRNAs, хотя необходимо более глубокое исследование этой возможности.
Вычислительный инструмент для разработки линкеров эпигРНК
В отличие от белковых линкеров, линкеры РНК более вероятно зависят от последовательности, так что один и тот же линкер может функционировать для одной эпигРНК, но препятствовать другой. Чтобы свести к минимуму возможность вмешательства со стороны линкера эпигРНК, мы разработали pegLIT (инструмент идентификации линкера пегРНК) (дополнительное обсуждение, дополнительная рис. 13), вычислительный инструмент, который идентифицирует последовательности линкера, предсказанные для минимальной пары оснований с остатком epegRNA. Для первоначальной проверки мы протестировали два набора из 15 evopreQ 9.0329 1 epegRNAs с разными линкерами, матрицирующими трансверсию C•G-to-A•T в RNF2 или делецию 15 п.н. в DNMT1 . В каждом наборе pegLIT рекомендовал пять линкеров; пять были предсказаны как пары оснований со спейсером, а пять были предсказаны как пары оснований с PBS. Использование разработанных pegLIT линкеров привело к умеренному повышению эффективности редактирования PE3 по сравнению с использованием разработанных вручную линкеров (в 1,2 раза выше для RNF2 и и в 1,1 раза выше для DNMT1 ) (дополнительный рис.
 13). В то время как взаимодействия спейсеров не оказывали существенного влияния на эффективность редактирования, взаимодействия линкер-PBS коррелировали со снижением эффективности редактирования PE3, что приводило к снижению эффективности редактирования в 1,3 и 1,1 раза по сравнению с линкерами pegLIT для RNF2 и DNMT1 соответственно. Два линкера с наихудшими показателями, которые привели к менее эффективному редактированию PE3 в 1,9 и 3,4 раза при RNF2 по сравнению с оптимальными последовательностями линкера, были правильно идентифицированы с помощью pegLIT как плохие оценки для взаимодействий с PBS (дополнительная рис. 13). Ближайшая близость линкера к PBS по сравнению со спейсером может дать взаимодействию линкер:PBS энтропийное преимущество по сравнению с парой линкер:спейсер. Затем мы попытались определить, могут ли линкерные последовательности, сконструированные pegLIT, повысить эффективность двух эпигРНК (шаблон трансверсии G•C-to-T•A в положении 9).
13). В то время как взаимодействия спейсеров не оказывали существенного влияния на эффективность редактирования, взаимодействия линкер-PBS коррелировали со снижением эффективности редактирования PE3, что приводило к снижению эффективности редактирования в 1,3 и 1,1 раза по сравнению с линкерами pegLIT для RNF2 и DNMT1 соответственно. Два линкера с наихудшими показателями, которые привели к менее эффективному редактированию PE3 в 1,9 и 3,4 раза при RNF2 по сравнению с оптимальными последовательностями линкера, были правильно идентифицированы с помощью pegLIT как плохие оценки для взаимодействий с PBS (дополнительная рис. 13). Ближайшая близость линкера к PBS по сравнению со спейсером может дать взаимодействию линкер:PBS энтропийное преимущество по сравнению с парой линкер:спейсер. Затем мы попытались определить, могут ли линкерные последовательности, сконструированные pegLIT, повысить эффективность двух эпигРНК (шаблон трансверсии G•C-to-T•A в положении 9). 0011 EMX1 и делеция 15 п.н. в VEGFA ), которые изначально не демонстрировали улучшенного редактирования (дополнительная рис. 4). Действительно, использование линкеров, разработанных pegLIT, повысило эффективность редактирования PE3 в 1,3 и 1,4 раза соответственно по сравнению с пегРНК для этих двух правок (дополнительная рис. 13). В совокупности эти результаты демонстрируют, что pegLIT облегчает использование epegRNAs для последовательного улучшения основных результатов редактирования.
0011 EMX1 и делеция 15 п.н. в VEGFA ), которые изначально не демонстрировали улучшенного редактирования (дополнительная рис. 4). Действительно, использование линкеров, разработанных pegLIT, повысило эффективность редактирования PE3 в 1,3 и 1,4 раза соответственно по сравнению с пегРНК для этих двух правок (дополнительная рис. 13). В совокупности эти результаты демонстрируют, что pegLIT облегчает использование epegRNAs для последовательного улучшения основных результатов редактирования.Мы также исследовали, улучшают ли линкеры, сконструированные с помощью pegLIT, активность эпэгРНК по сравнению с эпэгРНК без линкеров. По сравнению с epegRNAs mpknot без линкера добавление линкера, разработанного pegLIT, привело к значительному повышению эффективности редактирования, чем при использовании линкеров, разработанных вручную (дополнительные рисунки 3 и 13). Напротив, использование линкеров pegLIT с evopreQ 1 или tevopreQ 1 epegRNAs значительно не увеличивали редактирование по сравнению с epegRNAs без линкера (дополнительная рис.
 13). Поэтому мы рекомендуем использовать разработанные pegLIT линкеры в epegRNAs с использованием более крупных мотивов структурированной РНК, таких как mpknot.
13). Поэтому мы рекомендуем использовать разработанные pegLIT линкеры в epegRNAs с использованием более крупных мотивов структурированной РНК, таких как mpknot.Повышенная эффективность редактирования с помощью химически модифицированных эпэгРНК
Химически синтезированные гРНК обычно используются при трансфекции клеток мРНК или РНП 40 . Хотя синтетические гРНК могут включать химические модификации, повышающие устойчивость к экзонуклеолитической деградации 16,40 , мы предположили, что структурные мотивы все еще могут способствовать дополнительным улучшениям в сочетании с такими модификациями.
Чтобы проверить эту возможность, мы сравнили эффективность первичного редактирования синтетических tevopreQ 1 эпэгРНК с эффективностью синтетических эпэгРНК, которые устанавливают либо точечную мутацию, либо делецию 15 п. , RUNX1 и EMX1 ) в ячейках HEK293T. Как epegRNAs, так и pegRNAs содержали 2′-O-метильные модификации и фосфоротиоатные связи между первым и последним тремя нуклеотидами РНК.
 Для шести из семи протестированных pegRNAs соответствующие epegRNAs демонстрировали в 1,1–3,1 раза более высокое редактирование с неизменными соотношениями edit:indel (дополнительная рис. 14). Эти данные предполагают, что epegRNAs также улучшают результаты PE по сравнению с pegRNAs в приложениях, которые используют химически синтезированные и модифицированные pegRNAs.
Для шести из семи протестированных pegRNAs соответствующие epegRNAs демонстрировали в 1,1–3,1 раза более высокое редактирование с неизменными соотношениями edit:indel (дополнительная рис. 14). Эти данные предполагают, что epegRNAs также улучшают результаты PE по сравнению с pegRNAs в приложениях, которые используют химически синтезированные и модифицированные pegRNAs.Сконструированные пегРНК улучшают первичное редактирование терапевтически значимых мутаций
Подтвердив использование эпегРНК в качестве стратегии широкого улучшения активности ПЭ, мы затем сравнили активность эпегРНК, содержащих tevopreQ 1 , с активностью пегРНК для установки различных защитных или терапевтические генетические мутации. Мы успешно использовали epegRNAs для установки аллеля PRNP G127V, защищающего от прионной болезни человека 41,42 в HEK29.3Т-клетки с эффективностью в 1,4 раза выше, чем у канонической пегРНК (). Кроме того, мы использовали эпегРНК для коррекции наиболее распространенной причины болезни Тея-Сакса ( HEXA 1278+TATC ) в ранее сконструированных клеточных линиях HEXA 1278+TATC HEK с помощью плазмидной липофекции и в первичных фибробластах, полученных от пациентов, с помощью нуклеофекции in vitro , транскрибируемой мРНК и синтетической пегРНК (и).
 В обоих случаях мы наблюдали повышение эффективности редактирования для tevopreQ 9.0329 1 epegRNAs, содержащие разработанные pegLIT 8-нуклеотидные линкеры по сравнению с каноническими pegRNAs (в 2,8 раза выше в клетках HEK293T и в 2,3 раза выше в фибробластах, полученных от пациентов).
В обоих случаях мы наблюдали повышение эффективности редактирования для tevopreQ 9.0329 1 epegRNAs, содержащие разработанные pegLIT 8-нуклеотидные линкеры по сравнению с каноническими pegRNAs (в 2,8 раза выше в клетках HEK293T и в 2,3 раза выше в фибробластах, полученных от пациентов).Открыть в отдельном окне
Эффективность терапевтически значимого редактирования генома при первичном редактировании повышается за счет использования эпегРНК.
( a ) PE3-опосредованная установка мутации G127V в PRNP , которая защищает от прионной болезни человека 41,42 и ( b ) коррекция патогенной вставки c1278TATC в HEXA , которая вызывает болезнь Тея-Сакса как в клетках HEK293T, так и ( c ) в первичных фибробластах, полученных от пациента. ( d ) Сравнение PE2-опосредованной установки патогенных и защитных аллелей с использованием неоптимизированных epegRNAs или неоптимизированных pegRNAs в девяти геномных сайтах.
 Справочные обозначения SNP (rs) можно найти для всех мутаций в дополнительной таблице 6. Данные и планки погрешностей в a, d и e отражают среднее значение и стандартное отклонение трех независимых биологических повторов.
Справочные обозначения SNP (rs) можно найти для всех мутаций в дополнительной таблице 6. Данные и планки погрешностей в a, d и e отражают среднее значение и стандартное отклонение трех независимых биологических повторов.Установка терапевтически релевантных правок с использованием неоптимизированных epegRNAs
Дизайн и скрининг многих pegRNAs с различными матрицами PBS и RT является важным первым шагом в успешном использовании первичного редактирования 14 . Хотя общие правила, регулирующие длину и состав матрицы PBS и RT, были описаны 14,43 , идентификация оптимальных пегРНК часто требует тщательного скрининга конструкций пегРНК. Мы предположили, что epegRNAs могут поддерживать более эффективную установку терапевтически релевантных простых правок без обширной оптимизации pegRNA. Мы исследовали способность неоптимизированных pegRNAs и epegRNAs шаблонировать установку девяти защитных или патогенных точечных мутаций, используя PE2. Во всех случаях пегРНК и эпегРНК, использованные в этом эксперименте, содержали 13-нуклеотидный PBS и матрицу ОТ, содержащую 10 нуклеотидов, гомологичных целевому сайту после последнего отредактированного нуклеотида, за исключением случаев, когда 3′-удлинение начиналось с цитозина 9.
 0235 14 , и в этом случае он был расширен до ближайшего нуклеотида, отличного от C.
0235 14 , и в этом случае он был расширен до ближайшего нуклеотида, отличного от C.Мы исследовали pegRNAs, которые устанавливают терапевтически значимые мутации, связанные с болезнью Альцгеймера 44 , ишемической болезнью сердца 45,46 , диабетом 2 типа 47 , врожденным иммунитетом 48 , 6 902 нарушением дефицита ламин 3 CDKL5 4 90 дефицит 50 и синдром Ретта 51,52 . Мы сравнили результаты первичного редактирования с помощью пегРНК и соответствующего tevopreQ 9.0329 1 epegRNAs с 8-нуклеотидными линкерами pegLIT в клетках HEK293T (). Для каждой мишени тестировалась только одна конструкция pegRNA или epegRNA. В каждом случае epegRNAs превосходили pegRNAs по эффективности редактирования. Для пяти из девяти протестированных терапевтически значимых правок epegRNAs приводили к эффективности редактирования ≥20%, чего обычно достаточно для создания модельных клеточных линий. Для сравнения, только три из девяти пегРНК достигли такого уровня эффективности редактирования.
 Более высокая эффективность редактирования, опосредованная epegRNAs (в среднем в 2,8 раза выше, чем у pegRNAs), должна упростить производство гомозиготных клеточных линий, что является важным фактором для моделирования рецессивных мутаций. Точно так же неоптимизированные epegRNAs опосредовали вставку метки FLAG из 24 п.н. с эффективностью ≥10% в 5 из 15 протестированных сайтов; соответствующие пегРНК не достигли эффективности ≥10% ни на одном из протестированных сайтов (дополнительное обсуждение и дополнительная рис. 15). В совокупности эти данные демонстрируют, что epegRNAs упрощают производство модельных клеточных линий с PE.
Более высокая эффективность редактирования, опосредованная epegRNAs (в среднем в 2,8 раза выше, чем у pegRNAs), должна упростить производство гомозиготных клеточных линий, что является важным фактором для моделирования рецессивных мутаций. Точно так же неоптимизированные epegRNAs опосредовали вставку метки FLAG из 24 п.н. с эффективностью ≥10% в 5 из 15 протестированных сайтов; соответствующие пегРНК не достигли эффективности ≥10% ни на одном из протестированных сайтов (дополнительное обсуждение и дополнительная рис. 15). В совокупности эти данные демонстрируют, что epegRNAs упрощают производство модельных клеточных линий с PE.Здесь мы сообщаем о дизайне, характеристике и проверке сконструированных пегРНК для устранения основного узкого места в основном редактировании. Эти epegRNAs содержат мотив структурированной РНК 3′ от PBS, который предотвращает деградацию удлинения pegRNA и последующее образование некомпетентных к редактированию PE-комплексов, которые конкурируют за доступ к целевому геномному сайту.
 Мы обнаружили, что epegRNAs в целом улучшают эффективность PE во всех пяти протестированных клеточных линиях и первичных типах клеток, причем более значительные улучшения наблюдаются в клеточных линиях, трансфекция которых затруднена. Кроме того, мы заметили, что использование epegRNAs может повысить эффективность первичного редактирования при использовании химически модифицированных pegRNA, при установке терапевтически релевантных изменений в клетках человека и при использовании неоптимизированных конструкций pegRNA. Наконец, мы описываем вычислительную программу, которая ускоряет дизайн epegRNA, идентифицируя линкеры, которые минимизируют риск контрпродуктивной вторичной структуры. В целом, наши результаты показывают, что epegRNAs в целом улучшают результаты первичного редактирования в самых разных геномных локусах, типах редактирования (замены, вставки и делеции) и типах клеток.
Мы обнаружили, что epegRNAs в целом улучшают эффективность PE во всех пяти протестированных клеточных линиях и первичных типах клеток, причем более значительные улучшения наблюдаются в клеточных линиях, трансфекция которых затруднена. Кроме того, мы заметили, что использование epegRNAs может повысить эффективность первичного редактирования при использовании химически модифицированных pegRNA, при установке терапевтически релевантных изменений в клетках человека и при использовании неоптимизированных конструкций pegRNA. Наконец, мы описываем вычислительную программу, которая ускоряет дизайн epegRNA, идентифицируя линкеры, которые минимизируют риск контрпродуктивной вторичной структуры. В целом, наши результаты показывают, что epegRNAs в целом улучшают результаты первичного редактирования в самых разных геномных локусах, типах редактирования (замены, вставки и делеции) и типах клеток.Улучшения в первичном редактировании с помощью epegRNAs, вероятно, зависят от стратегии доставки.
 Способы доставки с более низким уровнем экспрессии, такие как некоторые вирусные векторы, могут более эффективно использовать epegRNAs, когда концентрация pegRNA ограничивается (дополнительная рис. 12). Точно так же дальнейшие улучшения в синтезе химически модифицированных РНК могут уменьшить преимущества epegRNAs за счет лучшего смягчения деградации. Кроме того, большая длина epegRNAs (дополнительно 37 нуклеотидов при использовании tevopreQ 1 ) является важным соображением при использовании синтетических epegRNAs, учитывая текущие проблемы химического синтеза более длинных РНК.
Способы доставки с более низким уровнем экспрессии, такие как некоторые вирусные векторы, могут более эффективно использовать epegRNAs, когда концентрация pegRNA ограничивается (дополнительная рис. 12). Точно так же дальнейшие улучшения в синтезе химически модифицированных РНК могут уменьшить преимущества epegRNAs за счет лучшего смягчения деградации. Кроме того, большая длина epegRNAs (дополнительно 37 нуклеотидов при использовании tevopreQ 1 ) является важным соображением при использовании синтетических epegRNAs, учитывая текущие проблемы химического синтеза более длинных РНК.Мы рекомендуем использовать эпигРНК для всех экспериментов по первичному редактированию, которые могут поддерживать немного более длинные пегРНК. Важно отметить, что исследователи, стремящиеся идентифицировать первичные агенты редактирования, которые устанавливают желаемое редактирование с максимально возможной эффективностью, должны продолжать тестировать множество epegRNAs, которые включают различные матричные последовательности и длины PBS и RT, а также различные sgRNA с разрывом при использовании PE3.
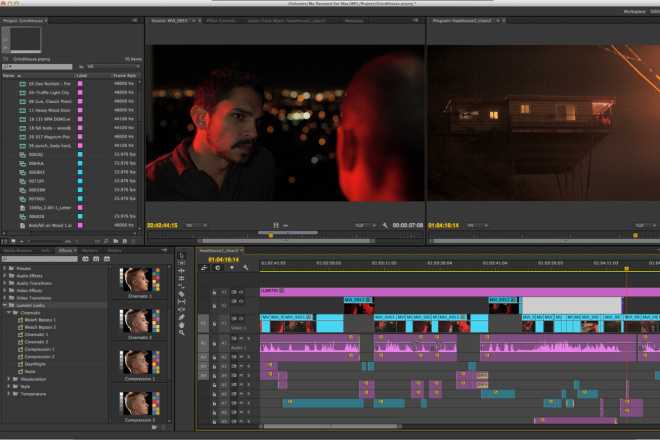 Включение вариантов каркаса направляющей РНК 38,39 также может дополнительно повысить эффективность PE в зависимости от сайта (дополнительные рисунки 11 и 12). Однако, как показано в этом исследовании, обширный скрининг может не понадобиться, если максимальная эффективность редактирования не является критически важной. В этих случаях epegRNA, содержащая урезанный мотив evopreQ 1 (tevopreQ 1 ) с длиной PBS 13 и матрицей, которая включает либо 10 нуклеотидов гомологии после целевого редактирования для небольших вставок, делеций и точечных мутаций — или 25 нуклеотидов гомологии для более крупных вставок или делеций — обеспечивает многообещающую отправную точку для дизайна epegRNA. Затем можно оптимизировать PBS, длину шаблона RT, последовательность каркаса и разрыв sgRNA, если наблюдаемая эффективность редактирования недостаточна.
Включение вариантов каркаса направляющей РНК 38,39 также может дополнительно повысить эффективность PE в зависимости от сайта (дополнительные рисунки 11 и 12). Однако, как показано в этом исследовании, обширный скрининг может не понадобиться, если максимальная эффективность редактирования не является критически важной. В этих случаях epegRNA, содержащая урезанный мотив evopreQ 1 (tevopreQ 1 ) с длиной PBS 13 и матрицей, которая включает либо 10 нуклеотидов гомологии после целевого редактирования для небольших вставок, делеций и точечных мутаций — или 25 нуклеотидов гомологии для более крупных вставок или делеций — обеспечивает многообещающую отправную точку для дизайна epegRNA. Затем можно оптимизировать PBS, длину шаблона RT, последовательность каркаса и разрыв sgRNA, если наблюдаемая эффективность редактирования недостаточна.Общие методы.
Плазмиды, экспрессирующие pegRNAs и epegRNAs, были клонированы с помощью сборки Gibson, сборки Golden Gate с использованием либо ранее описанной заказной акцепторной плазмиды 14 , либо недавно разработанных заказных плазмид-акцептора, содержащих обрезанный evopreQ 1 или mpknot (использование которых описано в дополнительном примечании 1), или синтезированы и клонированы Twist Biosciences.
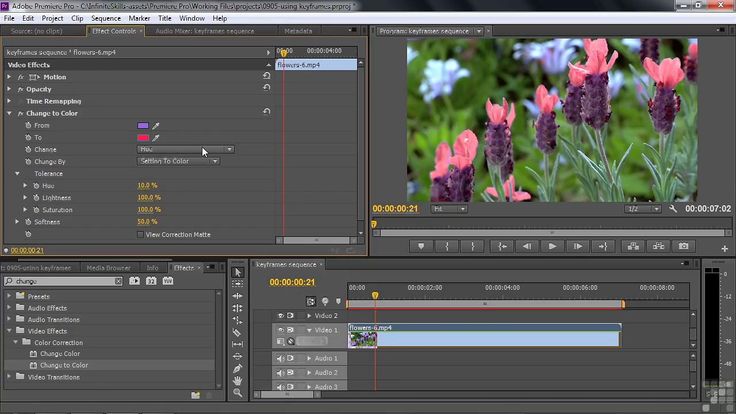 Плазмиды, экспрессирующие sgRNA, клонировали с помощью сборки Gibson или USER. Амплификацию ДНК проводили с помощью ПЦР с использованием Phusion U или High Fidelity Phusion Green Hot Start II (New England Biolabs). Плазмиды, экспрессирующие пегРНК, очищали с использованием минипрепаратов плазмид PureYield (Promega) при трансфекции HEK29.3T или наборами Plasmid Plus Midiprep (Qiagen) при трансфекции других типов клеток, в то время как плазмиды, экспрессирующие главные редакторы, очищали исключительно с использованием наборов Plasmid Plus Midiprep. Плазмиды, заказанные в Twist Biosciences, ресуспендировали в воде, не содержащей нуклеаз, и использовали непосредственно. Праймеры и фрагменты двухцепочечной ДНК заказывали в компании Integrated DNA Technologies (IDT). Необрезанные гели агарозы и нозерн-блоттинга представлены на дополнительных рисунках. 16 и 17.
Плазмиды, экспрессирующие sgRNA, клонировали с помощью сборки Gibson или USER. Амплификацию ДНК проводили с помощью ПЦР с использованием Phusion U или High Fidelity Phusion Green Hot Start II (New England Biolabs). Плазмиды, экспрессирующие пегРНК, очищали с использованием минипрепаратов плазмид PureYield (Promega) при трансфекции HEK29.3T или наборами Plasmid Plus Midiprep (Qiagen) при трансфекции других типов клеток, в то время как плазмиды, экспрессирующие главные редакторы, очищали исключительно с использованием наборов Plasmid Plus Midiprep. Плазмиды, заказанные в Twist Biosciences, ресуспендировали в воде, не содержащей нуклеаз, и использовали непосредственно. Праймеры и фрагменты двухцепочечной ДНК заказывали в компании Integrated DNA Technologies (IDT). Необрезанные гели агарозы и нозерн-блоттинга представлены на дополнительных рисунках. 16 и 17.Синтетические пегРНК и генерация транскрибируемой in vitro мРНК.
Синтетические пегРНК были заказаны в IDT и содержали модификации 2′-O-метила в первых и последних трех нуклеотидах и фосфоротиоатные связи между тремя первым и последним нуклеотидами и использовались непосредственно.
 Синтетические разрывные sgRNA были заказаны у Synthego и содержали модификации 2′-O-метила в трех первых и последних нуклеотидах и фосфоротиоатные связи между первыми тремя и последними двумя нуклеотидами. PE-кодируемая мРНК была транскрибирована in vitro с использованием протокола, описанного ранее 53 . Вкратце, кассету PE2, состоящую из 5′-UTR, последовательности Козака, ORF PE2 и 3′-UTR, клонировали в плазмиду, содержащую неактивный промотор T7 (dT7). Матрица транскрипции мРНК была создана с помощью ПЦР с использованием праймера для установки правильной последовательности промотора Т7 и обратного праймера, который устанавливал поли-А-хвост. мРНК получали с использованием набора HiScribe T7 High-Yield RNA Kit (New England Biolabs) в соответствии с инструкциями производителя, за исключением того, что N1-метилпсевдоуридинтрифосфат (Trilink) был заменен на уридинтрифосфат, а CleanCapAG (Trilink) был добавлен для обеспечения ко- транскрипционное кэпирование. Полученную мРНК очищали путем осаждения хлоридом лития и восстанавливали в буфере ТЕ (10 мМ Трис, 1 мМ ЭДТА, рН 8,0 при 25 °С).
Синтетические разрывные sgRNA были заказаны у Synthego и содержали модификации 2′-O-метила в трех первых и последних нуклеотидах и фосфоротиоатные связи между первыми тремя и последними двумя нуклеотидами. PE-кодируемая мРНК была транскрибирована in vitro с использованием протокола, описанного ранее 53 . Вкратце, кассету PE2, состоящую из 5′-UTR, последовательности Козака, ORF PE2 и 3′-UTR, клонировали в плазмиду, содержащую неактивный промотор T7 (dT7). Матрица транскрипции мРНК была создана с помощью ПЦР с использованием праймера для установки правильной последовательности промотора Т7 и обратного праймера, который устанавливал поли-А-хвост. мРНК получали с использованием набора HiScribe T7 High-Yield RNA Kit (New England Biolabs) в соответствии с инструкциями производителя, за исключением того, что N1-метилпсевдоуридинтрифосфат (Trilink) был заменен на уридинтрифосфат, а CleanCapAG (Trilink) был добавлен для обеспечения ко- транскрипционное кэпирование. Полученную мРНК очищали путем осаждения хлоридом лития и восстанавливали в буфере ТЕ (10 мМ Трис, 1 мМ ЭДТА, рН 8,0 при 25 °С). Последовательности пегРНК и sgРНК, используемые в этом исследовании, можно найти в дополнительной таблице 1. Список мотивов структурированной РНК, исследованных в этом исследовании, можно найти в дополнительной таблице 2.
Последовательности пегРНК и sgРНК, используемые в этом исследовании, можно найти в дополнительной таблице 1. Список мотивов структурированной РНК, исследованных в этом исследовании, можно найти в дополнительной таблице 2.Общие условия культивирования клеток млекопитающих.
Клетки HEK293T (ATCC CRL-3216), U2OS (ATCC HTB-96), K562 (CCL-243) и HeLa (CCL-2) были приобретены в ATCC, культивированы и пассированы в модифицированной Дульбекко среде Игла (DMEM) с добавлением с GlutaMax (Thermo Fisher Scientific), McCoy’s 5A Medium (Gibco), RPMI Medium 1640 plus GlutaMAX (Gibco) или Eagle’s Minimal Essential Medium (EMEM, ATCC) соответственно, каждая из которых дополнена 10% (об./об.) эмбриональной телячьей сыворотки (Гибко, квалифицированный). Клетки фибробластов пациентов с первичной болезнью Тея-Сакса были получены из Института Кориелла (кат. ID GM00221) и выращены в среде DMEM с низким содержанием глюкозы (Sigma Aldrich) и 10% (об./об.) FBS с добавлением дополнительных 2 мМ L-глутамина ( Термо Фишер Сайентифик).
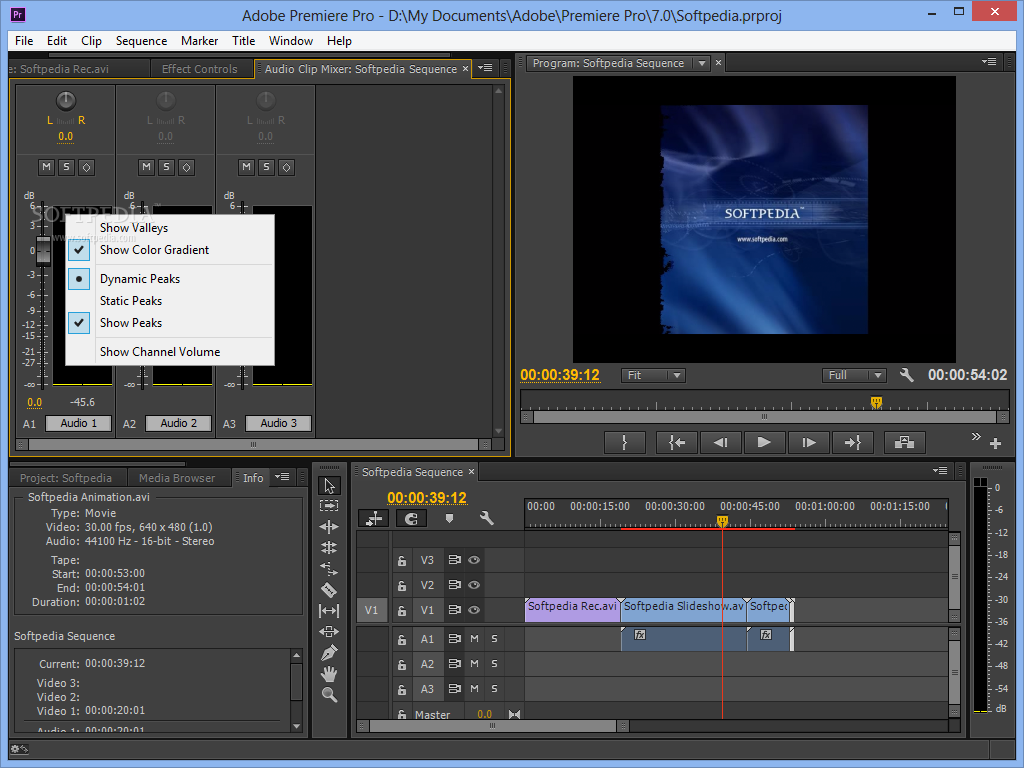 Все типы клеток инкубировали, поддерживали и культивировали при 37 °C с 5% CO 2 . Каждая клеточная линия была аутентифицирована соответствующим поставщиком и дала отрицательный результат на микоплазму.
Все типы клеток инкубировали, поддерживали и культивировали при 37 °C с 5% CO 2 . Каждая клеточная линия была аутентифицирована соответствующим поставщиком и дала отрицательный результат на микоплазму.Протоколы трансфекции культур тканей и нуклеофекции и подготовка геномной ДНК.
Для трансфекции 10 000 клеток HEK293T высевали на лунку 96-луночных планшетов (Corning). Через 16–24 часа после посева клетки трансфицировали примерно при 60% конфлюэнтности 0,5 мкл липофектамина 2000 (Thermo Fisher Scientific) в соответствии с протоколами производителя и 200 нг плазмиды PE, 40 нг плазмиды pegRNA и 13 нг sgRNA. плазмида (для PE3). При трансфекции уменьшенных количеств плазмид, кодируемых редактором, 0,5 мкл липофектамина 2000 использовали для трансфекции 20 нг плазмиды PE, 4 нг плазмиды pegRNA, 1,3 нг плазмиды sgRNA (для PE3) и 228 нг pUC19..
Для нуклеофекции клетки HEK293T подвергали электропорации с in vitro транскрибированной мРНК и синтетической пегРНК с использованием Lonza 4D Nucleofector с набором для клеточной линии SF (Lonza).
 200 000 клеток на электропорацию центрифугировали в течение 8 мин при 120 x g, затем промывали 1 мл PBS (Thermo Fisher Scientific). После второго центрифугирования клетки ресуспендировали в 5 мкл восстановленного буфера SF на образец и добавляли в микрокюветы.
200 000 клеток на электропорацию центрифугировали в течение 8 мин при 120 x g, затем промывали 1 мл PBS (Thermo Fisher Scientific). После второго центрифугирования клетки ресуспендировали в 5 мкл восстановленного буфера SF на образец и добавляли в микрокюветы.На каждую кювету по 17 мкл грузовой смеси (1 мкг мРНК PE2 в 0,5 мкл, 9Добавляли 0 пмоль пегРНК в 0,9 мкл и 60 пмоль никирующей sgРНК в 0,6 мкл и 15 мкл восстановленного буфера SF) и трижды пипетировали вверх и вниз для перемешивания. Клетки электропорировали по программе СМ-130, затем добавляли 80 мкл теплой среды и инкубировали 10 мин при комнатной температуре. Затем смесь пипетировали для перемешивания и добавляли 25 мкл в лунку 48-луночного планшета с конечным объемом культуры 250 мкл на лунку. Для экспериментов на клетках HeLa, U2OS и K562 800 нг PE2-экспрессирующей плазмиды, 200 нг плазмиды, экспрессирующей pegRNA, и 83 нг никирующей sgRNA-экспрессирующей плазмиды подвергали нуклеофекции в конечном объеме 20 мкл в 16-луночной полоске nucleovette ( Лонца).
 Клетки HeLa подвергали нуклеофекции с использованием набора SE Cell Line 4D-Nucleofector X (Lonza) с 2 × 10 5 клеток на образец (программа CN-114), согласно протоколу производителя. Клетки U2OS подвергали нуклеофекции с использованием набора SE Cell Line 4D-Nucleofector X (Lonza) с 2 × 10 5 клеток на образец (программа DN-100) в соответствии с протоколом производителя. Клетки K562 подвергали нуклеофекции с использованием набора SE Cell Line 4D-Nucleofector X (Lonza) с 2 × 10 5 клеток на образец (программа FF-120) в соответствии с протоколом производителя.
Клетки HeLa подвергали нуклеофекции с использованием набора SE Cell Line 4D-Nucleofector X (Lonza) с 2 × 10 5 клеток на образец (программа CN-114), согласно протоколу производителя. Клетки U2OS подвергали нуклеофекции с использованием набора SE Cell Line 4D-Nucleofector X (Lonza) с 2 × 10 5 клеток на образец (программа DN-100) в соответствии с протоколом производителя. Клетки K562 подвергали нуклеофекции с использованием набора SE Cell Line 4D-Nucleofector X (Lonza) с 2 × 10 5 клеток на образец (программа FF-120) в соответствии с протоколом производителя.Фибробласты, полученные от пациентов, подвергали электропорации с мРНК, кодирующей PE2, и синтетической пегРНК, а также надрезом sgRNA, как описано выше для HEK29.Клетки 3T с использованием набора для клеточной линии SE и 100 000 клеток, которые центрифугировали при 100 x g в течение 10 мин. Кроме того, в 48-луночный планшет добавляли 40 мкл восстановленных клеток вместо 25. Во всех случаях клетки культивировали через 3 дня после трансфекции, после чего среду удаляли и клетки промывали PBS (pH 7,4 при 23 °C).
 ) и затем лизировали добавлением 50 мкл для 96-луночных планшетов или 150 мкл для 48-луночных планшетов свежеприготовленного буфера для лизиса (10 мМ трис-HCl, pH 8 при 23 °C; 0,05% SDS; 25 мкг мл -1 Proteinase K (Qiagen)), и инкубацию при 37 °C в течение 1 часа или более, после чего протеиназу K инактивировали в течение 30 минут при 80 °C. Полученную гДНК хранили при температуре -20 °C до использования.
) и затем лизировали добавлением 50 мкл для 96-луночных планшетов или 150 мкл для 48-луночных планшетов свежеприготовленного буфера для лизиса (10 мМ трис-HCl, pH 8 при 23 °C; 0,05% SDS; 25 мкг мл -1 Proteinase K (Qiagen)), и инкубацию при 37 °C в течение 1 часа или более, после чего протеиназу K инактивировали в течение 30 минут при 80 °C. Полученную гДНК хранили при температуре -20 °C до использования.Получение и трансдукция лентивируса.
Лентивирусные плазмиды для переноса были разработаны таким образом, чтобы содержать пегРНК или эпегРНК при экспрессии с промотора U6 человека и маркер PuroR–T2A–BFP при экспрессии с основного промотора EF1α. Для упаковки лентивируса клетки HEK293T высевали на 6-луночные планшеты (Corning) при 7×10 5 клеток на лунку в среде DMEM с добавлением 10% FBS. При 60% слиянии через 16 часов после посева клетки трансфицировали 12 мкл липофектамина 2000 (Thermo Fisher Scientific) в соответствии с протоколом производителя и 1,33 мкг лентивирусной переносной плазмиды, 0,67 мкг pMD2.
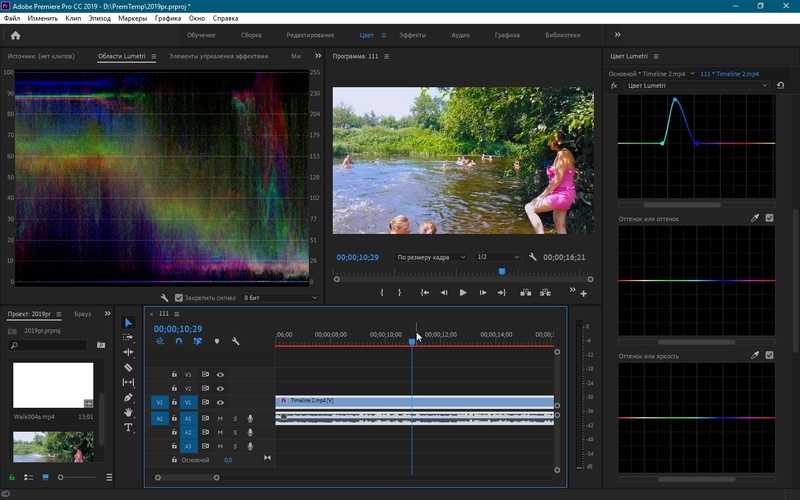 G (Addgene #12259) и 1 мкг psPAX2 ( Addgen #12260). Через 6 часов после трансфекции среду заменяли средой DMEM с добавлением 10% FBS. Через 48 часов после трансфекции супернатант вируса центрифугировали при 3000× g в течение 15 минут для удаления клеточного дебриса, фильтровали через фильтр 0,45 мкм PVDF (Corning) и хранили при –80 °C.
G (Addgene #12259) и 1 мкг psPAX2 ( Addgen #12260). Через 6 часов после трансфекции среду заменяли средой DMEM с добавлением 10% FBS. Через 48 часов после трансфекции супернатант вируса центрифугировали при 3000× g в течение 15 минут для удаления клеточного дебриса, фильтровали через фильтр 0,45 мкм PVDF (Corning) и хранили при –80 °C.Для трансдукции клеток пегРНК или эпегРНК 2 × 10 6 клеток HEK293T инфицировали 20 мкл лентивируса в среде DMEM с добавлением 10% FBS и 8 мкг/мл полибрена (Sigma-Aldrich) и центрифугировали при 1000 × g в течение 2 часов при 33 °C. Через 24 часа после трансдукции клетки пассировали в DMEM с добавлением 10% FBS и 2 мкг/мл пуромицина (Thermo Fisher Scientific) для начала селекции. Флуоресценцию BFP контролировали с помощью проточного цитометра CytoFLEX S (Beckman Couolter), чтобы гарантировать множественность заражения 0,2. Через 4 дня селекции пуромицином трансдуцировали HEK29.Клетки 3T высевали на 96-луночные планшеты (Corning) в количестве 1,6×10 4 клеток на лунку в среде DMEM с добавлением 10% FBS.
 Через 20 часов после посева клетки трансфицировали при 60-80% слиянии 200 нг плазмиды pCMV-PE2 и 0,5 мкл липофектамина 2000 (Thermo Fisher Scientific) в соответствии с протоколом производителя. Для экстракции геномной ДНК через 120 часов после трансфекции клетки промывали PBS (pH 7,4 при 23 °C) и лизировали в 10 мМ Tris-HCl, pH 8,0 при 23 °C; 0,05% ДСН; 800 единиц/мкл протеиназы К (New England BioLabs) при 37°С в течение 1,5 ч с последующей инактивацией фермента при 80°С в течение 30 мин.
Через 20 часов после посева клетки трансфицировали при 60-80% слиянии 200 нг плазмиды pCMV-PE2 и 0,5 мкл липофектамина 2000 (Thermo Fisher Scientific) в соответствии с протоколом производителя. Для экстракции геномной ДНК через 120 часов после трансфекции клетки промывали PBS (pH 7,4 при 23 °C) и лизировали в 10 мМ Tris-HCl, pH 8,0 при 23 °C; 0,05% ДСН; 800 единиц/мкл протеиназы К (New England BioLabs) при 37°С в течение 1,5 ч с последующей инактивацией фермента при 80°С в течение 30 мин.Высокопроизводительное секвенирование образцов геномной ДНК.
Интересующие геномные сайты были амплифицированы из образцов геномной ДНК и секвенированы на Illumina MiSeq, как описано ранее 14 . Нецелевые сайты Cas9 для HEK3 , EMX1 и FANCF ранее были идентифицированы с помощью Guide-Seq 28 . Праймеры, используемые для амплификации геномной ДНК клеток млекопитающих, перечислены в дополнительной таблице 3, а ампликоны перечислены в дополнительной таблице 4.
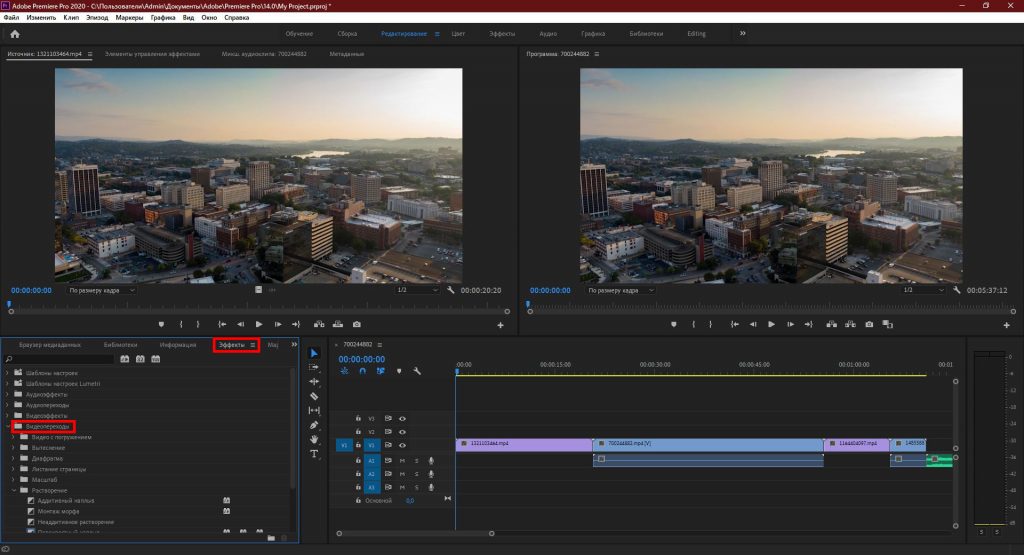 Считывания секвенирования демультиплексировали с использованием MiSeq Reporter (Illumina). Выравнивание последовательностей ампликонов с эталонной последовательностью выполняли с использованием CRISPResso2 (ссылка 54). Для всех количественных оценок эффективности простого редактирования эффективность редактирования рассчитывалась как процент чтений с желаемым редактированием без вставок от общего числа чтений со средней оценкой phred не менее тридцати. Для количественной оценки редактирования точечных мутаций CRISPResso2 запускали в стандартном режиме с включенным «discard_indel_reads». Выход редактирования рассчитывался как процент неотброшенных прочтений, содержащих редактирование, деленный на общее количество прочтений. Для редактирования вставки или удаления CRISPResso2 запускался в режиме HDR, используя желаемый аллель в качестве ожидаемого аллеля и с включенным «discard_indel_reads». Эффективность редактирования рассчитывалась как процентное соотношение чтений, выровненных по HDR, к общему количеству чтений.
Считывания секвенирования демультиплексировали с использованием MiSeq Reporter (Illumina). Выравнивание последовательностей ампликонов с эталонной последовательностью выполняли с использованием CRISPResso2 (ссылка 54). Для всех количественных оценок эффективности простого редактирования эффективность редактирования рассчитывалась как процент чтений с желаемым редактированием без вставок от общего числа чтений со средней оценкой phred не менее тридцати. Для количественной оценки редактирования точечных мутаций CRISPResso2 запускали в стандартном режиме с включенным «discard_indel_reads». Выход редактирования рассчитывался как процент неотброшенных прочтений, содержащих редактирование, деленный на общее количество прочтений. Для редактирования вставки или удаления CRISPResso2 запускался в режиме HDR, используя желаемый аллель в качестве ожидаемого аллеля и с включенным «discard_indel_reads». Эффективность редактирования рассчитывалась как процентное соотношение чтений, выровненных по HDR, к общему количеству чтений.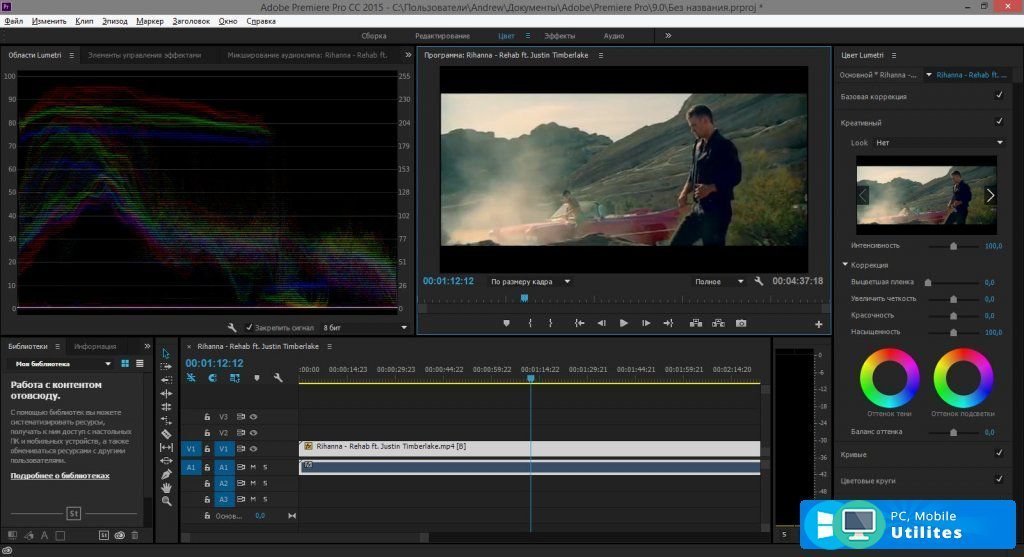 Для всех экспериментов частота инделей рассчитывалась как количество отброшенных прочтений, деленное на общее количество прочтений. Для экспериментов с участием PE2 чтения анализировали на наличие инделей в пределах 10 нуклеотидов вверх и вниз по течению от ник-сайта пегРНК включительно. Для экспериментов с участием PE3 чтения анализировали на наличие вставок между 10 нуклеотидами выше ник-сайта пегРНК и ниже ник-сайта sgRNA включительно. Нецелевое редактирование определяли количественно, как описано ранее 14 .
Для всех экспериментов частота инделей рассчитывалась как количество отброшенных прочтений, деленное на общее количество прочтений. Для экспериментов с участием PE2 чтения анализировали на наличие инделей в пределах 10 нуклеотидов вверх и вниз по течению от ник-сайта пегРНК включительно. Для экспериментов с участием PE3 чтения анализировали на наличие вставок между 10 нуклеотидами выше ник-сайта пегРНК и ниже ник-сайта sgRNA включительно. Нецелевое редактирование определяли количественно, как описано ранее 14 .RTqPCR тотальной РНК.
10 000 клеток HEK293T на лунку высевали в 96-луночные планшеты. Через 16–24 часа после посева клетки трансфицировали примерно при 60% конфлюэнтности 0,5 мкл липофектамина 2000, 200 нг плазмиды PE2 и 40 нг плазмиды пегРНК или эпеРНК в соответствии с протоколами производителя. Через три дня тотальную РНК выделяли с помощью универсального набора AllPrep DNA/RNA/miRNA (Qiagen). Набор Power SYBR Green Cells-to-C T (Thermo Fisher Scientific) использовали для создания кДНК с использованием случайных гексамеров и для проведения количественной ПЦР с прямыми и обратными праймерами, которые амплифицируют каркас пегРНК в соответствии с протоколами производителя.
 Последовательности праймеров доступны в дополнительной таблице 5.
Последовательности праймеров доступны в дополнительной таблице 5.Анализы чувствительности к экзонуклеазам in vitro.
пегРНК или эпегРНК, содержащие либо mpknot, либо evopreQ 1 , были получены с использованием набора для синтеза РНК HiScribe T7 Quick High Yield RNA (New England BioLabs) из ПЦР-амплифицированных матриц, содержащих последовательность промотора Т7, в соответствии с протоколами производителя с последующей очисткой с помощью Monarch Набор для очистки РНК (New England BioLabs). Ядерные экстракты были приготовлены из 3 миллионов клеток HEK293T, выращенных до 70–80% слияния в соответствии с протоколами производителя, с использованием набора EpiQuik Nuclear Extraction Kit (EpiGentek). Анализы проводили в реакционных смесях объемом 10 мкл, содержащих 20 мМ трис-HCl (pH 7,5 при 23 °C), 5 мМ MgCl 2 , 50 мМ NaCl, 2 мМ DTT, 1 мМ NTP и 0,8 ед/мкл рекомбинантного ингибитора рибонуклеазы RNaseOUT (40 ед/мкл; ThermoFisher Scientific) для ингибирования эндонуклеазной активности.
 3 мкл свежего ядерного лизата использовали для деградации 0,5 мкг РНК-субстрата на реакцию. После инкубации реакционных смесей при 37 °С в течение 20 мин продукты деградации разделяли на 2,0% агарозных гелях, окрашенных SYBR Gold. Степень деградации определяли с использованием программного обеспечения ImageJ (NIH). Чтобы определить, является ли Cas9мог защитить каркас sgRNA от деградации экзонуклеазами, 1 нМ пегРНК или эпигРНК инкубировали в присутствии или в отсутствие 100 нМ nCas9 при комнатной температуре в течение 10 минут, чтобы обеспечить связывание nCas9-H840A с пегРНК. Анализы деградации проводили в 10 мкл реакционных смесей, содержащих 20 мМ Tris-HCl (pH 7,5 при 23 °C), 5 мМ MgCl 2 , 50 мМ NaCl, 2 мМ DTT, 1 мМ dNTP и 0,8 U/ мкл ингибитора рекомбинантной рибонуклеазы RNaseOUT. 3 мкл свежего ядерного лизата использовали для деградации Cas9.никазо-связанная пегРНК или эпегРНК. После инкубации реакционных смесей при 37°С в течение 10 мин к реакционной смеси добавляли 1 мкл раствора протеазы К (Qiagen) и инкубировали при комнатной температуре в течение 10 мин для инактивации нуклеаз.
3 мкл свежего ядерного лизата использовали для деградации 0,5 мкг РНК-субстрата на реакцию. После инкубации реакционных смесей при 37 °С в течение 20 мин продукты деградации разделяли на 2,0% агарозных гелях, окрашенных SYBR Gold. Степень деградации определяли с использованием программного обеспечения ImageJ (NIH). Чтобы определить, является ли Cas9мог защитить каркас sgRNA от деградации экзонуклеазами, 1 нМ пегРНК или эпигРНК инкубировали в присутствии или в отсутствие 100 нМ nCas9 при комнатной температуре в течение 10 минут, чтобы обеспечить связывание nCas9-H840A с пегРНК. Анализы деградации проводили в 10 мкл реакционных смесей, содержащих 20 мМ Tris-HCl (pH 7,5 при 23 °C), 5 мМ MgCl 2 , 50 мМ NaCl, 2 мМ DTT, 1 мМ dNTP и 0,8 U/ мкл ингибитора рекомбинантной рибонуклеазы RNaseOUT. 3 мкл свежего ядерного лизата использовали для деградации Cas9.никазо-связанная пегРНК или эпегРНК. После инкубации реакционных смесей при 37°С в течение 10 мин к реакционной смеси добавляли 1 мкл раствора протеазы К (Qiagen) и инкубировали при комнатной температуре в течение 10 мин для инактивации нуклеаз. Всю оставшуюся РНК выделяли с использованием набора Monarch RNA Cleanup Kit (New England Biolabs) для анализа с помощью RT-qPCR.
Всю оставшуюся РНК выделяли с использованием набора Monarch RNA Cleanup Kit (New England Biolabs) для анализа с помощью RT-qPCR.Обнаружение pegRNAs и epegRNAs в клеточных лизатах с помощью нозерн-блоттинга.
Клетки HEK293T трансфицировали плазмидами, кодирующими PE2 и pegRNA или epegRNA, как описано выше. Клетки лизировали через 3 дня и выделяли общую РНК с использованием универсального набора AllPrep DNA/RNA/miRNA (Qiagen) в соответствии с инструкциями производителя. Количество мРНК PE2 определяли с помощью RT-qPCR, и это значение использовали для нормализации лизатов до той же концентрации мРНК PE2. Лизаты разделяли с помощью PAGE с использованием 10% денатурирующего геля PAGE (Criterion, Biorad). Лестницу оцРНК метили с 3′-конца дигоксигенин-ddUTP с использованием терминальной трансферазы (набор для мечения 3′-конца олигонуклеотидов DIG, Roche) и использовали в качестве маркера. Другими используемыми маркерами были пегРНК, транскрибированная in vitro, и матрица эпигРНК при +1 вставке тега FLAG в HEK3 и HEK293T клеточные лизаты, содержащие HEK3 , нацеленную на sgRNA.
 Перенос и перекрестное связывание РНК с мембраной нозерн-блоттинга в основном следовали ранее описанным процедурам обнаружения малых РНК 55 . РНК переносили на положительно заряженную нейлоновую мембрану (Roche) с использованием полусухого геля Trans-Blot SD при 20 В в течение 1 часа (1–3 мА/см 2 ). Затем РНК сшивали с мембраной путем замачивания в водном растворе 0,162 М EDC (1-этил-3-(3-диметиламинопропил)карбодиимид, Sigma Aldrich) и 0,17 М 1-метилимидазола (Sigma Aldrich, pH 8,0 при 23 °C). ) при 60°С в течение 1 часа. Блоты несколько раз кратко промывали водой, обработанной DEPC, а затем предварительно гибридизовали в гибридизационном буфере Ultrahyb (набор northern Max, Thermo Fisher) при 68 °C в течение 2 часов. Зонд РНК, комплементарный 64 нуклеотидам каркаса sgRNA (дополнительная таблица 5), был создан и помечен телом с помощью дигоксигенина-UTP посредством транскрипции in vitro с T7 и с использованием стартового набора DIG Northern (Roche). 5 пмоль меченого зонда добавляли к 0,5 мл буфера Ultrahyb и инкубировали в течение 5 мин при 70°C, а затем добавляли к предварительно гибридизованному блоту при 68°C.
Перенос и перекрестное связывание РНК с мембраной нозерн-блоттинга в основном следовали ранее описанным процедурам обнаружения малых РНК 55 . РНК переносили на положительно заряженную нейлоновую мембрану (Roche) с использованием полусухого геля Trans-Blot SD при 20 В в течение 1 часа (1–3 мА/см 2 ). Затем РНК сшивали с мембраной путем замачивания в водном растворе 0,162 М EDC (1-этил-3-(3-диметиламинопропил)карбодиимид, Sigma Aldrich) и 0,17 М 1-метилимидазола (Sigma Aldrich, pH 8,0 при 23 °C). ) при 60°С в течение 1 часа. Блоты несколько раз кратко промывали водой, обработанной DEPC, а затем предварительно гибридизовали в гибридизационном буфере Ultrahyb (набор northern Max, Thermo Fisher) при 68 °C в течение 2 часов. Зонд РНК, комплементарный 64 нуклеотидам каркаса sgRNA (дополнительная таблица 5), был создан и помечен телом с помощью дигоксигенина-UTP посредством транскрипции in vitro с T7 и с использованием стартового набора DIG Northern (Roche). 5 пмоль меченого зонда добавляли к 0,5 мл буфера Ultrahyb и инкубировали в течение 5 мин при 70°C, а затем добавляли к предварительно гибридизованному блоту при 68°C. Гибридизацию оставляли на ночь. Затем блоты дважды промывали в промывочном растворе с низкой жесткостью (набор northernMax, эквивалентный 2x SSC и 0,1% SDS) в течение 5 минут при комнатной температуре и дважды в промывочном растворе с высокой жесткостью (набор northernMax, эквивалентный 0,1x SSC и 0,1% SDS). в течение 15 мин при 68°С. Затем блоты промывали промывочным буфером (0,1 М малеиновая кислота, 0,15 М NaCl, 0,3% (об./об.) Tween 20, pH 7,5 при 23 °C) в течение 5 мин при комнатной температуре, а затем инкубировали в блокирующем буфере (DIG Northern Starter). комплект) в течение 30 мин при комнатной температуре. Затем блоты инкубировали в блокирующем буфере с добавлением антитела против дигоксигенина-AP (DIG Northern Starter Kit) в течение 30 минут при комнатной температуре, а затем дважды промывали промывочным буфером в течение 15 минут при комнатной температуре. Затем блоты уравновешивали буфером для обнаружения (0,1 М трис-HCl, 0,1 М NaCl, pH 9)..5 23 °C) в течение 5 мин перед удалением в папку для разработки.
Гибридизацию оставляли на ночь. Затем блоты дважды промывали в промывочном растворе с низкой жесткостью (набор northernMax, эквивалентный 2x SSC и 0,1% SDS) в течение 5 минут при комнатной температуре и дважды в промывочном растворе с высокой жесткостью (набор northernMax, эквивалентный 0,1x SSC и 0,1% SDS). в течение 15 мин при 68°С. Затем блоты промывали промывочным буфером (0,1 М малеиновая кислота, 0,15 М NaCl, 0,3% (об./об.) Tween 20, pH 7,5 при 23 °C) в течение 5 мин при комнатной температуре, а затем инкубировали в блокирующем буфере (DIG Northern Starter). комплект) в течение 30 мин при комнатной температуре. Затем блоты инкубировали в блокирующем буфере с добавлением антитела против дигоксигенина-AP (DIG Northern Starter Kit) в течение 30 минут при комнатной температуре, а затем дважды промывали промывочным буфером в течение 15 минут при комнатной температуре. Затем блоты уравновешивали буфером для обнаружения (0,1 М трис-HCl, 0,1 М NaCl, pH 9)..5 23 °C) в течение 5 мин перед удалением в папку для разработки. Затем к блоту по каплям добавляли CDP- Star , блот накрывали и инкубировали при комнатной температуре от 5 минут до ночи перед визуализацией с помощью Biorad ChemiDoc MP. Уровни pegRNA и epegRNA определяли денситометрией с использованием ImageJ.
Затем к блоту по каплям добавляли CDP- Star , блот накрывали и инкубировали при комнатной температуре от 5 минут до ночи перед визуализацией с помощью Biorad ChemiDoc MP. Уровни pegRNA и epegRNA определяли денситометрией с использованием ImageJ.Анализ связывания epegRNA.
Микромасштабный термофорез (MST) был проведен с использованием Monolith NT.Automated (Nanotemper) с капиллярами с первоклассным покрытием для определения Cas9.аффинности связывания с pegRNAs и epegRNAs или с dsDNA в комплексе с pegRNAs или epegRNAs. Реакции связывания проводили при 25 °C в 20 мкл буфера HBS-P (10 мМ HEPES, pH 7,4 при 23 °C, 150 мМ NaCl, 0,05% по объему поверхностно-активного вещества P20), содержащего 1 мМ MgCl 2 . Концентрация Mg 2+ была выбрана так, чтобы имитировать расчетную концентрацию свободного Mg 2+ в клетках человека 56 . Для экспериментов по связыванию РНК РНК транскрибировали in vitro , как описано ранее, и 3′-метилировали с помощью pCp-Cy5 (Jena Bioscience) с использованием РНК-лигазы T4 1 (New England BioLabs) в соответствии с протоколами производителя, модифицированными для включения 10 мкМ pCp-Cy5.
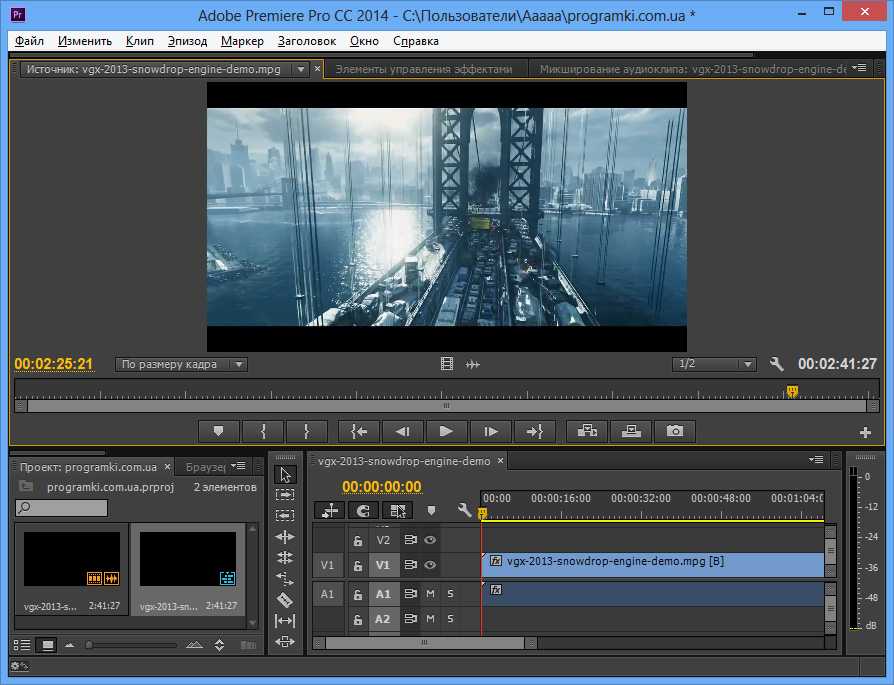 Cy5 с последующей очисткой с помощью набора Monarch RNA Cleanup (New England BioLabs). 1 нМ Cy5-меченую РНК денатурировали в течение 3 минут при 72 °C, выдерживали на льду в течение 1 минуты, а затем инкубировали с SpCas9.-H840A (Integrated DNA Technologies) в течение 30 минут при 25 °C перед анализом MST. Для экспериментов по связыванию двухцепочечной ДНК субстрат двухцепочечной ДНК собирали путем медленного отжига Cy5-меченого обратного олигонуклеотида и немеченого прямого олигонуклеотида, соответствующего геномному локусу HEK3 (дополнительная таблица 5). RNP Cas9 получали путем инкубации dSpCas9 (Integrated DNA Technologies) в течение 30 минут при 25 °C в буфере HBS-P с 1 мМ MgCl 2 и 50-кратным молярным избытком немеченой пегРНК или эпегРНК для поддержания насыщенных условий связывания РНК. Cas9Затем RNP инкубировали с 1 нМ субстрата двухцепочечной ДНК, меченной Cy5, в течение 30 минут при 25 ° C перед анализом MST. Cy5 демонстрирует существенное изменение интенсивности, связанное с температурой (TRIC), что обеспечивает достаточное усиление сигнала для обнаружения взаимодействий белок / пегРНК или двухцепочечной ДНК / РНП.
Cy5 с последующей очисткой с помощью набора Monarch RNA Cleanup (New England BioLabs). 1 нМ Cy5-меченую РНК денатурировали в течение 3 минут при 72 °C, выдерживали на льду в течение 1 минуты, а затем инкубировали с SpCas9.-H840A (Integrated DNA Technologies) в течение 30 минут при 25 °C перед анализом MST. Для экспериментов по связыванию двухцепочечной ДНК субстрат двухцепочечной ДНК собирали путем медленного отжига Cy5-меченого обратного олигонуклеотида и немеченого прямого олигонуклеотида, соответствующего геномному локусу HEK3 (дополнительная таблица 5). RNP Cas9 получали путем инкубации dSpCas9 (Integrated DNA Technologies) в течение 30 минут при 25 °C в буфере HBS-P с 1 мМ MgCl 2 и 50-кратным молярным избытком немеченой пегРНК или эпегРНК для поддержания насыщенных условий связывания РНК. Cas9Затем RNP инкубировали с 1 нМ субстрата двухцепочечной ДНК, меченной Cy5, в течение 30 минут при 25 ° C перед анализом MST. Cy5 демонстрирует существенное изменение интенсивности, связанное с температурой (TRIC), что обеспечивает достаточное усиление сигнала для обнаружения взаимодействий белок / пегРНК или двухцепочечной ДНК / РНП. Как правило, энергия лазерного возбуждения была установлена на 20 %, а мощность ИК-лазера была установлена на высокую для всех показаний. Все измерения были выполнены в трех повторностях, причем серия разведений для каждой повторности проводилась отдельно, с использованием серийных разведений либо dSpCas9, либо dSpCas9.или SpCas9-H840A от 100 нМ до 0 нМ. Данные были проанализированы в Prism 9 путем выполнения логистической регрессии по логарифму ([белок]) и изменению сигнала с точкой данных концентрации 0 нМ, установленной на 0,1 нМ для целей регрессии.
Как правило, энергия лазерного возбуждения была установлена на 20 %, а мощность ИК-лазера была установлена на высокую для всех показаний. Все измерения были выполнены в трех повторностях, причем серия разведений для каждой повторности проводилась отдельно, с использованием серийных разведений либо dSpCas9, либо dSpCas9.или SpCas9-H840A от 100 нМ до 0 нМ. Данные были проанализированы в Prism 9 путем выполнения логистической регрессии по логарифму ([белок]) и изменению сигнала с точкой данных концентрации 0 нМ, установленной на 0,1 нМ для целей регрессии.Активация транскрипции на основе Cas9.
10 000 клеток HEK293T на лунку высевали в 96-луночные планшеты с черными стенками (Corning). Через 16–24 часа после посева клетки трансфицировали примерно при 60% конфлюэнтности 0,5 мкл липофектамина 2000 в соответствии с протоколами производителя и 100 нг dCas9.– плазмида VPR, 30 нг репортерной плазмиды GFP, 15 нг плазмиды iRFP и 20 нг плазмиды sgRNA, pegRNA или epegRNA. Через три дня клетки измеряли на предмет флуоресценции GFP и iRFP с использованием устройства для считывания микропланшетов Infinite M1000 Pro (Tecan).
 Флуоресценцию GFP нормализовали по флуоресценции iRFP после вычитания фонового сигнала флуоресценции от необработанных клеток.
Флуоресценцию GFP нормализовали по флуоресценции iRFP после вычитания фонового сигнала флуоресценции от необработанных клеток.Анализ терминальной дезоксинуклеотидилтрансферазы.
10 000 клеток HEK293T на лунку высевали в 96-луночные планшеты. Через 16–24 часа после посева клетки трансфицировали примерно при 60% конфлюэнтности 0,5 мкл липофектамина 2000 в соответствии с протоколами производителя и 200 нг плазмиды PE2 и 40 нг плазмиды пегРНК или эпеРНК. Через 24 часа из клеток выделяли геномную ДНК с помощью набора Agencourt DNAdvance (Beckman Coulter) в соответствии с инструкциями производителя. 3′-концы были присоединены к гуанозину с использованием терминальной дезоксинуклеотидилтрансферазы (New England Biolabs) в соответствии с инструкциями производителя. Затем образцы снова очищали с использованием набора Agencourt DNAdvance перед ПЦР-амплификацией для высокопроизводительного секвенирования ДНК с использованием локус-специфического прямого праймера и олиго-C (C 18 ) обратная грунтовка.
 Последовательности праймеров доступны в дополнительной таблице 5. Промежуточные продукты первичного редактирования были количественно определены с использованием пользовательского сценария Python, доступного в дополнительном примечании 3.
Последовательности праймеров доступны в дополнительной таблице 5. Промежуточные продукты первичного редактирования были количественно определены с использованием пользовательского сценария Python, доступного в дополнительном примечании 3.Дизайн линкера с помощью pegLIT.
Для разработки линкерных последовательностей эпигРНК мы написали специальный алгоритм, инструмент идентификации линкера пегРНК или pegLIT, который ищет линкерные последовательности заданной длины, минимизирующие спаривание оснований с остальной частью пегРНК. Чтобы сократить время вычислений, в этой процедуре используется имитация отжига для максимизации подоценок 57 , каждая из которых соответствует подпоследовательности пегРНК: спейсер, PBS, матрица или скаффолд. Соответственно, разные линкерные последовательности могут быть созданы для одной и той же эпигРНК, если алгоритм выполняется несколько раз. Во время оптимизации линкер с более высокой оценкой в любой паре линкеров определялся путем сравнения их дискретизированных подпоказателей в порядке следующего приоритета подпоследовательности: спейсер, PBS, шаблон и затем каркас.
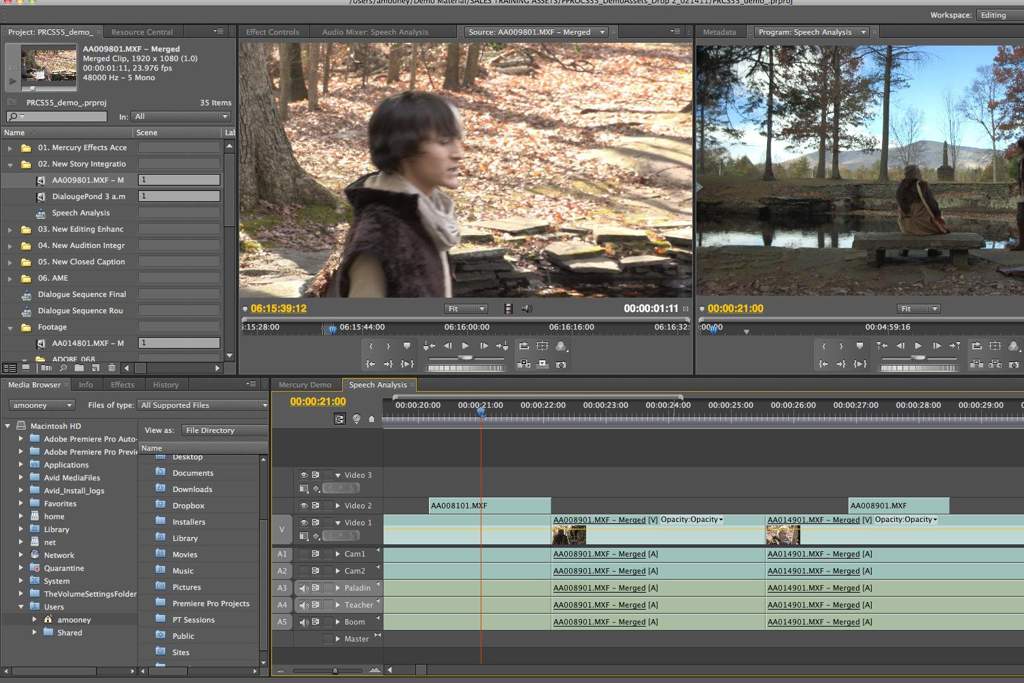 Каждая подшкала рассчитывается с использованием вероятностей пар оснований, рассчитанных программой ViennaRNA 2.0 (ссылка 23) при стандартных параметрах (37 °C, 1 М NaCl, 0,05 М MgCl 9 ).0329 2 ), как дополнение средней вероятности того, что нуклеотид в линкере образует пару оснований с любым нуклеотидом в рассматриваемой подпоследовательности пегРНК, где среднее значение берется по всем основаниям в линкере. Линкерные последовательности с содержанием AC < 50% и те, которые приводят к pegRNA, содержащей последовательно четыре одинаковых нуклеотида, удаляются из рассмотрения 39,58 . При необходимости алгоритм выполняет иерархическую агломеративную кластеризацию 100 линкеров с наивысшей оценкой и выводит по одному линкеру на кластер, чтобы способствовать разнообразию последовательностей в конечном результате. Наша реализация pegLIT на Python общедоступна на liugroup.us, а код можно найти в дополнительном примечании 2 или на github.com/sshen8/peglit.
Каждая подшкала рассчитывается с использованием вероятностей пар оснований, рассчитанных программой ViennaRNA 2.0 (ссылка 23) при стандартных параметрах (37 °C, 1 М NaCl, 0,05 М MgCl 9 ).0329 2 ), как дополнение средней вероятности того, что нуклеотид в линкере образует пару оснований с любым нуклеотидом в рассматриваемой подпоследовательности пегРНК, где среднее значение берется по всем основаниям в линкере. Линкерные последовательности с содержанием AC < 50% и те, которые приводят к pegRNA, содержащей последовательно четыре одинаковых нуклеотида, удаляются из рассмотрения 39,58 . При необходимости алгоритм выполняет иерархическую агломеративную кластеризацию 100 линкеров с наивысшей оценкой и выводит по одному линкеру на кластер, чтобы способствовать разнообразию последовательностей в конечном результате. Наша реализация pegLIT на Python общедоступна на liugroup.us, а код можно найти в дополнительном примечании 2 или на github.com/sshen8/peglit.Доступность данных
Данные высокопроизводительного секвенирования депонированы в базу данных NCBI Sequence Read Archive по адресу PRJNA707486.
 Плазмиды, кодирующие выбранные векторы экспрессии pegRNA и векторы клонирования Golden Gate, были депонированы в Addgene для распространения.
Плазмиды, кодирующие выбранные векторы экспрессии pegRNA и векторы клонирования Golden Gate, были депонированы в Addgene для распространения.Доступность кода
Реализация pegLIT на Python общедоступна на peglit.liugroup.us, а код можно найти в дополнительном примечании 2 или на github.com/sshen8/peglit.
Полный СИ
Нажмите здесь для просмотра. (6.5M, pdf)
Вкладка Sup
Щелкните здесь для просмотра. (123K, xlsx)
Эта работа была поддержана NIH США U01Al142756, RM1HG009490, R01EB031172 и R35GM118062, Медицинским институтом Говарда Хьюза и Фондом Лулу. Дж.В.Н. и А.В.А. были поддержаны постдокторскими стипендиями Джейн Коффин Чайлдс, PBR, SPS, KAE и PJC. были поддержаны стипендиями выпускников NSF, G.A.N. был поддержан постдокторской стипендией Хелен Хэй Уитни. Мы благодарим доктора Анахиту Виейру за помощь в редактировании этой рукописи, Мэри О’Рейли, Елену Берг и команду Broad Institute Pattern за помощь в разработке рисунка, Шона МакГрири и Кехуи Сян за полезные обсуждения процедур нозерн-блоттинга и Макса Шена за полезные обсуждения.
 по кодированию pegLIT.
по кодированию pegLIT.Конкурирующие интересы
Авторы являются соавторами патентов, поданных Институтом Броуда на основное редактирование. Д.Р.Л. является консультантом и соучредителем компаний Prime Medicine, Beam Therapeutics и Pairwise Plants, которые используют редактирование генома. А.В.А. в настоящее время является сотрудником Prime Medicine.
Дополнительная информация Дополнительное обсуждение, дополнительные рисунки 1–16, дополнительные таблицы 1–6, дополнительные примечания 1–3
1. Komor AC, Badran AH & Liu DR Технологии на основе CRISPR для манипулирования геномами эукариот. Клетка 168, 20–36 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
2. Анзалоне А.В., Коблан Л.В. и Лю Д.Р. Редактирование генома с помощью нуклеаз CRISPR-Cas, базовых редакторов, транспозаз и основных редакторов. Нат Биотехнолог 38, 824–844 (2020). [PubMed] [Google Scholar]
3. Калло Г. и другие. Редактирование генома CRISPR-Cas9 вызывает усечение хромосом в масштабе мегабаз.
 Нац Коммуна
10, 1136 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Нац Коммуна
10, 1136 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]4. Косицки М., Томберг К. и Брэдли А. Репарация двухцепочечных разрывов, индуцированная CRISPR-Cas9, приводит к большим делециям и сложным перестройкам. Нат Биотехнолог 36, 765–771 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
5. Боровяк К., Фу Б., Ян Ф., Доу Б. и Брэдли А. Выявление скрытых сложностей геномных перестроек, созданных с помощью Cas9. научный представитель 7, 12867 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
6. Enache OM и другие. Cas9 активирует путь р53 и выбирает мутации, инактивирующие р53. Нат Жене 52, 662–668 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
7. Хаапаниеми Э., Ботла С., Перссон Дж., Шмирер Б. и Тайпале Дж. Редактирование генома CRISPR-Cas9 вызывает реакцию повреждения ДНК, опосредованную p53. Нат Мед 24, 927–930 (2018). [PubMed] [Google Scholar]
8. Игорь Р.Дж. и другие. p53 ингибирует инженерию CRISPR-Cas9 в плюрипотентных стволовых клетках человека.
 Нат Мед
24, 939–946 (2018). [PubMed] [Google Scholar]
Нат Мед
24, 939–946 (2018). [PubMed] [Google Scholar]9. Лейбовиц М.Л. и другие. Хромотрипсис как целевое следствие редактирования генома CRISPR-Cas9. Нат Жене 10.1038/s41588-021-00838-7 (2021). [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
10. Burgio G & Teboul L Предвидение и выявление побочного ущерба при редактировании генома. Тенденции Жене 36, 905–914 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
11. Cox DB, Platt RJ & Zhang F Терапевтическое редактирование генома: перспективы и проблемы. Нат Мед 21, 121–131 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
12. Komor AC, Kim YB, Packer MS, Zuris JA & Liu DR Программируемое редактирование целевого основания в геномной ДНК без расщепления двухцепочечной ДНК. Природа 533, 420–424 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
13. Gaudelli NM и другие. Программируемое редактирование оснований от A*T до G*C в геномной ДНК без расщепления ДНК. Природа 551, 464–471 (2017).
 [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]14. Анзалон А.В. и другие. Редактирование генома с помощью поиска и замены без двухцепочечных разрывов или донорской ДНК. Природа 576, 149–157 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
15. Houseley J & Tollervey D Множество путей деградации РНК. Клетка 136, 763–776 (2009). [PubMed] [Google Scholar]
16. Хендель А. и другие. Химически модифицированные направляющие РНК улучшают редактирование генома CRISPR-Cas в первичных клетках человека. Нат Биотехнолог 33, 985–989 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
17. Geisberg JV, Moqtaderi Z, Fan X, Ozsolak F & Struhl K Общий анализ периодов полураспада изоформ мРНК выявляет стабилизирующие и дестабилизирующие элементы в дрожжах. Клетка 156, 812–824 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
18. Wu X & Bartel DP Широко распространенное влияние 3′-концевых структур на процессинг и стабильность мРНК млекопитающих.
 Клетка
169, 905–917 e911 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Клетка
169, 905–917 e911 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]19. Браун Дж. А. и другие. Структурное понимание стабилизации некодирующей РНК MALAT1 с помощью двойной тройной спирали. Nat Struct Мол Биол 21, 633–640 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
20. Рот А. и другие. Рибопереключатель, селективный в отношении предшественника кеуозина preQ1, содержит необычно маленький аптамерный домен. Nat Struct Мол Биол 14, 308–317 (2007). [PubMed] [Google Scholar]
21. Анзалоне А.В., Лин А.Дж., Заирис С., Рабадан Р. и Корниш В.В. Перепрограммирование эукариотической трансляции с помощью лиганд-чувствительных синтетических РНК-переключателей. Нат Методы 13, 453–458 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
22. Houck-Loomis B и другие. Зависимый от равновесия ретровирусный переключатель мРНК регулирует трансляционную запись. Природа 480, 561–564 (2011). [Бесплатная статья PMC] [PubMed] [Google Scholar]
23.
 Лоренц Р.
и другие.
Пакет ViennaRNA 2.0. Алгоритмы Мол Биол
6, 26 (2011). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Лоренц Р.
и другие.
Пакет ViennaRNA 2.0. Алгоритмы Мол Биол
6, 26 (2011). [Бесплатная статья PMC] [PubMed] [Google Scholar]24. Schene IF и другие. Основное редактирование для функционального восстановления в моделях заболеваний, полученных от пациентов. Нац Коммуна 11, 5352 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
25. Kim DY, Moon SB, Ko JH, Kim YS и Kim D Беспристрастное исследование особенностей основных систем редактирования в клетках человека. Нуклеиновые Кислоты Res 48, 10576–10589 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
26. Гао П. и другие. Прайм-редактирование у мышей показывает важность одного основания в управлении экспрессией тканеспецифического гена. Геном Биол 22, 83 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
27. Lin Q, et al. Высокоэффективное первичное редактирование с оптимизированными парными пегРНК в растениях. Нат Биотехнолог (2021). 10.1038/s41587-021-00868-w. [PubMed] [CrossRef] [Google Scholar]
28.
 Tsai SQ
и другие.
GUIDE-seq обеспечивает полногеномное профилирование нецелевого расщепления нуклеазами CRISPR-Cas. Нат Биотехнолог
33, 187–197 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Tsai SQ
и другие.
GUIDE-seq обеспечивает полногеномное профилирование нецелевого расщепления нуклеазами CRISPR-Cas. Нат Биотехнолог
33, 187–197 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]29. Schmid M & Jensen TH Экзосома: многоцелевая машина для распада РНК. Тенденции биохимии 33, 501–510 (2008). [PubMed] [Google Scholar]
30. Ибрагим Х., Вилуш Дж. и Вилуш С.Дж. Распознавание РНК экзонуклеазами с 3′-на 5′: взгляд с субстрата. Биохим биофиз акта 1779, 256–265 (2008). [Бесплатная статья PMC] [PubMed] [Google Scholar]
31. Green L, Kim CH, Bustamante C & Tinoco I Jr. Характеристика механического развертывания псевдоузлов РНК. Джей Мол Биол 375, 511–528 (2008). [Бесплатная статья PMC] [PubMed] [Google Scholar]
32. Чавес А. и другие. Высокоэффективное Cas9-опосредованное программирование транскрипции. Нат Методы 12, 326–328 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
33. Hu JH и другие. Разработанные варианты Cas9 с широкой совместимостью с PAM и высокой специфичностью ДНК.
 Природа
556, 57–63 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Природа
556, 57–63 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]34. Nahar S и другие. Мотив G-квадруплекса на 3′-конце sgRNA повышает эффективность редактирования генома на основе CRISPR-Cas9. Химическая коммуна 54, 2377–2380 (2018). [PubMed] [Академия Google]
35. Штекельберг А.Л. и другие. Свернутая вирусная некодирующая РНК блокирует экзорибонуклеазы клетки-хозяина через конформационно-динамическую структуру РНК. Proc Natl Acad Sci USA 115, 6404–6409 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
36. Cate JH и другие. Кристаллическая структура рибозимного домена группы I: принципы упаковки РНК. Наука 273, 1678–1685 (1996). [PubMed] [Google Scholar]
37. Федор М.Ю. и Вестхоф Э. Рибозимы: первые 20 лет. Мол Ячейка 10, 703–704 (2002). [PubMed] [Академия Google]
38. Чен Б. и другие. Динамическая визуализация геномных локусов в живых клетках человека с помощью оптимизированной системы CRISPR/Cas. Клетка 155, 1479–1491 (2014).
 [Бесплатная статья PMC] [PubMed] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [Google Scholar]39. Йост М. и другие. Титрование экспрессии генов с использованием библиотек систематически аттенуированных направляющих РНК CRISPR. Нат Биотехнолог 38, 355–364 (2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
40. Basila M, Kelley ML & Smith AVB Минимальная схема модификации 2′-O-метилфосфотиоатной связи синтетических направляющих РНК для повышения стабильности и эффективности CRISPR-Cas9редактирование генов во избежание клеточной токсичности. PLoS Один 12, e0188593 (2017). [Бесплатная статья PMC] [PubMed] [Google Scholar]
41. Мид С. и другие. Новый вариант защитного прионного белка, который колокализуется при воздействии куру. N Engl J Med 361, 2056–2065 (2009). [PubMed] [Google Scholar]
42. Asante EA и другие. Встречающийся в природе вариант человеческого прионного белка полностью предотвращает прионные заболевания. Природа 522, 478–481 (2015). [Бесплатная статья PMC] [PubMed] [Google Scholar]
43.
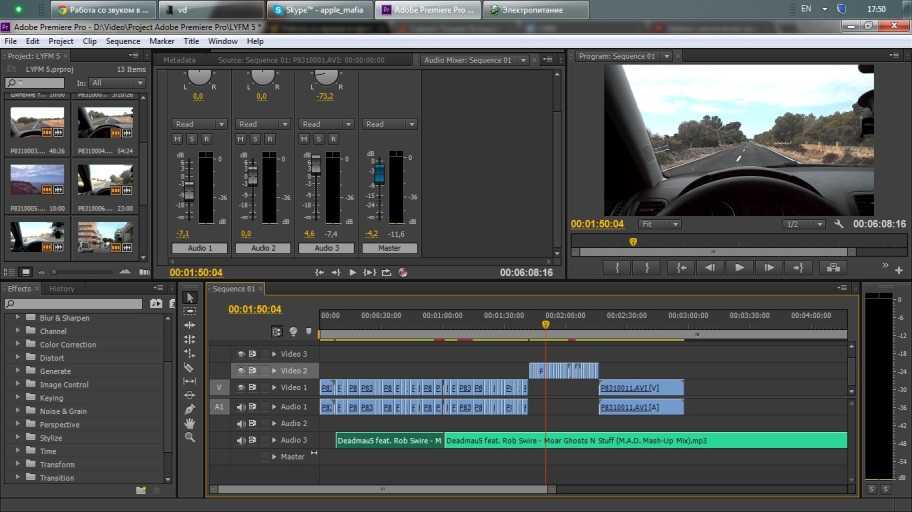 Kim HK
и другие.
Прогнозирование эффективности основных направляющих РНК для редактирования в клетках человека. Нат Биотехнолог (2020). [PubMed] [Академия Google]
Kim HK
и другие.
Прогнозирование эффективности основных направляющих РНК для редактирования в клетках человека. Нат Биотехнолог (2020). [PubMed] [Академия Google]44. Йонссон Т. и другие. Мутация в APP защищает от болезни Альцгеймера и возрастного снижения когнитивных функций. Природа 488, 96–99 (2012). [PubMed] [Google Scholar]
45. Абифадель М. и другие. Мутации PCSK9 вызывают аутосомно-доминантную гиперхолестеринемию. Нат Жене 34, 154–156 (2003). [PubMed] [Google Scholar]
46. Бустами Дж. и другие. Белок-переносчик эфира холестерина (CETP) полиморфизм I405V и сердечно-сосудистые заболевания у восточноевропейских европеоидов — перекрестное исследование. БМС Гериатр 16, 144 (2016). [Бесплатная статья PMC] [PubMed] [Google Scholar]
47. Фланник Дж. и другие. Мутации с потерей функции в SLC30A8 защищают от диабета 2 типа. Нат Жене 46, 357–363 (2014). [Бесплатная статья PMC] [PubMed] [Google Scholar]
48. Сакунтабхай А. и другие. Вариант промотора CD209 связан с тяжестью болезни денге.
 Нат Жене
37, 507–513 (2005). [Бесплатная статья PMC] [PubMed] [Google Scholar]
Нат Жене
37, 507–513 (2005). [Бесплатная статья PMC] [PubMed] [Google Scholar]49. Olson HE и другие. Циклин-зависимая киназоподобная 5-дефицитная недостаточность: клинический обзор. Педиатр Нейрол 97, 18–25 (2019). [Бесплатная статья PMC] [PubMed] [Google Scholar]
50. Аль-Сааиди Р. и другие. Мутация LMNA p.Arg321Ter, связанная с дилатационной кардиомиопатией, приводит к снижению экспрессии и искаженному соотношению белков ламина А и ламина С. Разрешение ячейки опыта 319, 3010–3019 (2013). [PubMed] [Google Scholar]
51. Ип JPK, Меллиос Н. и Сур М. Синдром Ретта: понимание генетических, молекулярных и схемных механизмов. Нат Рев Нейроски 19, 368–382 (2018). [Бесплатная статья PMC] [PubMed] [Google Scholar]
52. Христодулу Дж., Гримм А., Махер Т. и Беннеттс Б. RettBASE: база данных вариаций MECP2 IRSA — новая эволюционирующая база данных мутаций. Хум Мутат 21, 466–472 (2003). [PubMed] [Google Scholar]
53. Гауделли Н.М. и другие. Направленная эволюция редакторов оснований аденина с повышенной активностью и терапевтическим применением.

